La Bioinformática: el nuevo “gabinete de curiosidades” Understand article
El tratamiento de los “datos masivos” en la biología molecular está cambiando la forma de trabajar de los científicos.
En el siglo XVI, un gabinete de curiosidades (o Wunderkammer) fue una manera popular de exhibir una colección privada de objetos extraordinarios. Especies animales, esqueletos, minerales, extraños objetos hechos a mano e intrigantes antigüedades del Nuevo Mundo se pudieron mostrar con gran encanto, despertando en los visitantes el sentido de la curiosidad en esa nueva era de las maravillas.
Con el tiempo, los gabinetes de curiosidades dejaron paso a los museos más modernos. Al igual que los gabinetes, los museos satisficieron dos tendencias profundamente humanas: la curiosidad y, el deseo de coleccionar y preservar el conocimiento.
Hoy en día, esas mismas tendencias, combinadas con las nuevas tecnologías y un tsunami de datos genéticos, están liderando el mayor cambio en las ciencias de la vida: la democratización del acceso. También, la catalogación del mundo visible de las especies biológicas, los científicos pueden ahora secuenciar el ADN de millones de especies e introducir la información en bases de datos, junto con otros datos de biología molecular. El resultado es un nuevo tipo de colección: un catálogo en continuo crecimiento de información biológica que puede ayudar a los científicos de cualquier lugar a dar sentido al mundo vivo.
Pero todos estos datos necesitan ser procesados y, la disciplina de bioinformática – que combina la biología con las ciencias computacionales – se ha desarrollado para desempeñar esta función.

Spencer Phillips/EMBL-EBI
La creación del gabinete
Laboratorios de investigación de todo el mundo producen miles de datos, que se guardan en base de datos especializadas – como las del Instituto Europeo de Bioinformática (EMBL-EBI), situado cerca de Cambridge, Reino Unidow1. La principal responsabilidad para EMBL-EBI es asegurar que los datos que guardan sean accesibles al público, de modo que, las colecciones se mantengan abiertas para los investigadores de cualquier lugar. “Ha sido en estos años que esta clase de accesibilidad se ha hecho factible, gracias a la mejora en los canales de comunicación, pero ahora lo esperan los usuarios”, dice Andy Yates, uno de los jefes de grupo en el EMBL-EBI. “El acceso a los datos es crucial para cualquiera que haga ciencia. Con un gabinete de curiosidades tradicional, el coleccionador era la máxima autoridad. Nosotros hacemos que los contenidos -y nosotros mismos – estén abiertos para volver a analizarse y revisarse. Es un paso necesario si queremos que nuestras fuentes sean realmente útiles”, dice.
Organización de los datos

en la EMBL-EBI: el centro de
datos acoge una extensa
cantidad de datos digitales,
usando cientos de servidores.
EMBL-EBI
Los gabinetes de curiosidades tradicionales organizaban las cosas por tipos. Las bases de datos modernas ordenan las fuentes de datos biológicas de la misma manera – en categorías. En las bases de datos, la información y las categorías están interconectadas, de modo que las bases de datos son como un gabinete de curiosidades “inteligente” o multidimensional.
Hoy en día, indexar las fuentes de datos públicas es tan fundamental como lo era en las primeras colecciones para hacer que los grupos de datos sean fáciles de encontrar entre los petabytes de datos. Sin indexar, no hay forma de saber qué hay en una base de datos o cómo acceder a ellos. Y, la descripción de los conjuntos de datos – llamados metadatos – también es necesaria: “Sin los metadatos, consultar una base de datos es como deambular a través del sótano del Louvre a ciegas, esperando encontrar la Mona Lisa”, declara Yates.
Para hacer estos conjuntos de datos, que se han conseguido con mucho esfuerzo, reusables para otros científicos, los procesadores de datos revisan cuidadosamente cada dato aportado para asegurar que cumplen los requisitos necesarios. Estos requisitos están reflejados en unas normas ampliamente aceptadas conocidas por su abreviación FAIR: fácil de encontrar, disponible, fácil de intercambiar y utilizar la información intercambiada y, reutilizable. Los conjuntos de datos científicos deben además estar puestos en contexto y unidos a la publicación científica que los describen.
Visualización de datos
Junto con la organización, la forma de visualizar los datos es también importante: poder “ver” la conexión entre datos anima a la gente a seguir investigando. “La primera diferencia evidente entre un gabinete de curiosidades y una base de datos es el contenido”, explica Jee-Hyub Kim, antiguo buscador de datos en EMBL-EBI. “Por otro lado, una colección de objetos físicos te provoca inmediatamente una sensación. Simplemente imagina cómo debe de sentirse alguien que nunca ha visto el océano cuando ve y toca una estrella de mar o un coral. Es difícil crear esta relación con algo que es tan intangible como lo son los datos. Es por eso por lo que necesitas una buena interfaz y herramientas de visualización – para permitir al usuario explorar e interactuar con un conjunto de datos o un objeto digital.
Un ejemplo de una herramienta de visualización es el Banco de Datos de Proteína en Europa (PDBe)w2, un recurso para recopilar, organizar y diseminar los datos de estructuras macromoleculares, como las proteínas. Aparte de ser un repositorio para los científicos que estudian proteínas, PDBe permite a los usuarios ver e interactuar digitalmente, modelos tridimensionales de proteínas. Estas visualizaciones pueden ser accesibles desde cualquier dispositivo conectado a internet en el mundo, incluidos móviles y tablets.
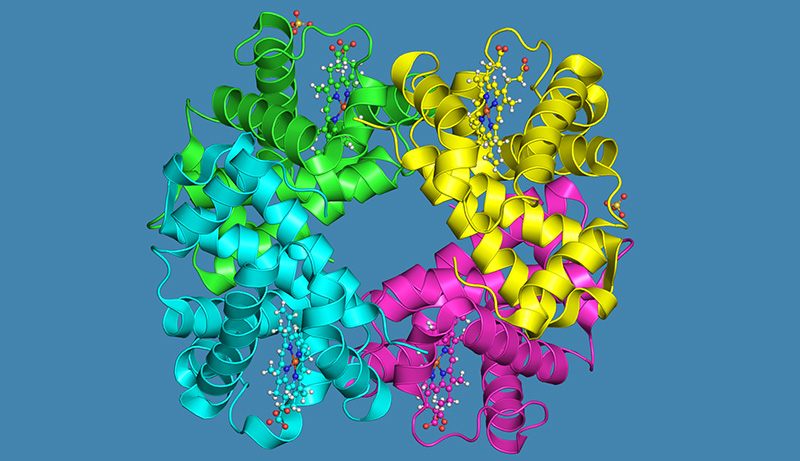
PDBe
Nuevos métodos, nuevos conocimientos
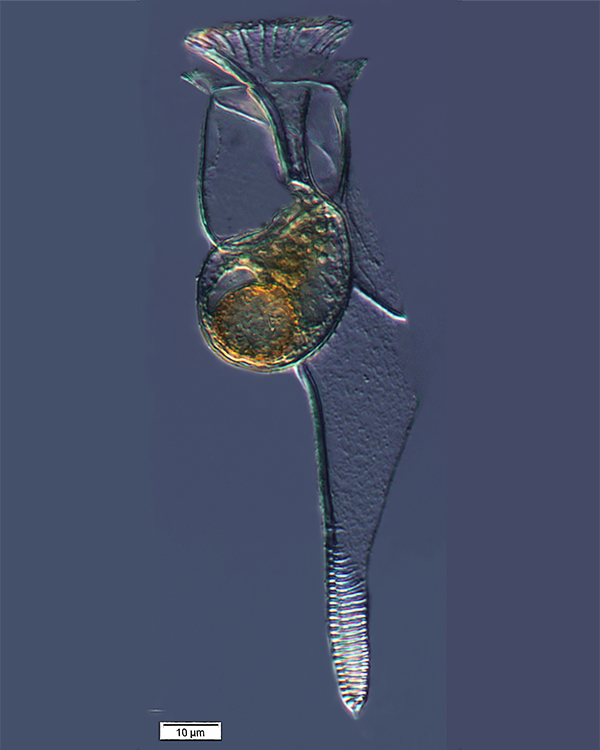
Histioneis elongata recogidos
por el equipo del Tara en el
sur del Océano Pacífico
tintinnidguy/Flickr.com
Entonces, ¿cómo están disponibles tantos datos que están cambiando la forma de hacer ciencia? Según Chuck Cook, director de los servicios científicos en EMBL-EBI, los científicos son cada vez más dependientes de los datos masivos – y, aquellos que no usan los datos masivos estarán a la cola a nivel profesional. “Según nos especializamos más, desarrollar experimentos aislados es más complicado. Para profundizar en la investigación, necesitamos colaborar con gente de especialidades muy diferentes.”
“Los biólogos tienen que convertirse, en cierto modo, en programadores”, afirma Yates. “Así es como las cuestiones científicas están cambiando. El investigador planteará una hipótesis y la confirmará o invalidará a través de la exploración de grandes fuentes de datos. Esto requiere cierto grado de conocimientos en programación”.
Al haber comenzado a analizar estos conjuntos de datos de forma masiva, los científicos están revelando nuevos conocimientos. Por ejemplo, los datos de la expedición Oceanográfica Tara, en la que un buque oceanográfico ha recorrido más de 300.000 Km alrededor del mundo desde el 2004, ha permitido descubrir más de 40 millones de nuevos genes y está ayudando a los científicos a entender el ecosistema invisible que mantiene la cadena alimenticia.
Los científicos durante el trayecto toman sistemáticamente muestras de plancton de todos los océanos del mundo, después lo llevan a tierra para la secuenciación del ADN y el análisis. “La secuenciación de las muestras del Tara nos ha permitido ‘ver’ alguna diversidad de vida en los océanos”, dice Rob Finn, un jefe de equipo de recursos metagenómicos de EMBL-EBI. “El primer grupo de 40 millones de genes identificados en las muestras oceánicas del Tara son principalmente procariotas – especies de bacterias que no habíamos visto antes. Pero en la segunda ronda de datos, identificamos unos 117 millones de genes eucariotas y, aún queda mucho camino por recorrer”, afirma.
Los detalles esenciales
A la luz de este creciente flujo de datos, ¿cuáles son los grandes retos que debe afrontar la biología en los próximos años? “Antes de los datos abiertos, un científico trabajaba en una proteína, gen o sistema experimental, probablemente durante toda su carrera”, comenta la profesora de investigación Janet Thornton, Directora Emérita del EMBL-EBI. “Viendo la situación, era prácticamente imposible. Hoy día, podemos hacer observaciones a escala genómica y de especies”, dice. Pero Thornton piensa que este cambio también plantea mayores retos: descubrimientos realmente importantes en biología aún permanecen sin los detalles esenciales.
“Todavía necesitamos investigar más profundamente esos detalles para comprender algunas cuestiones fundamentales, como ¿por qué los organismos envejecen?”, afirma. “Iniciativas como los Atlas de Células Humanasw3 son muy buenos ejemplos de todos los detalles desconocidos que aún necesitamos entender antes de empezar a explicar porqué las cosas funcionan. El siguiente paso será traducir todo este conocimiento a áreas del día a día, como la medicina, agricultura y biodiversidad”.
Al igual que los coleccionistas que construyeron los primeros gabinetes de curiosidades, los científicos están aún meticulosamente catalogando cada cosa que aprenden sobre la forma y función de la vida y, uniendo todo esto consiguen hacer más descubrimientos.
Agradecimientos
Este artículo está basado en una publicación original de EMBL etc., reproducido con todos los permisos.
Web References
- w1 – EMBL-EBI es el lugar donde se guardan los datos masivos de biología. El instituto alberga y comparte datos de experimentos en ciencias de la vida realizados en todo el mundo y, sus científicos desarrollan investigación básica en biología computacional. EMBL-EBI es uno de los de los lugares del Laboratorio Europeo de Biología Molecular y está ubicado a las afueras de Cambridge, Reino Unido.
- w2 – PDBe es una base de datos para datos estructurales en tres dimensiones relacionados con grandes moléculas biológicas, como proteínas y ácidos nucleicos. Los modelos están disponibles de forma gratuita para los científicos y estudiantes de todo el mundo.
- w3 – El Atlas Celular Humano pretende mapear cada célula individual del cuerpo humano usando tecnologías de secuenciación de una sola célula. Esta colaboración de toda la comunidad científica internacional une a biólogos, clínicos, genetistas, ingenieros informáticos y otros.
Resources
- Puedes encontrar más información sobre las expediciones Tara y las investigaciones en ecología en la página web de Tara.
- Para leer un artículo sobre las expediciones Tara en Science in School, consultar:
- Peyrot R (2015) Tara: una odisea oceánica. Science in School 33.
Institutions
Review
Este artículo muestra una idea fundamental para muchas disciplinas, desde las ciencias naturales a la economía: la gran cantidad de datos y conocimiento que poseemos necesita ser organizado de forma profesional de modo que pueda ser accesible para investigadores de todo el mundo.
Para el aprendizaje de la biología, el artículo puede ser usado para presentar el papel de los datos masivos y la bioinformática en la biología molecular y, para señalar cómo las nuevas tecnologías de la computación pueden ayudar a los científicos a comparar y visualizar secuencias de ADN y de proteínas. Esto puede animar a los estudiantes a sumergirse ellos mismos en las múltiples posibilidades que las tecnologías de la comunicación están abriendo en la ciencia.
El artículo también puede usarse para fomentar el conocimiento de la increíble biodiversidad que todavía no ha sido descubierta en océanos y en otros hábitats naturales inexplorados.
Jesús López Alonso, profesor de biología, IES La Gándara, España





