Evolució en acció: patògens Understand article
Els patògens que amenacen la salut humana evolucionen constantment per tal de mantenir una posició avantatjada respecte a les nostres defenses. Però com podem seguir aquests canvis a nivell genètic, fins i tot quan aquests s’estan esdevenint.
Una de les principals forces de l’evolució és la selecció natural – en la qual els individus amb una particular dotació genètica tenen una major descendència que la resta en un determinat ambient, derivant en adaptacions d’aquest individu que li permeten sobreviure, mentre que la resta moren.
Biblioteca Science Photo/De Angelis, Maurizio
Tal i com va establir Darwin, aquest procés de selecció natural positiva – en el qual les característiques que incrementen la capacitat de sobreviure i reproduir-se d’un determinat organisme esdevenen dominants en una població – contribueixen a la diversitat que podem observar en les espècies en l’actualitat. La selecció natural és també però, un factor en processos que posen en perill la nostra pròpia supervivència, com l’expansió de les infeccions víriques i l’emergència de les resistències a antibiòtics en patògens bacterials. Tot i això, gràcies a una sèrie d’experiments enginyosos (vegeu, per exemple, Bryk, 2017), i els avanços en la tecnologia de seqüenciació de gens, com les màquines de seqüenciació compacte, avui en dia podem seguir aquest procés de forma ràpida i precisa no només al laboratori, sinó gairebé a tot arreu del món. Aquest fet és molt important per a entendre l’evolució dels microorganismes que causen malalties.
En aquest article, ens centrem en dos estudis rellevants que investiguen amb deteniment com aquests patògens evolucionen constantment, revelant els patrons de canvi genètic – i els factors limitants – darrere d’aquest procés.
La resistència als antibiòtics sota el punt de mira
Un exemple molt clar d’evolució per selecció natural és l’emergència de resistència als antibiòtics en els bacteris. L’ús d’antibiòtics genera una pressió selectiva en la població de bacteris, que muten i es reprodueixen molt ràpidament. Com a resultat, qualsevol variant genètica que protegeixi els bacteris dels antibiòtics serà retinguda en la població, mentre que la resta de variants desapareixeran.
Aquest procés ha estat captat fotogràficament pel Dr Michael Baym, un investigador de la Universitat de Harvard, EUA, autodefinit com a combatent contra la resistència als antibiòtics. Al 2015, Baym va establir un dispositiu per a visualitzar com els bacteris resistents s’escampen en un ambient amb presència d’antibiòtics. Va utilitzar una placa de Petri rectangular gegant, 60 cm d’ample per 120 cm de llarg (una placa de Petri estàndard mesura 9 cm de diàmetre), plena d’agar, un medi gelatinós per a fer créixer bacteris, tenyit de color negre. Crucialment, va infusionar l’agar amb concentracions creixents d’antibiòtic al llarg de la placa (figura 1), des de absència d’antibiòtic als extrems fins a 1000 unitats d’antibiòtic a les seccions centrals – que haurien de ser suficients per matar immediatament qualsevol bacteri. Baym va preparar un punt de mira amb una càmera sobre la placa, de manera que qualsevol colònia de bacteris pogués ser clarament visible com a punts blancs sobre el fons d’agar negre. Aleshores, va inocular la placa amb bacteris Escherichia coli (E. coli) els extrems de la placa, on no hi havia antibiòtic – i va esperar que comencés el procés (figura 2).

Laboratori Kishony, Escola Mèdica de Harvard i Technion – Institut de Tecnologia d’Israel
Així doncs, què va passar? Els bacteris d’E. coli es podien moure en la capa superior de l’agar, de manera que quan els bacteris es quedaven sense aliment al seu voltant es movien cap a una altra regió (figura 3). No obstant, el seu creixement era inhibit per l’antibiòtic present en l’àrea adjacent, de manera que només aquells bacteris que presentaven mutacions que els permetien sobreviure a l’exposició a antibiòtics eren capaços d’expandir-se més lluny. D’aquesta manera, la primera regió que contenia antibiòtic (en una concentració d’una unitat) va ser inicialment envaïda per mutants simples. Aquests mutants es van multiplicar i la seva descendència va disseminar-se sobre tota la regió – fins que es van trobar amb la següent regió que contenia una concentració d’antibiòtic més elevada. Al límit de la regió, els bacteris es van aturar una altra vegada fins que van emergir noves mutacions que van permetre una tolerància a l’antibiòtic més elevada.
Aquest procés es va repetir en cada límit fins que, després d’11 dies de creixement bacterià, els bacteris d’E. coli van arribar a cobrir tota la superfície de la placa gegant. Els bacteris del centre de la placa eren els que van evolucionar fins a adquirir una resistència a l’antibiòtic 1000 vegades superior als bacteris que es trobaven a prop dels extrems (figura 4). Cada pas en aquest procés va ser captat en un vídeo que ha esdevingut famósw1.

Laboratori Kishony, Escola Mèdica de Harvard i Technion – Institut de Tecnologia d’Israel

Laboratori Kishony, Escola Mèdica de Harvard i Technion – Institut de Tecnologia d’Israel

Laboratori Kishony, Escola Mèdica de Harvard i Technion – Institut de Tecnologia d’Israel
Com que els investigadors van poder veure en l’avançament dels bacteris regió per regió, van poder agafar mostres dels bacteris en els punts crítics on s’havien generat els mutants. A través de la seqüenciació dels genomes d’aquests bacteris, van poder determinar de manera exacte els diferents canvis que es van produir en cada pas en l’evolució de l’adquisició de la resistència (figura 5).
Va resultar que moltes de les mutacions van tenir lloc múltiples vegades, de manera que sembla que algunes vies d’adquisició de resistència són més comunes que d’altres. A més, algunes mutacions es van produir en gens que aparentment no tenen cap relació amb els antibiòtics, aclarint un punt important en l’adquisició de resistència: la necessitat d’adaptar-se als antibiòtics és una càrrega en els processos metabòlics bàsics, de manera que en l’absència d’antibiòtics, els bacteris resistents sovint creixen de manera més lenta que els no-resistents. Aquestes mutacions addicionals són necessàries per a compensar la càrrega metabòlica, i aquesta podria ser una altra raó per la qual hi ha una pausa en el creixement en els límits de les diferents regions de la placa, on s’incrementen les concentracions d’antibiòtic.
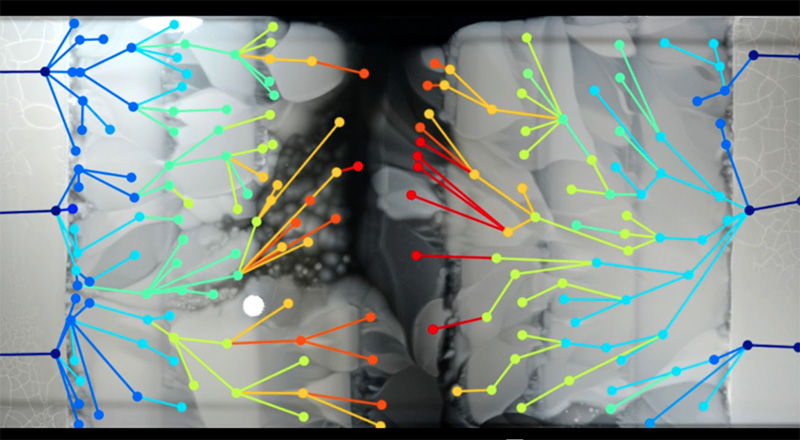
Laboratori Kishony, Escola Mèdica de Harvard i Technion – Institut de Tecnologia d’Israel
En un pas més enllà en aquest experiment, Baym i els seus col·laboradors van fer una altra observació crucial. Si la diferència de concentracions d’antibiòtic que generaven entre regions adjacents era molt més gran, cap dels mutants podia sobreviure en la regió següent i el creixement bacterià s’atura completament en el primer límit. Aquest fenomen s’explica pel mateix principi: quan la pressió selectiva dels antibiòtics en l’ambient és molt elevada, és gairebé impossible pels bacteris de créixer, perquè els mecanismes necessaris per a sobrepassar aquesta càrrega requereixen masses mutacions en una sola vegada – i per tant, els bacteris moren.
L’epidèmia de l’Ebola
Un altre exemple de l’evolució dels microbis patogènics que ha estat intensivament estudiat és un dels patògens humans més letal: el virus de l’Ebola. L’epidèmia d’Ebola 2013-2015 fou la més llarga i gran fins ara, amb 28 686 casos i 11 323 morts registrades el juny de 2016. La duració de l’epidèmia va coincidir amb l’arribada de tecnologia de seqüenciació de gens ràpida que va poder ser implementada en el camp, permetent als investigadors traçar l’evolució d’aquest virus a mesura que aquest infectava nous pacients.
Els científics que estudiaven l’Ebola, dirigits pels genetistes Dr Pardis Sabeti (Universitat de Harvard), Dr Jeremy Luban (Universitat de Massachussets Escola de Medicina) establerts als EUA i el Dr Andrew Rambaut (Universitat d’Edimburg), van construir un ‘arbre genealògic” del virus a mesura que s’escampava a través de l’oest de l’Àfrica durant dos anys (Park, 2015). Cada branca de l’arbre representa un nou set de mutacions que permeté al virus créixer i multiplicar-se millor que altres soques. Comparant els genomes virals recollits de pacients en diferents països i en diferents moments, l’equip va establir que la principal variant del virus es va originar a Guinea – o que només fou capaç d’escampar-se després d’adquirir cinc noves mutacions. Mentre s’escampava des de Guinea fins a Sierra Leone al maig de 2014, una nova mutació aparegué en aquesta regió (Diehl et al., 2016)w2. Fou trobada en un únic pacient, però aquesta va ser tan exitosa que el 97% dels més de 200 genomes de l’Ebola analitzats en l’estudi descendeixen d’aquest mutant. Aquest és una història d’èxit remarcable d’una soca que havia emergit només dos anys abans (figura 6).
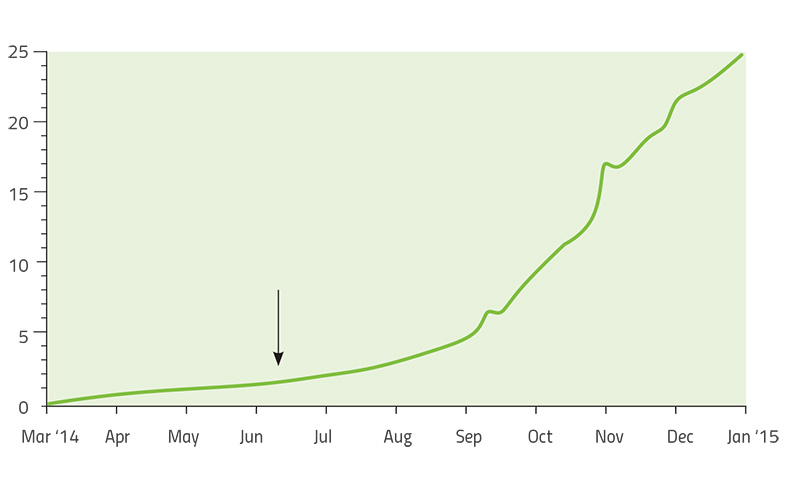
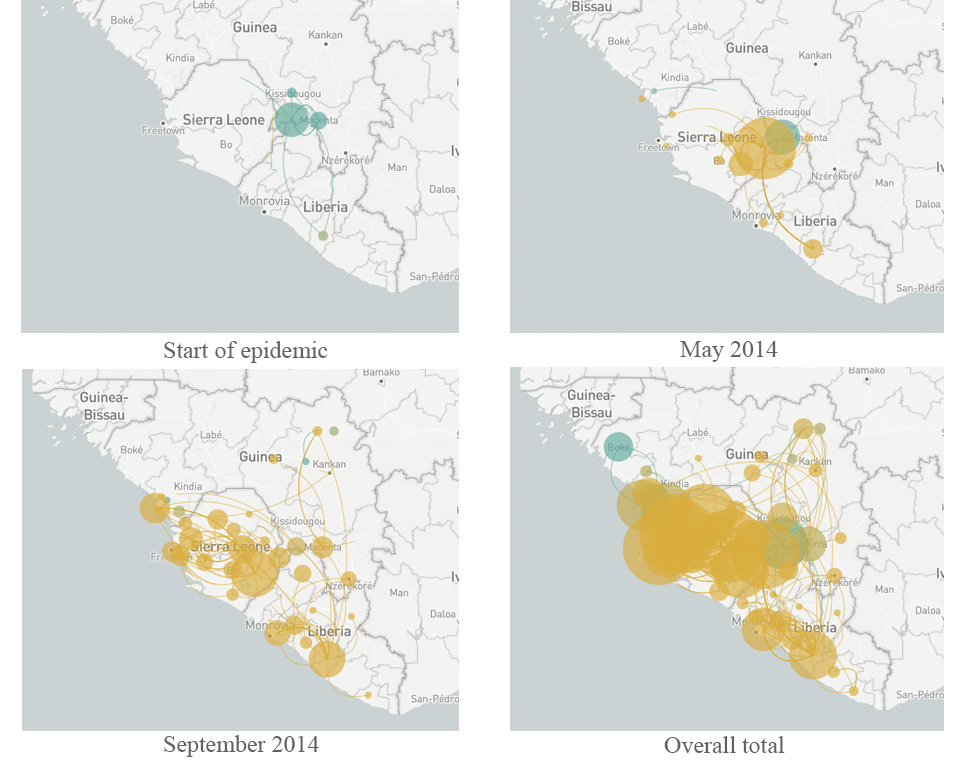
(Font: nextrain.org)
Encara més important, l’estudi va revelar que alguns gens en el genoma del virus de l’Ebola van canviar molt més del que era esperable, incloent aquells per a molècules proteiques localitzades a la part externa del virus. Es creu que questes proteïnes víriques són una diana dels anticossos humans que lluiten contra la infecció. Així doncs, canvis ràpids en aquestes proteïnes ajuden al virus a evitar la seva neutralització per part de l’hoste – un patró que és, per tant, conduït per selecció natural. Aquests gens han esdevingut un focus prometedor per a la recerca amb l’objectiu de combatre l’expansió del virus en una futura epidèmia. El fet d’esbrinar més coses sobre els mecanismes d’evolució també està ajudant als investigadors a desenvolupar estratègies efectives per a combatre els patògens en un futur.
References
- Bryk J (2017) Evolution in action: the 67 000-generation experiment. Science in School 41: 24-29.
- Park D et al. (2015) Ebola virus epidemiology, transmission, and evolution during seven months in Sierra Leone. Cell 161 1516-1526. doi: 10.1016/j.cell.2015.06.007
- Diehl W E et al. (2016) Ebola virus glycoprotein with increased infectivity dominated the 2013–2016 epidemic. Cell 167 1088–1097. doi: 10.1016/j.cell.2016.10.014
Web References
- w1 – Mira el video creat per Michael Baym i els seus col·laboradors mostrant l’expansió d’E. Coli a través de les regions que presenten un increment de les concentracions en la ‘megaplaca’.
- w2 – Mira una animation mostrant l’expansió del virus de l’Ebola, i com la nova soca que va emergir al voltant del juny de 2014 va afectar aquesta expansió.
Resources
- Visita la pàgina web The Atlantic per a veure un article accessible sobre l’experiment de Baym i les seves implicacions..
- Llegeix l’article científic en la revista Science que relata l’experiment de Baym:
- Baym M et al. (2016) Spatiotemporal microbial evolution on antibiotic landscapes. Science 53: 1147-1151. doi: 10.1126/science.aag0822
- Mira la xerrada de l’investigador Pardis Sabeti TED talk sobre combatre l’Ebola.
- Llegeix la publicació en un bloc sobre la feina de vigilància de l’Ebola, amb links a mitjans de comunicació sobre la investigació i tecnologia que hi ha darrera.
- Es pot trobar una guia sobre l’Ebola a Médecins Sans Frontières website.
Review
Un dels principals problemes a l’hora d’ensenyar l’evolució és la dimensió del temps. Degut que la vida humana és curta compara amb el marc temporal en el qual es produeixen els grans processos evolutius, el concepte de canvi al llarg del temps és una dificultat bàsica per a entendre l’evolució Així doncs, exemples de processos evolutius que tenen lloc durant un període de temps compatible amb la percepció humana ofereix oportunitats per superar aquests problemes.
L’experiment descrit en aquest article ofereix una bona visió de com els elements del temps i les mutacions afecten l’evolució. Els mestres podrien utilitzar aquest article per a explicar l’evolució, demostrant que l’evolució ocórrer durant un període de temps curt. Aquest article també és útil per a explicar el rol central que tenen les mutacions durant l’evolució, com a font de diversitat que després és intervinguda per la selecció natural. Donat que l’experiment inclou una idea familiar – l’ús d’antibiòtics – és fàcil d’entendre.
Panagiotis K Stasinakis, mestre de biologia, 4t Institut de Zografou, Grècia





