Supporting materials
Analisi delle impronte genetiche: una storia forense (Word)
Analisi delle impronte genetiche: una storia forense (Pdf)
Download
Download this article as a PDF

Traduzione di Daniela Caleppa. Nelle popolari serie TV poliziesche, le impronte genetiche sono comunemente usate per identificare i criminali. Sara Müller e Heike Göllner-Heibült hanno visitato la scena del crimine.
L’idea di distinguere gli individui a seconda delle loro caratteristiche genetiche non è nuova. Scoperta nel 1900 da Karl Landsteinerw1, la suddivisione del sangue nei gruppi sanguigni del sistema AB0 fu il primo marcatore genetico ad essere usato nelle scienze forensi e fu successivamente integrato dal sistema MN (1927) e dal fattore Rh (1937).
Tuttavia, anche analizzando simultaneamente i tre sistemi che determinano i gruppi sanguigni, si otterrebbero risultati identici in un soggetto ogni dieci; è proprio questa uguaglianza a rendere le trasfusioni sanguigne possibili. Per le scienze forensi, però, ciò è uno svantaggio:i risultati possono dimostrare che il campione di sangue prelevato non viene dal sospettato X, ma non possono provare con sicurezza che esso provenga davvero dalla Persona Sospetta Y.
Negli anni Settanta e Ottanta vi furono progressi nell’analisi di differenti forme di enzimi (isoenzimi) nei globuli rossi e nel siero sanguigno. La certezza che il campione provenisse dal sospettato dipendeva dal numero di proteine analizzate (solitamente quattro); questa sicurezza è denominata potere di discriminazione. Il potere di discriminazione fornito dalla combinazione di queste tecniche era ancora pari a 1:1000, sicuramente meglio dell’1:10 ottenuto dall’analisi dei gruppi sanguigni, ma ancora non sufficiente. Per avere un maggior potere di discriminazione, si ebbe bisogno di guardare più da vicino la nostra composizione genetica.
Il genoma umano è composto da 46 cromosomi accoppiati, di cui una metà proviene dal corredo cromosomico dalla madre e l’altra metà dal padre. L’essere umano ha quindi una coppia di ciascun cromosoma (eccetto i cromosomi sessuali maschili) e due esemplari dello stesso gene.
Il componente principale dei cromosomi è l’acido desossiribonucleico (DNA), che contiene le informazioni necessarie alla produzione delle proteine indispensabili alla vita. In realtà , soltanto circa il 4% dei tre miliardi di coppie di basi (bp, dall’inglese base pair) codifica per proteine; il resto è solo un “riempitivo”, che consiste in sequenze ripetute, organizzate a grappoli. Se si compara il DNA di due esseri umani, la maggior parte di esso è identico; la variabilità si trova soprattutto in queste sequenze ripetute.
Soggetti differenti possono avere diverse ripetizioni di queste sequenze: una persona può arrivare ad averne cinque in una determinata regione del DNA (locus), un’altra può averne sette. Utilizzando dei campioni, ad esempio sangue o sperma, si possono analizzare le sequenze ripetute in svariate regioni del DNA; questo processo è denominato analisi delle impronte genetiche. Proprio come le impronte digitali, le impronte genetiche possono essere usate per distinguere gli individui gli uni dagli altri.
Sebbene il termine “impronta genetica” (o profilo genetico) sia di uso comune, non è sempre noto come esso comprenda due tecniche molto diverse, una sola delle quali è utilizzata nelle moderne scienze forensi.

La prima metodologia di analisi delle impronte genetiche fu ideata nel 1984 da Alec Jeffreysw2, utilizzando sequenze note come DNA minisatellite ipervariabile (VNTR, da variable number tandem repeat, come ad esempio la sequenza D1S80, (AGGACCACCAGGAAGG)n). Queste sequenze di 10-100 bp per ripetizione, possono essere analizzati usando gli enzimi di restrizione, che lavorano come forbici molecolari per tagliare il DNA in sequenze definite (sequenze di riconoscimento). Nel nostro intero genoma, una sequenza di riconoscimento con 6 bp ricorrerà all’incirca 730 000 volte.
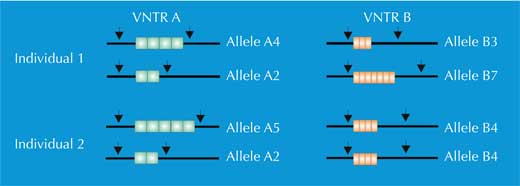
Ciò significa che il taglio del genoma con particolari enzimi di restrizione può fornire circa 730 000 frammenti di restrizione di diversa lunghezza. Ed è proprio a questo punto che entra in gioco la VNTR: il numero di restrizioni di un particolare grappolo di VNTR si differenzia da un soggetto all’altro e di conseguenza varia anche anche la lunghezza del corrispondente frammento di restrizione (Figura 1). Questo fenomeno è chiamato polimorfismo della lunghezza dei frammenti di restrizione.
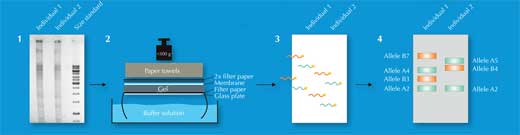
Solo alcuni dei 730 000 frammenti di restrizione si diversificano da un individuo all’altro – troppo pochi per essere individuati dall’occhio umano. Gli scienziati usarono quindi una tecnica chiamata Southern blotting, che permetteva di visualizzare solo le sequenze di interesse. Essi separavano i frammenti di restrizione a seconda della loro misura e li sottoponevano ad elettroforesi, utilizzando un gel su cui le molecole di DNA elettricamente cariche si muovevano a causa della corrente elettrica a cui erano sottoposte. La distanza percorsa era determinata dalla grandezza dei frammenti (Figura 2, Passo 1). In seguito, essi trasferivano il DNA su una membrana e vi applicavano una sonda radioattivamente marcata e complementare alle VNTR di interesse (Figura 2, Passo 2) . La sonda ibridizzata si poneva sulle sequenze corrispondenti (Figura 2, Passo 3) e, collocando il filtro su un film per raggi X, gli scienziati ottenevano un’immagine delle bande radioattivamente marcate, ciascuna rappresentata dalla diversa lunghezza di un frammento (Figura 2, Passo 4). Quest’immagine era l’impronta genetica.
Ma quante VNTR occorreva comparare per distinguere con certezza un individuo dall’altro? Nel caso gli scienziati scegliessero VNTR con una sufficiente variabilità (ad esempio la ripetizione D1S80, che può essere riprodotta dalle 15 alle 41 volte), essi avrebbero dovuto comparare quattro diverse VNTR per avere un potere di discriminazione pari a 1:1 milione (molto meglio del 1:10 offerto dal sistema AB0).
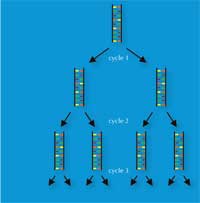
Nel 1983 Kary Mullis elaborò la PCR, ossia la tecnica della reazione a catena della DNA polimerasi(dall’inglese Polymerase Chain Reaction), che gli valse il Premio Nobel per la Chimicaw3, w4. Quest’invenzione, insieme alla scoperta, nei tardi anni Ottanta, delle ripetizioni in tandem semplici (STR), chiamate anche DNA microsatellite, con sequenze ripetute di 2-9 bp, spianarono la strada per la tecnica di analisi veloce delle impronte genetiche, usata oggi dalle scienze forensi.
La PCR permise che una regione di interesse del DNA (come ad esempio la STR a 4 bp, conosciuta come D18S51 (AGAA)n), fosse amplificata esponenzialmente, generando un miliardo di copie di una singola molecola di DNA in poche ore (Figura 3). Per gli scienziati forensi, ciò significò poter analizzare anche campioni estremamente piccoli – come ad esempio campioni di 30 cellule (Si veda la Tabella 1 per comparazione con l’analisi delle impronte genetiche basata sulla tecnica RFLP).
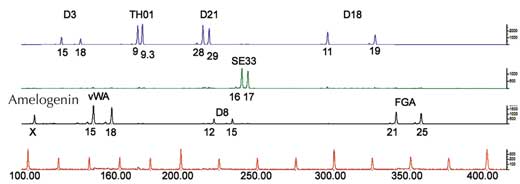
Per l’analisi PCR, occorrono ripetizioni STR affiancate a sequenze identiche in ogni essere umano (si denominano tali sequenze conservate). Quindi si utilizzano primer, ossia molecole brevi complementari alle sequenze conservate (geni 1134 e 1135 in Figura 4), per avviare la PCR. Una volta che il DNA è stato amplificato, può essere separato sia con elettroforesi su gel (Figura 5) sia, nelle moderne scienze forensi, tramite sequenziamento elettroforetico automatico (Figura 6), e visualizzato come impronta genetica.

L’essere umano ha due copie di ciascun cromosoma ed ha quindi anche due copie di ciascuna STR . Se, per ogni copia di STR, qualcuno avesse lo stesso numero di ripetizioni (come ad esempio lo stesso allele), l’analisi PCR rivelerebbe una sola dimensione di frammenti di DNA:la persona in questione sarebbe omozigote per l’allele STR (la freccia verde di Figura 5, corrispondente all’individuo 2 in Figura 4). Se i due cromosomi portassero alleli non identici per quella ripetizione STR, si vedrebbero due dimensioni di frammenti e si potrebbe affermare che la persona è eterozigote (la freccia rossa di Figura 5, corrispondente all’individuo 1 di Figura 4).
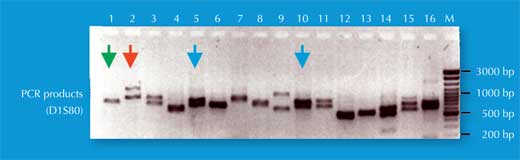
Se si analizza solamente una ripetizione STR, la possibilità che due individui non imparentati abbiano la stessa impronta genetica basata sulla PCR è alta, tra 1:2 e 1:100 (si vedano le frecce blu in Figura 5). Ciò è dovuto al fatto che le STR hanno minor numero di alleli e una più bassa eterozigosità delle VNTR usate per l’analisi delle impronte genetiche basata sulla RFLP. Per superare questo svantaggio, si analizzano simultaneamente ripetizioni STR multiple; con 16 STR, di prassi utilizzate dalle scienze forensi in Germania, si può ottenere un potere di discriminazione di 1:10 miliardi (equivalente cioè ad un solo individuo in tutta la popolazione mondiale; Figura 6).
| RFLP | PCR | |
|---|---|---|
|
Quantità di DNA di partenza |
30–50 µg |
Almeno 200 pg (circa 30 cellule) per un completo pattern di ripetizioni STR |
|
Sensibilità |
+ |
+++ |
|
Quantità di DNA richiesta per l’analisi |
Da giorni a settimane |
Non è necessario genoma completo; sono sufficienti anche solo prodotti di degradazione, vista la brevità delle sequenze coinvolte (la lunghezza totale della sequenza di una ripetizione STR, incluse ripetizioni multiple e sequenze affiancate, è approssimativamente di 50 – 500 bp) |
|
Tempo |
Da giorni a settimane |
Ore |
|
Potere di discriminazione per regione |
+++ |
+ |
|
Unità di ripetizione |
Da 10 a 100 bp |
Da 2 a 9 bp (nelle scienze forensi, principalmente 4 bp) |
|
Rilevamento automatico |
Impossibile |
Alto rendimento di elaborazione dei campioni |
|
Numero di regioni convalidate (importante se sono coinvolti parenti) |
Numero limitato |
Numero consistente |
|
Rischio di contaminazione |
+ |
+++ |
|
Sono richieste ulteriori misure di sicurezza? |
Sì (data la radioattività delle sonde) |
No (non è utilizzata alcuna sonda radioattiva) |

Ora sappiamo cos’è l’ analisi delle impronte genetiche, ma come si usa? Nelle indagini forensi è largamente utilizzata l’analisi delle impronte genetiche basata sulla PCR: essa permette alla polizia di escludere o identificare i sospetti sulla base del loro materiale genetico, come follicoli, epitelio, liquido seminale, saliva o sangue (in basso è possibile eseguire il download di una storia campionew5). Tuttavia la sola analisi delle impronte genetiche non è una prova sufficiente alla condanna, dato che parenti prossimi possono possedere impronte genetiche molto simili (ed i gemelli omozigoti le hanno solitamente identiche).

Inoltre, per complicare le indagini forensi internazionali, sebbene una norma europea preveda di analizzare 16 STR, ogni Paese può decidere quali ripetizioni analizzare, rendendo così i confronti molto più difficili.
Il metodo basato sulla PCR è anche usato per i test di paternità, nella diagnosi di molte malattie genetiche (come ad esempio il morbo di Huntington), nell’identificazione di vittime delle catastrofi, nell’elaborazione di alberi genealogici, nella ricerca di persone scomparse e nelle indagini su personaggi storici (ad esempio, l’ultimo Zar di Russia e la sua famiglia). In altri campi, esso può essere usato a scopi di salvaguardia (ad esempio per analizzare l’avorio confiscato), per l’analisi delle sostanze stupefacenti (analizzando sezioni di piante di cannabis), per controllare la qualità del cibo e dell’acqua (identificando microbi contaminanti), nella medicina (per rilevare infezioni virali, come l’HIV, l’epatite o l’influenza) e nelle indagini contro il bioterrorismo (per identificare ceppi microbici).
L’ analisi delle impronte genetiche basata sulla RFLP, sebbene sia ormai obsoleta, grazie ai vantaggi del metodo PCR (Tabella 1), è ancora usata per la classificazione di piante e animali in ricerche di base. Essa è particolarmente utile nel caso si possiedano informazioni insufficienti sul genoma delle specie – si ricordi che per il metodo PCR occorrono regioni altamente variabili tra individui, affiancate da regioni di riferimento di sequenze conosciute.
Isolare il DNA a scuola offre agli studenti un’esperienza entusiasmante, durante la quale essi realizzano di trovarsi di fronte alla codifica delle informazioni genetiche di un organismo: pochi filamenti di DNA, d’aspetto simile a fili di lana, precipitati in alcool. E’ semplice eseguire quest’esperimento a scuola, usando salivaw6 (o cellule epiteliali,da kit disponibili in commercio), piselli (Madden, 2006), pomodori, cipollew7 o estratto di timo di vitello (è consigliabile controllare se la scuola permetta di utilizzare quest’estratto)w8.

La PCR risultante da una specifica STR nel DNA umano, per esempio dalle regioni D1S80 o TH01, può essere analizzata a scuolaw6, usando, se necessario, kit in commercio ad un prezzo ragionevolew9. Se la scuola non avesse accesso ad un termociclatore, l’amplificazione del DNA può essere eseguita in tre recipienti d’acqua, sebbene questo processo risulti noioso e impegnativo.
Se questa strumentazione non fosse disponibile, esistono kit che simulano e semplificano l’intero processo di analisi delle impronte genetichew10. Essi contengono frammenti di DNA che simulano l’amplificazione di diversi alleli di una singola STR o VNTR. (Infatti, essi sono frammenti di restrizione del DNA di plasmide o fago lambda). Il DNA necessita di elettroforesi e di conseguente colorazione, in modo che gli studenti possano essere in grado di comparare sequenze di DNA “amplificate” di un campione di prova, con quelle di molti sospettati. Questo processo è sicuramente diverso dall’individuazione di ripetizioni STR amplificate con un sequencing elettroforetico automatizzato (ed inoltre non rappresenta accuratamente la visualizzazione delle VNTR con il Southern blotting, dato che il DNA è colorato direttamente sul gel), ma nondimeno esso dimostra i principi del processo analitico. Quando si utilizzano questi kit di simulazione, gli studenti devono essere ben coscienti che gli esperimenti danno l’impressione di una facile identificazione delle differenze tra un individuo e l’altro, mentre in realtà non è affatto così.
Le autrici voglio ringraziare Wolfgang Nellen per il suo contributo alla stesura dell’articolo e per aver messo a disposizione gratuitamente le istruzioni di Science Bridge.
Esse sono altresì grate a Shelley Goodman per il suo consiglio di utilizzare kit commerciali a scuola.
In un’intervista per Science in School, Alec Jeffreys parla della sua scoperta:
Hodge R, Wegener, A-L (2006) Alec Jeffreys interview: a pioneer on the frontier of human diversity. Science in School 3: 16-19. www.scienceinschool.org/2006/issue3/jeffreys
Queste istruzioni sono solitamente disponibili solo per i membri di Science Bridge, ma i lettori di quest’articolo possono richiederle gratuitamente a sara.mueller@sciencebridge.net
Per conoscere come isolare il DNA di un pomodoro (istruzioni in inglese), si veda: http://ucbiotech.org/edu/edu_aids/TomatoDNA.html
Klug WS, et al. (2008) Concepts of Genetics 9th edition. San Francisco, CA, USA: Pearson. ISBN: 9780321524041
Goodwin W, Linacre A, Hadi S (2010) An Introduction to Forensic Genetics. Chichester, UK: Wiley-Blackwell. ISBN: 978-0470710197
Wallace-Müller K (2011) The DNA detective game. Science in School 19: 30-35. www.scienceinschool.org/2011/issue19/detective
Patterson L (2009) Getting a grip on genetic diseases. Science in School 13: 53-58. www.scienceinschool.org/2009/issue13/insight
L’idea di usare il DNA per identificare una persona è incredibile. L’impronta genetica di un individuo si basa su sequenze che non sono utilizzate nella codifica, come possono quindi essere create? E’ ciò che spiega questo articolo.
La possibilità di identificare i criminali da database di DNA fa sorgere molte domande: è etico conservare il DNA umano in tali database, oppure è una violazione dei diritti dell’individuo? Sarebbe meglio se ci fosse un’impronta genetica del DNA di ciascun essere umano, oppure soltanto di chi è stato arrestato? Quanto a lungo si dovrebbe conservare il profilo genetico di una persona?
Gli studenti potrebbero voler ragionare sulla possibilità che l’impronta genetica aiuti nella diagnosi di malattie genetiche, così come sul loro utilizzo nella lotta al bracconaggio e nella salvaguardia delle specie a rischio di estinzione. Essi potrebbero anche essere in grado di provarne la tecnica da soli, come esperimento reale o simulato.
Shelley Goodman, Regno Unito