Reconstruire un génome, c’est comme essayer de résoudre un puzzle Teach article
Comment les scientifiques font-ils pour assembler des informations génétiques issues de séquençages ? Amusez-vous avec ces deux puzzles pour le découvrir.
Reconstructing genomes is like solving puzzles
Caractériser la diversité microbienne qui vit sur et à l’intérieur de nous, nous permet de comprendre les rôles que ces microbes peuvent potentiellement avoir sur la santé humaine et certaines maladies. Les scientifiques utilisent la technologie du séquençage d’ADN afin d’analyser le matériel génétique de ces échantillons microbiens pour déterminer les espèces présentes.
Au lieu de fournir la séquence génomique complète d’un organisme provenant d’un échantillon microbien, le séquençage d’ADN produit des centaines voire des milliers de petits morceaux linéaires d’ADN microbien. Afin d’identifier le ou les organismes présent(s), les scientifiques utilisent souvent des outils informatiques qui leur permettent de combiner ces fragments d’ADN en fragments contigus (appelés contigs).
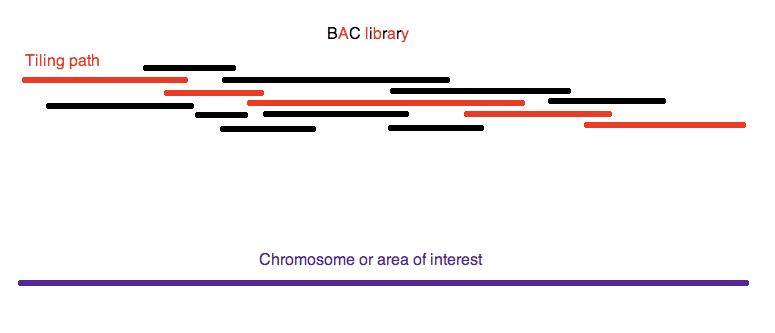
ceux-ci sont isolés puis découpés en fragments pour en faire un recueil appelé
librairie, dans le cas présent en utilisant des chromosomes artificiels bactériens (CABs).
En plus de la séquence d’intérêt, les CABs contiennent des éléments qui
permettent à ces fragments d’être copiés et séquencés. Cela donne une collection de
fragments de séquences qui se chevauchent et qui peuvent être alignés grâce à des
ordinateurs pour reconstruire la séquence originale.
Becchamm – Own work, CC BY-SA 3.0.
Cette ressource pédagogique permet aux enseignants d’introduire auprès des étudiants, à travers la métaphore du puzzle, ce concept de reconstruction génomique d’une simple bactérie et de communautés microbiennes complexes.
Introduction scientifique
Le séquençage ADN détermine la séquence d’acides nucléiques formant l’information héréditaire unique d’un organisme. En sortie, le séquençage génère des centaines voire des milliers de petits morceaux d’ADN microbien linéaires, ces-derniers sont des fragments du génome complet. L’étape suivant le séquençage d’ADN repose sur la combinaison (ou l’assemblage) de ces petits fragments en fragments plus longs contigus grâce à des outils informatiques.
La reconstruction du génome d’une simple bactérie
Le génome d’une simple bactérie est généralement:
- Circulaire
- Bicaténaire
- Plus ou moins long, mais de manière générale dans l’ordre du million de paires de bases.
Les technologies de séquençage d’ADN les plus communes (incluant le séquençage dit de second génération) génèrent des morceaux d’ADN qui sont:
- Linéaires
- Monocaténaires
- Courts (de 35 à 400 paires de bases).
Par conséquent, vous comprenez dans quelle mesure reconstruire un génome peut représenter un puzzle difficile : il faut construire son image complète à partir de ses toutes petites pièces.
Comment faire pour reconstruire la séquence génomique d’une simple bactérie ? Dans le plus simple des cas, notre organisme a déjà été séquencé et son génome a été déposé dans une banque de données publiques (telle que l’ EMBL-EBI’s European Nucleotide Archive, ENA). Dans ce cas, nous pouvons utiliser cette séquence pour nous aider à reconstruire le puzzle, de la même manière que si vous utilisiez la photo sur le dessus de votre boite de puzzle. Cette approche dite « de comparaison », identifie l’origine d’un fragment spécifique d’ADN en le comparant à une séquence connue.
Cependant, ceci est bien entendu une approche simpliste : à cause de leur taux de mutation élevé, le génome d’une bactérie est rarement totalement identique au génome de référence. C’est pourquoi, nous devons être prêt à accepter que la comparaison ne soit pas parfaite, et que les différences puissent indiquer les endroits les plus intéressants, dans la mesure où ils ont été proprement validés.
La reconstruction de génomes d’une communauté microbienne complexe
Quelles sont les difficultés en plus au cours de la reconstruction de génomes issus d’une communauté microbienne complexe telle que le microbiome intestinal?
- Il y a plusieurs génomes mélangés.
- Nous ne savons pas quelle séquence appartient à quel génome.
- Nous n’avons pas de génome de référence pour nous aider à reconstruire le génome de chaque bactérie dans la communauté.
- Même si les séquences ont une certaine « profondeur » (il y a beaucoup de pièces dans ce puzzle), il est fort possible que nous n’ayons pas toutes les séquences dans leur entièretés (il y a des trous qui forment des pièces manquantes).
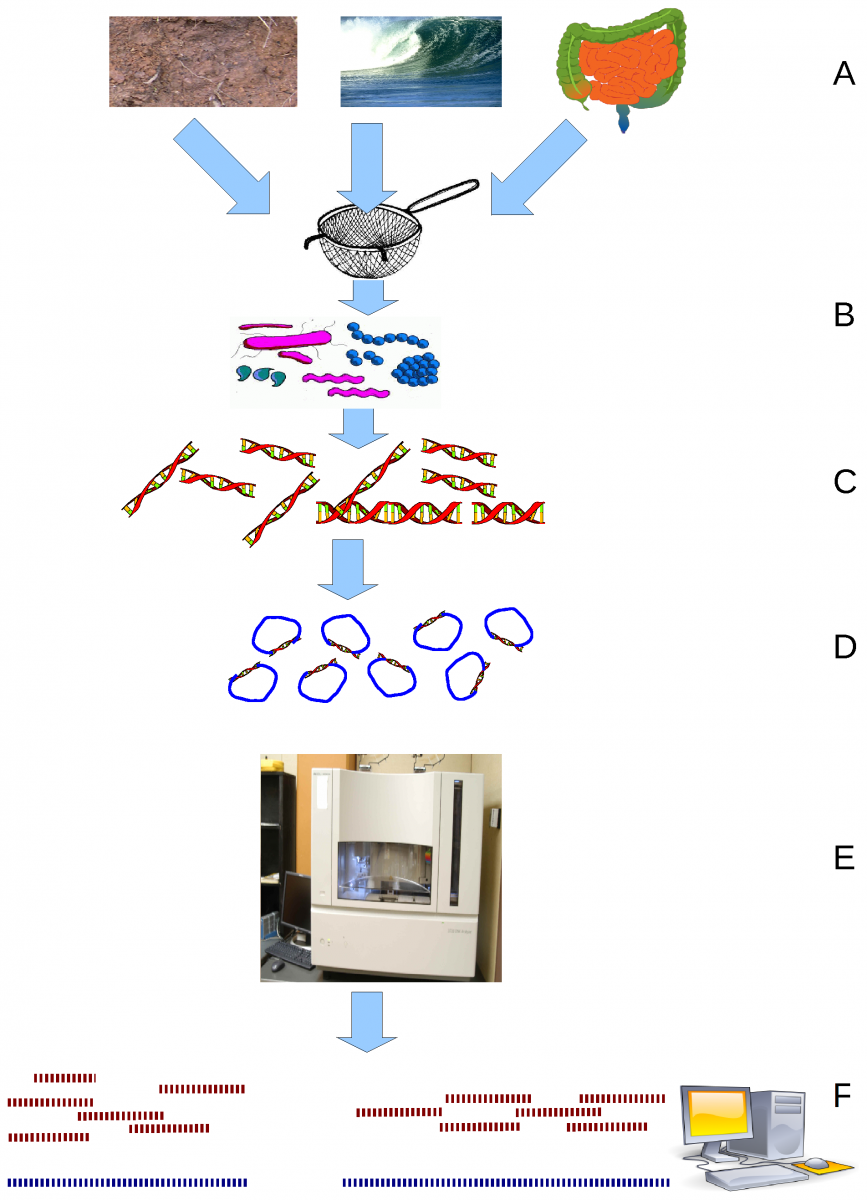
(A) Échantillonner l’habitat ; (B) Filtrer les particules,
typiquement par taille ; (C) Extraction de l’ADN et lyse ;
(D) Clonage et librairie ; (E) Séquençage des clones ; (F)
Assemblage des séquences.
John C. Wooley, Adam Godzik, Iddo Friedberg, CC BY 2.5, via Wikimedia Commons
Afin de pallier à ce problème plus complexe, il existe plusieurs stratégies qui ressemblent à ce que vous feriez instinctivement face à un puzzle:
- S’il y a des pièces du puzzle pour lesquelles nous avons une référence, nous débutons avec ça.
- Si des morceaux se ressemblent (proches génétiquement, ou dans la métaphore du puzzle, ils ont le même motif ou la même couleur), nous les regroupons.
- Si des morceaux s’imbriquent très bien (dans le jargon bio-informatique, ils peuvent être « assemblés »), nous supposons qu’ils vont ensemble.
- Si l’une des pièces a une fonction connue (dans notre métaphore, un coin du puzzle), nous tentons de deviner leur position.
Le problème de données manquantes
Cependant, comme nous l’avons mentionné précédemment, il est possible qu’il y ait des pièces manquantes qui soient nécessaires pour reconstruire l’image complète du puzzle. Étant donné que l’image est le point de départ de l’étude de la composition bactérienne de l’échantillon (lesquelles sont présentes ) et par la suite de leur possible fonction (ce qu’elles peuvent accomplir), imaginez l’impact qu’aurait ce manque de données sur sa résolution. Prenez une seconde pour réfléchir à l’impact qu’aurait une pièce manquante : outre le fait d’entraver la bonne compréhension de cette communauté microbienne, cela suppose que l’on peut décrire ce que l’on voit, mais qu’il est impossible de prétendre d’extraire des informations de ce qui est invisible. Plus simplement, si j’attrape des chaussettes dans mon tiroir et aucune n’est rouge, je ne peux pas en conclure que je n’ai aucune chaussette rouge. Pourquoi ? La complexité de la communauté microbienne est trop élevée pour nos capacités d’échantillonnage; et donc, il y aura systématiquement des données manquantes.
Consignes
Dans cette ressource pédagogique, les puzzles de génomes microbiens représentent des métaphores utiles pour illustrer comment les chercheurs étudient les données génétiques brutes produites par le séquençage ADN pour en extraire un génome bactérien unique (Puzzle 1) ou le génome d’une communauté complexe telle que le microbiome intestinal (Puzzle 2).
Les deux puzzles de cette activité sont en ligne : le premier est une figure de l’espèce bactérienne Escherichia coli et peut être utilisée comme métaphore pour la reconstruction d’un génome simple, le deuxième est une image de plusieurs microbes et peut être utilisée comme métaphore pour la reconstruction d’une communauté microbienne. Retrouver les deux puzzles ci-dessous:
Puzzle 2: Communauté microbienne
Vous trouverez ci-dessous un aperçu de quelques stratégies pour inclure les puzzles génomiques microbiens dans vos leçons de génomique.
- Introduisez les notions de génome bactérien, séquençage génomique et reconstruction d’un génome simple.
- Introduisez la métaphore du puzzle en proposant à vos étudiants de faire le Puzzle 1. Les étudiants peuvent s’aider de l’image de référence et se chronométrer pour connaître le temps nécessaire pour finir l’activité.
- Introduisez la notion de communauté microbienne et discutez des défis à surmonter pour reconstruire les différents génomes dans ce cas. Vous pouvez demander aux étudiants d’identifier les similitudes et différences lors de l’étape de reconstruction.
- Guidez les étudiants aux travers des défis décrits dans les points 1 à 4 de la section ‘Introduction scientifique’.
- Discutez avec les étudiants de comment ils résoudraient le puzzle sans image de référence.
- Demandez aux étudiants de faire le Puzzle 2. Cette fois, les étudiants vont devoir finir le puzzle sans l’image de référence et comparer le temps mis par rapport au Puzzle 1.
- Discutez avec les étudiants dans quelle mesure il serait difficile de finir le puzzle s’ils n’avaient pas toutes les pièces ? Aidez les étudiants à parler de a) la possibilité qu’ils se trompent sur le placement d’une pièce, b) la possibilité qu’ils ne puissent pas identifier un (ou plusieurs) microbes au sein de la communauté.
- Commencer à conclure que le temps supplémentaire nécessaire pour reconstruire le génome d’une communauté complexe par rapport à un génome simple est appelé « complexité de calcul ». La complexité de calcul décrit l’augmentation en complexité qui est elle-même due à l’augmentation du temps et de la puissance de calcul nécessaire (besoin de plus de puissance informatique que de main-d’œuvre) pour extraire la composition et la fonction d’une communauté microbienne en comparaison à une simple bactérie.
- Finissez par conclure que finir le puzzle, dans ce cas la reconstruction fiable du génome d’une communauté microbienne, n’est que le début de l’étude du microbiome ! Une fois que les génomes sont reconstruits, il est possible d’identifier les différentes espèces bactériennes. L’étape suivante suppose de caractériser en détail ces communautés, ceci est à la base de la recherche sur ces communautés microbiennes et leur interaction avec nous, humains.
Acknowledgements
Cet article a été adapté depuis l’article original de EMBL-ELLS Teaching Material.
Resources
- Craquer le code génétique avec cette activité éducative.
- Lisez cette étude sur l’ évolution d’Escherichia coli couvrant 67 000 générations.
- Apprenez en plus sur la découverte de la structure de l’ADN et comment l’extraire d’oignons ou de pois en classe.
- Qu’est-ce-que sont les mutations cancérigènes et comment sont-elles identifiées ? Découvrez les réponses avec cette activité éducative.
- Plus de ressources sur le microbiome humain.





