Rompecabezas del genoma microbiano Teach article
¿Cómo dan, los científicos, sentido a la información genómica a partir de los datos de secuenciación? Juega a estos dos divertidos puzzles en línea para descubrirlo.
Reconstruir genomas es como resolver rompecabezas
Caracterizar la composición de muchos de los microbios que viven dentro y fuera de nosotros ayuda a comprender las posibles funciones que estos microbios tienen en la salud y enfermedad humana. Los científicos usan la tecnología de secuenciación del ADN para analizar el material genético de muestras microbianas e identificar las especies presentes.
En vez de proporcionar la secuencia genómica completa de los organimos de una muestra microbiana, la secuenciación del ADN da lugar a cientos o miles de fragmentos de AND microbiano lineares y cortos de ADN. Para tratar de identificar el organismo(s), los científicos habitualmente usan estos fragmentos de ADN y los combinan formando segmentos contiguos (“contigs”) usando métodos informáticos.
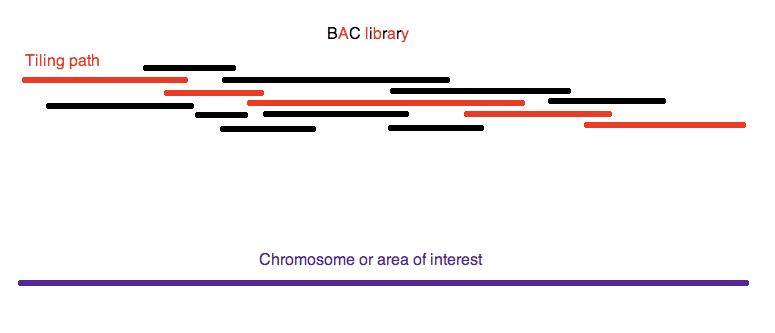
Becchamm/Wikimedia – Trabajo propio, CC BY-SA 3.0.
Este recurso educativo guía al professor sobre cómo usar una simple metáfora con un puzzle para mostrar a los estudiantes el concepto de reconstrucción del genóma de una sola bacteria y de complejas comunidades microbianas.
Introducción científica
La secuenciación de ADN determina la secuencia de ácidos nucleicos de la información hereditaria de un único organismo. Como resultado, se generan cientos o miles de pequeños fragmentos de AND microbiano lineal, que son fragmentos del ADN genómico completo. El siguiente paso tras la secuenciación del ADN, por lo tanto, precisa de combinar (ensamblar) esos fragmentos en fragmentos de ADN contiguos (contigs) usando métodos informáticos.
Reconstrucción del genoma de una sola bacteria
El genoma de una sola bacteria es por lo general:
- Circular
- De doble cadena
- De longitud variable pero, generalmente, de un tamaño del orden del millón de pares de bases
Las tecnologías de secuenciación de ADN comunmente usadas (denominadas secuenciación de segunda generación) generan fragmentos de ADN que son:
- Lineares
- De cadena simple
- Cortos (35 a 400 pares de bases)
Por lo tanto, puedes imaginar la tarea de reconstruir genomas como un problema en cierto modo similar a un rompecabezas complicado: necesitamos reconstruir una imagen completa a partir de sus piezas.
¿Cómo hacemos exactamente cuando pretendemos reconstruir la secuencia genómica de una sola bacteria? En los casos más sencillos, nuestro organismo ha sido ya secuenciado y la secuencia de su genoma ha sido depositada en una central de datos pública (como es la EMBL-EBI Archivo Europeo de Nucleótidos, ENA). En este caso, podemos usar esta secuencia para ayudarnos a reconstruir el “rompecabezas”, de la misma forma que harías al mirar la imagen de la tapa de la caja del rompecabezas. Este método se denomina “mapeo”- identifica de donde viene una pieza específica de ADN comparándola con una referencia conocida.
Recuerda que esto es, evidentemente, una simplificación del proceso: debido a la alta tasa de mutación, casí nunca un genoma bacteriano secuenciado es totalmente idéntico al del genoma de referencia. Debemos aceptar, por lo tanto, que el mapeo no será perfecto, y que los desajustes en sí mismos, si se prueban suficientemente, serían los puntos más interesantes del genoma.
Reconstrucción del genoma de comunidades microbianas complejas
¿Cuáles son los retos añadidos cuando se reconstruyen genomas de una comunidad microbiana compleja como el microbioma de tu intestino?
- Hay multiples genomas mezclados.
- No sabemos qué secuencia pertenece a qué genoma.
- No temenos un genoma de referencia para ayudarnos a reconstruir el genoma de cada una de las bacterias de la comunidad.
- Incluso si las secuencias tienen un cierto “detalle” (por ejemplo, recopilamos varias piezas del puzzle), probablemente no hayamos reunido todas las secuencias (por ejemplo, permaneceremos con piezas perdidas de la imagen completa).
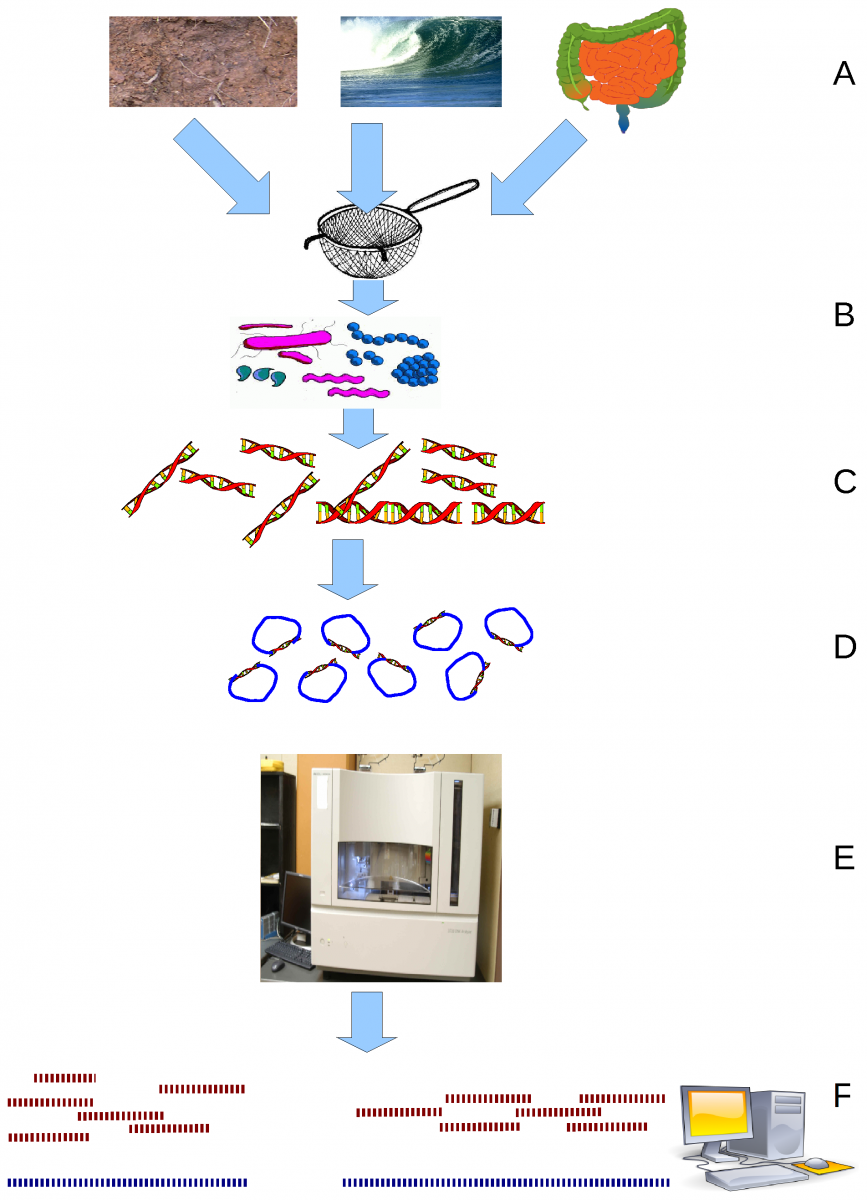
John C. Wooley, Adam Godzik, Iddo Friedberg/Wikimedia, CC BY 2.5
Para resolver este problema más complejo, hay varias estrategias que, una vez más, se asemeja a lo que harías de forma instintiva con un puzzle:
- Si hay alguna parte del puzzle de la que tengamos alguna referencia, construimos esa primero.
- Si alguna de las partes “se parece” (por ejemplo, son genéticamente similares, o, siguiendo con la metáfora del puzzle, quizás tengan el mismo color o patrón), las agrupamos.
- Si algunas partes encajan muy bien (en la jerga bioinformática, pueden “ensamblarse”), asumimos que son inseparables.
Si alguna de las partes tiene una función conocida (en la metáfora del puzzle, las esquinas o piezas de los bordes), tratamos de deducir a dónde pertenece.
El problema de los datos perdidos
Como se ha indicado, sin embargo, podemos no tener todas las piezas que necesitamos para reconstruir por completo la imagen. Ya que esta imagen es el punto de inicio para investigaciones sobre la composición bacteriana de la muestra (qué hay) y, por lo tanto, de su posible función (qué podrían estar haciendo), párate un minuto a pensar sobre el impacto de la parte perdida de los datos: a parte de obstaculizar el conocimiento completo de la comunidad microbiana, también debemos enteder que podemos describir lo que vemos, pero no podemos afirmar ninguna conclusión a partir de lo que no vemos. Plantéate, simplemente, que si cojo algunos calcetines de mi cajón y ninguno es rojo, no puedo concluir que no tengo calcetines rojos. ¿Por qué? La complejidad general de la comunidad microbiana es demasiado grande para nuestra capacidad de muestreo; por lo tanto, finalizaremos con datos que no están.
Instrucciones
En este recurso educativo, los rompecabezas de genomas microbianos son usados como metáfora para ilustrar como los investigadores se mueven desde datos genéticos brutos producidos por la secuenciación del ADN hasta una vision general del genoma de una sola bacteria (rompecabezas 1) o de una compleja comunidad como la muestra microbiana del intestino (rompecabezas 2).
La actividad del rompecabezas consiste en dos rompecabezas en línea: el primero muestra una versión en caricatura de una bacteria Escherichia coli y puede ser usado como una metáfora para la reconstrucción de un solo genoma. La segunda es un dibujo que representa múltiples microorganismos y puede ser usado como una metáfora para la reconstrucción de los genomas de los microorganismos de una comunidad. Puedes accede a los rompecabezas en los enlaces de abajo:
Rompecabezas 1: un solo microorganismo
Rompecabezas 2: comunidad microbriana
Aquí abajo, puedes encontrar un guión con distintas posibilidades para incluir los rompecabezas del genoma microbiano en tus clases de genómica.
- Presenta los conceptos de genoma bacteriano, secuenciación genómica y reconstrucción genómica de un solo genoma.
- Presenta la metáfora del rompecabezas pidiendo a los estudiantes hacer el rompecabezas 1. Los alumnos pueden usar la imagen de referencia como ayuda y pueden registrar el tiempo necesario para completar el rompecabezas.
- Presenta el concepto de comunidad microbiana y debate sobre el reto de reconstruir genomas microbianos, en este caso, pidiendo a los estudiantes que identifiquen similitudes y diferencias para reconstruir un único genoma.
- Guía a los estudiantes a través de los retos descritos en los puntos 1-4 en la sección de “introducción científica”.
- Debate con los estudiantes sobre cómo podrían resolver el rompecabezas sin imagen de referencia.
- Pide a los estudiantes hacer el rompecabezas 2. Esta vez, los estudiantes deben completar el puzzle sin mirar a la imagen de referencia, y comparar el tiempo que tardan en completar el rompecabezas con el tiempo que tardaron en resolver el rompecabezas 1.
- Debate con los estudiantes ¿cómo resolver el rompecabezas podría ser más complicado si no tuvieran todas las piezas del rompecabezas? Guía a los alumnos mencionándoles que a) deben quitar una pieza y b) deberían no identificar uno (o más) de los microorganismos de la comunidad.
- Comienza tu conclusión señalando que, el tiempo extra que se necesita para reconstruir gemomas de comunidades complejas en comparación con el genoma de una sola bacteria, es llamado “complejidad computacional”. La complejidad computacional describe el incremento de la complejidad, que es debida a la necesidad de aumentar el tiempo y la potencia computacional (ejemplo, más potencia del servidor que de mano de obra), requerida para recuperar la composición y función de una comunidad compleja comparada con la de una sola bacteria.
- Termina las conclusiones destacando que reconstruir el rompecabezas, por ejemplo reconstruyendo una imagen fiable de los genomas de la comunidad microbiana, ¡es solo el primer paso en el análisis del microbioma! Una vez que los genomas están reconstruidos, podemos identificar las especies bacterianas en la muestra. En el siguiente paso, esas comunidades pueden ser descritas y caracterizadas en detalle, constituyendo la base para investigar cuestiones sobre las comunidades microbianas y sus interacciones con nosotros, los humanos.
Acknowledgements
Este artículo fue adaptado a partir del artículo original publicado como EMBL-ELLS Material didáctico.
Resources
- Inicia el concepto de Código Genético con esta actividad didáctica.
- Lee un studio sobre el rastreo de la evolución de la bacteria Escherichia coli sobre 67 000 generaciones.
- Estudia el descubrimiento de la estructura del ADN y cómo extraerlo a partir de cebollas y guisantes en tus clases.
- ¿Qué son las mutaciones del cancer y cómo se identifican? Descubre la respuesta en esta actividad didáctica.
- Más recursos en el microbioma humano.





