Prendre le virus à son propre jeu Understand article
Traduit par C. Pugieux. L’équipe de Winfried Weissenhorn, qui travaille au sein du Laboratoire Européen de Biologie Moléculaire European Molecular Biology Laboratory de Grenoble, France, a découvert une nouvelle manière d’appréhender une classe de virus dangereux, en les enfermant dans…
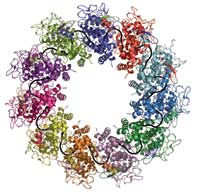
(de couleurs différentes) attachées
à une molécule d’ARN (en noire).
Image reproduite avec l’aimable
autorisation de Winfried Weissenhorn
C’est peut-être l’une des maladies des plus redoutées au monde. Une fois les symptômes typiques apparus (déséquilibre, bavement, peur de l’eau et paralysie), l’issue est quasiment fatale. Seules cinq personnes ont survécu à la rage, mais quatre d’entre elles ont subi des dommages neurologiques permanents. Bien que la vaccination et l’utilisation de traitements efficaces suite à l’exposition au virus aient largement contribué à la réduction de l’impact de la maladie en Europe et en Amérique du Nord, il n’existe toujours pas de traitement curatif. La rage continue de menacer sérieusement de nombreux pays en voie de développement, en faisant jusqu’à 70 000 victimes par an.
Fait inquiétant, le virus de la rage, ainsi que d’autres virus similaires, semble être en recrudescence actuellement dans les pays développés, se propageant aux personnes en contact avec des chauves-souris. La découverte d’un nouveau moyen de lutte contre ce virus par Winfried Weissenhorn, Rob Ruigrok et leurs collègues, arrive donc à point. Ces équipes de recherche, basées au sein du Laboratoire Européen de Biologie Moléculaire European Molecular Biology Laboratory de Grenoble, France, et de l’unité de recherche voisine travaillant sur les Interactions Virus-Cellule hôte Unit for Virus Host Cell Interactions (UVHCI), ont découvert la structure de la protéine qui protège le matériel génétique du virus contre le système immunitaire humain, jusqu’à ce que le virus ait l’opportunité de se multiplier. Il serait possible de concevoir des médicaments qui emprisonneraient le génome viral dans sa propre enveloppe de protection afin de prévenir sa réplication.
Mais les résultats obtenus ont une portée qui va bien au-delà du spectre de la rage. Il semble que de nombreux autres virus, dont le virus Ebola, Borna et de la rougeole, ont des enveloppes similaires protégeant leurs génomes, ce qui signifie que ces résultats pourraient également contribuer à la connaissance de l’origine de tels virus et de leur évolution. Le génome du virus de la rage, comme celui d’autres virus, se présente sous forme d’ARN simple brin, molécule semblable à l’ADN qui porte nos gènes. Mais contrairement à l’ADN, l’information codée par l’ARN viral ne peut pas être directement utilisée pour la fabrication de protéines nécessaire à la formation du virus. C’est une copie complémentaire de la séquence, une sorte de photographie négative chimique, qui est nécessaire. Ainsi, avant que le virus puisse produire les protéines qui constituent l’enveloppe protectrice de son génome, il doit convertir le brin négatif de son ARN en brin positif qui, lui, peut être traduit en protéines. Le virus a également besoin de répliquer son génome afin de se multiplier.

Image reproduite avec l’aimable
autorisation de EMBL Photolab
Ces processus comportent de nombreux dangers pour le virus. Les cellules mammaliennes, incluant les cellules humaines, ont un système de défense qui attaque et détruit les ARN étrangers. C’est pour cette raison que le virus protège son génome vulnérable à l’intérieur d’une nucléocapside, sorte d’enveloppe de protection constituée d’une protéine appelée nucléoprotéine, qui le protège jusqu’à ce qu’il pénètre dans une cellule et prenne en otage la machine cellulaire afin de se répliquer et produire ses protéines virales. En plus de protéger le génome, la nucléoprotéine régule l’équilibre entre la production de protéines nécessaire à la réplication virale et le processus de réplication même, car les deux processus ne peuvent pas se dérouler simultanément. C’est pour cela que la nucléocapside joue un rôle clé dans la vie du virus.
Jusqu’à présent, cependant, les seules connaissances qu’avaient les scientifiques sur le fonctionnement des nucléoprotéines provenaient d’images floues de microscopie électronique, montrant comment les nucléoprotéines polymérisent sur le génome pour former les nucleocapsides. Mais ces images n‘ont révélée que peu d’informations sur la structure de la protéine elle-même. Pour en savoir plus, Rob Ruigrok du laboratoire UVHCI et Winfried Weissenhorn ont collaboré pour obtenir des cristaux de la nucléoprotéine afin de déterminer sa structure à l’aide de rayons X de haute intensité, disponibles au Service de Radiation du Synchrotron Européen, European Synchrotron Radiation Facility, également situé à Grenoble. Les cristaux sont des structures symétriques qui, lorsque soumis à des rayons X, produisent un spectre de diffraction très ordonné à partir duquel les scientifiques peuvent déduire la forme de la molécule avec précision. En déterminant la structure de la nucléoprotéine, les scientifiques pourraient concevoir des médicaments qui enfermeraient le génome viral dans sa propre enveloppe protectrice.
Le projet débuta dans le laboratoire de Rob, où son équipe étudiait déjà depuis le milieux des années 90 les nucléoprotéines d’un certain nombre de virus à ARN à brin négatif. Le virus de la rage s’est révélé être l’un des plus faciles à manipuler. Aurélie Albertini, une doctorante du laboratoire, avait réussit à produire la nucléoprotéine de la rage dans des cellules d’insectes en culture cellulaire. La protéine s’enroulait autour des molécules d’ARN des cellules hôtes, formant ainsi des anneaux de 9 à 13 protéines. L’équipe de Rob Ruigrok avait déjà découvert que ces protéines se comportaient comme des nucléocapsides miniatures, et des études de microscopie électronique avaient fourni des données précises sur leur structure. Il s’avéra que ces nucleoplasmides ressemblaient beaucoup au nucléoplasmide du virus de la rage et donc ce résultat pourrait permettre l’étude de la structure de la nucléoprotéine organisée autour de l‘ARN.
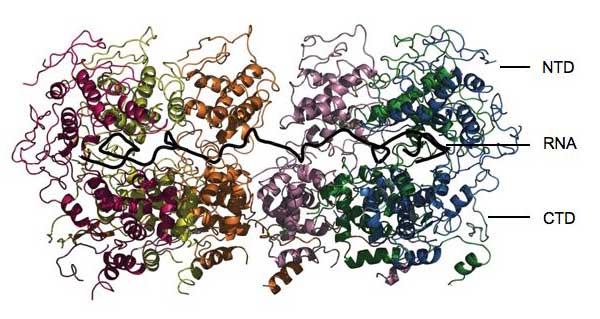
Image reproduite avec l’aimable autorisation de Winfried Weissenhorn
Aurélie commença à travailler sur la production de cristaux du complexe nucléoprotéine-ARN, projet sur lequel elle fut rejoint plus tard par Amy Wernimont, une post-doctorante du laboratoire de Winfried. Mais les molécules n’ont pas joué le jeu, Aurélie et Amy ont dû dépenser beaucoup d’énergie avant de finalement obtenir des cristaux qui ont permis à Amy de déterminer la structure avec une résolution de 4 Ångströms. Cette résolution est suffisante pour distinguer la structure de la protéine, mais insuffisante pour appréhender la séquence d’acides aminés qui constituent les blocs individuels de la protéine. Le problème venait du fait que les cellules qu’Aurélie utilisait ne produisaient la nucléoprotéine qu’en quantité infinitésimale.
Par chance, le service de la plate-forme de cristallographie à haut-débit, dirigé par Josan Marquez a permis de résoudre le problème, ne nécessitant que de faibles quantités de la nucléoprotéine. “La plateforme robotisée de cristallographie a été incroyable” se rappelle Winfried. “Si l’on avait eu besoin de produire l’échantillon en grande quantité, nous n’aurions pas pu optimiser ainsi les conditions de cristallisation”. L’équipe avait trouvé les meilleures conditions, la concentration précise de la protéine et le mélange compliqué de produits chimiques, qui leur ont finalement permis d’obtenir une résolution de 3.5 Ångströms, résolution suffisante pour construire un modèle de la structure de la protéine. Raimond Ravelli leur a également prêté main forte, en les assistant lors de l’acquisition des résultats et pour le réglage précis du temps d’exposition aux rayons X afin d’optimiser les résultats et d’offrir un soutient continue à différentes étapes de la recherche de la structure de la protéine.
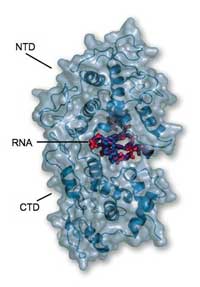
révèle que l’ARN est totalement
encerclé à la jonction des
domaines NTD (en haut) et CTD
(en bas) et n’est donc pas
accessible aux enzymes de
dégradation de l’hôte ou à la
polymérase.
Image reproduite avec l’aimable
autorisation de Winfried Weissenhorn
Les résultats ont révélé que la nucléoprotéine s’accroche complètement autour de l’ARN, ce qui le protège tels des bijoux de famille dans le coffre d’une banque. “Ainsi, aucune enzyme ne peut l’atteindre et l’attaquer”, raconte Winfried. La protéine est formée de deux unités fonctionnelles appelées domaines. Le premier domaine, appelé CTD, encercle un côté de l’ARN et se colle au domaine CTD d’une nucléoprotéine voisine, aidant à former une enveloppe hélicoïdale. Le second domaine, appelé NTD, se repose de l’autre côté de l’ARN et n’est pas vraiment en contact avec les nucléoprotéines voisines. La structure finale ressemble à une charnière protégeant l’ARN en l’entourant. “La nucléoprotéine va permettre à l’ARN de ne pas être reconnu par le système immunitaire de l’hôte” explique Winfried. “Mais comment le génome devient accessible pour sa réplication et sa traduction?”
La réponse se situe dans les structures à deux fils qui dépassent de chaque domaine. Ces structures pourraient agir comme des charnière, en poussant la région NTD en haut et en tournant la pince de nucléoprotéines, permettant aux enzymes virales d’accéder à de petites sections du génome. Une protéine particulière, appelée phosphoprotéine P, est peut-être impliquée. Elle relie l’ARN polymérase, enzyme nécessaire pour la répliquation du génome, à la nucléoprotéine, et peut s’accrocher à l’un des côtés de la charnière pour déplacer le NTD du chemin.
Ce mécanisme à charnière offre une stratégie de lutte contre les virus tel que celui de la rage en développant des médicaments qui le bloqueraient. “Si notre modèle, qui décrit l’ouverture de la charnière, est correct, nous pourrions forcer la charnière à rester fermée”, dit Winfried. Cela bloquerait la réplication virale. Enfermé ainsi à l’intérieur de son enveloppe de protéines, le génome viral serait ainsi rendu inoffensif et serait tôt ou tard éliminé par la cellule. La prochaine étape du développement d’un médicament serait une étude systématique de petites molécules chimiques qui auraient la capacité de s’accrocher sur la charnière, bloquant ainsi l’ouverture de la nucléocapside.
“Ces découvertes peuvent également éclairer sur l’évolution des virus à ARN à brin négatif”, dit Winfried. Des espèces apparentées de virus peuvent avoir des séquences génomiques très différentes, ce qui ne permet pas de conclure sur leurs liens évolutifs simplement en comparant leurs séquences. Pour les structures, par contre, c’est une autre histoire. Une même structure physique peut être obtenue à partir d’une variété de gènes et de séquences d’acides aminées. Donc même si des gènes évoluent et changent radicalement, les structures qu’ils codent peuvent révéler de forts liens d’évolution entre virus.
Les images de microscopie électronique, d’ARN d’autres virus à ARN brin négatif, tels que les virus de la rougeole, du virus de Marburg et la structure du cristal d’un virus Borna, suggèrent que leurs nucléoprotéines ont une structure à charnière articulée similaire. Cette observation suggère que ces virus utilisent une tactique similaire à celle du virus de la rage pour protéger leurs ARNs. Ces virus pourraient donc être la cible de médicaments qui bloqueraient l’ouverture de leur enveloppe de protection. “Cela suggère également qu’ils ont un ancêtre commun” dit Winfried. “A partir de l’analyse de leurs séquences, vous ne pourriez pas penser qu’ils ont un ancêtre commun” ajouta-t-il. “Je pense qu’il y avait probablement une nucléocapside ancestrale, qui a ensuite divergée comme les virus ont évolué pour infecter différents types de cellules”.
Resources
- Les travaux décrits dans cet article ont fait l’objet d’une publication scientifique en 2006 dans la revue internationale ‘Science’:Albertini AA, Wernimont AK, Muziol T, Ravelli RB, Clapier CR, Schoehn G, Weissenhorn W, Ruigrok RW (2006) Structure des cristaux du complexe virus de la rage of the rabies virus nucleoprotein-RNA complex. Science 313: 360-363.
- Pour en savoir plus sur le travail de Winfried Weissenhorn et de Rob Ruigrok, visiter le site Internet www2.ujf-renoble.fr/pharmacie/laboratoires/gdrviro/
Institutions
Review
Cet article souligne la nature de recherches scientifiques en tant qu’entité dynamique et continuelle/perpetuelle.
Comme chaque secret moléculaire du cycle de vie même du plus simple organisme est revele par l’utilisation de technologies sophistiquées, il y a des répercussions pour des avancées dans le domaine des traitements médicaux et du control des maladies.
Marie Walsh, République d’Irlande





