Supporting materials
Huella gen é tic a : una historia forense (Word)
Huella gen é tic a : una historia forense (Pdf)
Download
Download this article as a PDF

Traducido por Jorge J. Pérez-Maceira. En las series de detectives populares de televisión, la huella genética es generalmente usada para identificar criminales. Sara Müller y Heike Göllner-Heibült echan una mirada detrás de las escenas.
La idea de distinguir personas por sus características genéticas no es nueva. El descubrimiento en 1900, de los tipos sanguíneos ABO por Karl Landsteinerw1 fue el primer marcador genético en ser usado en ciencia forense, complementado más tarde con los grupos sanguíneos MN (1927) y el factor Rhesus (1937).
Incluso cuando analizamos los tres grupos sanguíneos simultáneamente, aún así, una de cada diez personas da resultados idénticos; esto hace que las transfusiones de sangre sean posibles. Para fines forenses, sin embargo, es una desventaja, los resultados pueden decir que la muestra de sangre no viene del sospechoso X, pero no pueden indicar con algún nivel aceptable de certeza que viene del sospecho Y.
Los avances que fueron realizados en los 70 y 80, con el análisis de diferentes formas de enzimas (isoenzimas) en glóbulos rojos y en suero sanguíneo. La certeza que la muestra realmente viniera del sospechoso dependía del número de proteínas analizadas (habitualmente cuatro); se denomina a dicha certeza poder de discriminación. El poder de discriminación que estas técnicas combinadas ofreció era todavía sólo de 1:10.000, mejor que el 1:10 del análisis de grupos sanguíneos, pero todavía no lo suficiente bueno. Para conseguir un poder mejor, necesitábamos echar un vistazo más de cerca en nuestra composición genética.
El genoma humano consiste en 46 pares de cromosomas: 23 de nuestra madre, 23 de nuestro padre. Por lo tanto, tenemos dos de cada cromosoma (excepto – en el caso de los hombres – del cromosoma sexual) y así dos copias de cada gen.
El componente principal de los cromosomas es el ácido desoxirribonucleico (ADN), el cual contiene la información para construir las proteínas necesarias para la vida. Sin embargo, de nuestros 3 mil millones de pares de bases (bp) (N.T.: Un billón en el original en inglés), sólo alrededor del 4% codifica para proteínas, el resto es a menudo simplemente “de relleno” consistente en secuencias repetitivas organizadas en grupos. Si comparas el ADN de dos humanos, la mayoría es idéntico, con la variabilidad encontrada principalmente en estas secuencias repetitivas.
Diferentes personas pueden tener diferentes números de repeticiones de estas secuencias: una persona puede tener cinco repeticiones en un locus de ADN específico (sitio); otra persona puede tener siete. Utilizando muestras, por ejemplo de sangre o semen, podemos analizar las secuencias repetitivas en varios loci de ADN; este análisis se denomina huella genética (genetic fingerprint). Como las huellas dactilares, las huellas genéticas pueden usarse para distinguir individuos.
Aunque el término “huella genética” (o perfil genético (genetic profiling)) es comúnmente usado, no todo el mundo es consciente de que en realidad abarca dos técnicas muy diferentes, sólo una de las cuales se utiliza comúnmente en la ciencia forense en la actualidad.

El primer método de huella genética fue inventado en 1984 por Alec Jeffreysw2, quién utilizó secuencias de ADN repetido denominadas Número variable de repeticiones en tándem (variable number tandem repeats o VNTR; por ejemplo, la secuencia D1S80, (AGGACCACCAGGAAGG)n). Estas secuencias, de 10-100 bp por repetición, pueden ser investigadas usando enzimas de restricción, los cuales trabajan como tijeras moleculares para cortar el ADN en secuencias definidas (secuencias de reconocimiento). En nuestro genoma completo, una secuencia de reconocimiento de 6bp tendrá lugar alrededor de 730.000 veces.
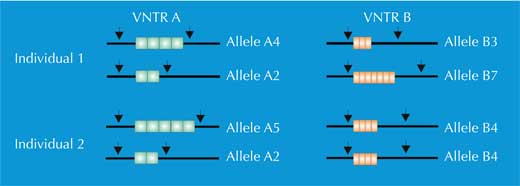
Esto significa que si cortas el genoma con una enzima de restricción particular, conseguirás alrededor de 730.000 fragmentos de restricción de longitudes variables. Y esto es donde las VNTR se vuelven importantes: el número de repeticiones en un grupo de VNTR particular puede varias entre individuos, lo que significa que la longitud del correspondiente fragmento de restricción variará entre individuos también (Figura 1). Denominamos a este fenómeno Polimorfismos de longitud de fragmentos de restricción (restriction fragment length polymorphism o RFLP).
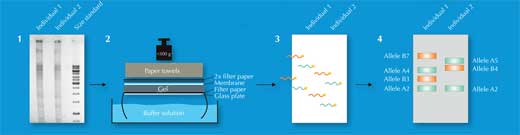
De los 730.000 fragmentos de restricción, sólo algunos diferirán entre individuos – demasiado pocos para ser detectados a ojo. En cambio, los científicos utilizan una técnica denominada Southern blotting, la cual deja sólo las secuencias de interés para ser visualizadas. Para hacer esto, separan los fragmentos de restricción según su tamaño con un gel de electroforesis, utilizando una corriente eléctrica para pasar las moléculas cargadas de ADN a través del gel. La distancia que viaja esta determinada por la medida del fragmento (Figura 2, Paso 1). Luego, transfieren el ADN a una membrana (Figura 2, Paso 2) y aplican una sonda etiquetada radioactivamente que es complementaria al(a los) VNTR(s) de interés. La sonda hibrida (engancha) con la secuencia coincidente (Figura 2, Paso 3) y la membrana se coloca sobre una película de radiografía, los científicos consiguen un imagen de las bandas etiquetas con radiactividad, cada cual representa una longitud diferente de fragmento (Figura 2, Paso 4). Esta imagen es la huella genética.
Así que, ¿Cuántos VNTR necesitan ser comparados para distinguir de forma fiable entre individuos? Si los científicos escogen VNTRs con suficiente variación (por ejemplo, D1S80, el cuál puede ser repetido cualquiera entre 15 a 41 veces), ellos sólo necesitan para comparar cuatro diferentes VNTR para tener un poder de discriminación de 1:1 millón – mucho mejor que el 1:10 ofrecido por el tipo sanguíneo ABO.
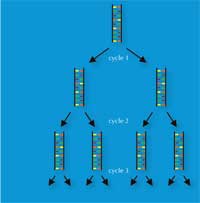
La invención de Kary Mullis, en 1983, de la reacción en cadena de la polimerasa (Polymerase Chain Reaction o PCR) le hizo ganar el premio Nobel en Químicaw3, w4. Esta invención, junto con el descubrimiento a finales de 1980 de los polimorfismos de repeticiones cortas en tándem (short tandem repeats o STR) – secuencias repetitivas de 2-9bp, denominadas microsatélites – pavimentó la manera para una técnica de huella genética de alta velocidad que los científicos forenses utilizan hoy.
La PCR habilita para que un locus de ADN de interés (por ejemplo, el STR de 4bp denominado como D18S51, (AGAA)n), sea amplificado exponencialmente, generando mil millones de copias de una sola molécula de ADN en pocas horas (Figura 3). Para los científicos forenses, esto tiene la ventaja de hacer el análisis de incluso muestras muy minúsculas – tan sólo 30 células (ver Tabla 1 para una comparación con la huella genética basada en RFLP).
Para análisis de PCR, necesitamos STR flanqueados por secuencias que sean idénticas en todos los seres humanos (decimos que estas secuencias están conservadas). Entonces utilizamos primers – moléculas cortas que son complementarias a la secuencia conservada del flanco (genes 1134 y 1135 en la Figura 4) – para iniciar la PCR. Una vez el ADN ha sido amplificado, lo podemos separar ya sea por gel de electroforesis (Figura 5) o, en las ciencias forenses modernas, por secuenciación automatizada electroforética (Figura 6), y visualizarlo como una huella genética.

Tenemos dos copias de cada cromosoma, así que también tenemos dos copias de cada STR. Si, para cada copia del STR, alguien tiene el mismo número de repeticiones (por ejemplo, el mismo alelo), el análisis de PCR revela sólo una medida del fragmento de ADN: la persona es homocigota para este alelo STR (flechas verdes en la Figura 5, correspondientes al individuo 2 en la Figura 4). Si los dos cromosomas llevan alelos no idénticos para este STR, vemos dos medidas del fragmento y decimos que la persona es heterocigota (flechas rojas en la Figura 5, correspondientes al individuo 1 de la Figura 4).
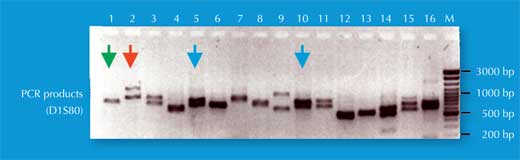
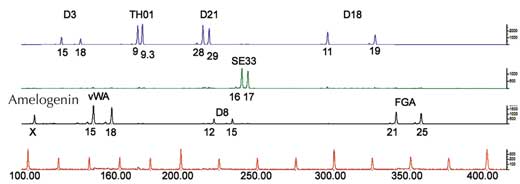
Si sólo analizamos un STR, la posibilidad de dos personas no relacionadas tengan la misma huella genética basada en PCR es alta – entre 1:2 y 1:100 (flechas azules en la Figura 5). Esto es porque los STR tienen menos alelos y más baja heterocigosidad que los VNTR utilizados en huellas genéticas basadas en RFLP. Para superar esta desventaja, analizamos múltiples STR simultáneamente, con 16 STR, como es común en casos forenses en Alemania, podemos conseguir un poder de discriminación de 1:10 mil millones (equivalente a una persona en la población mundial; Figura 6).
| RFLP | PCR | ||
|---|---|---|---|
|
Cantidad de ADN inicial |
30–50 µg |
Al menos 200 pg (sobre 30 células) para un completo patrón STR |
|
|
Sensibilidad |
+ |
+++ |
|
|
Calidad del ADN requerida para el análisis |
Genoma completo |
Ningún genoma completo es necesario; los productos de degradación también son suficientes debido a las secuencias cortas implicadas (longitud total de las secuencias de un STR, incluyendo las múltiples repeticiones y las secuencias flanqueantes, aproximadamente 50– 500 bp) |
|
|
Tiempo |
Días a semanas |
Horas |
|
|
Poder de discriminación por locus |
+++ |
+ |
|
|
Unidad de repetición |
10 bp a 100 bp |
2 bp a 9 bp (en casos forenses, principalmente 4bp) |
|
|
Detección automatizada |
No posible |
Posible el procesamiento de muestras de alto rendimiento |
|
|
Número de loci validados (importante si los parientes están implicados) |
Número limitado |
Número grande |
|
|
Riesgo de contaminación |
+ |
+++ |
|
|
¿Se requieren medidas de seguridad adicionales? |
Si (debido a las sondas radioactivas) |
No (ninguna sonda radiactiva) |

Ahora sabemos qué es una huella genética, pero, ¿Cómo se utiliza? La huella genética basada en PCR es ampliamente aplicada en las investigaciones forenses: permite a la policía excluir o identificar sospechosos en base a material genético como un folículo capilar, piel, semen, saliva o sangre (véase la historia que puede ser descargada a continuaciónw5). Una huella genética sola, sin embargo, no es suficiente evidencia para una condena, dado que parientes cercanos pueden tener huellas muy similares (y los gemelos homocigóticos normalmente los tienen idénticos).
Y para complicar las investigaciones forenses internacionales, a pesar de que hay una recomendación europea para analizar 16 STR, cada país puede decidir cuáles STR analizar, lo cual hace las comparaciones difíciles.

El método basado en PCR es también utilizado en humanos para los test de paternidad, diagnóstico de muchas enfermedades genéticas (por ejemplo, la enfermedad de Huntingdon), identificación de víctimas de desastres, seguimiento de árboles familiares, localización de personas desaparecidas e investigación de figuras históricas (por ejemplo, el último Zar de Rusia y su familia). En otros organismos, puede usarse con propósitos de conservación (por ejemplo, para analizar el marfil confiscado), en investigaciones de fármacos (por ejemplo, para analizar plantas de cannabis incautadas), para control alimentario o de calidad de agua (por ejemplo, para identificación de microbios contaminantes), en medicina (por ejemplo, para detectar infecciones virales como VIH, hepatitis o gripe) y en investigaciones de bioterrorismo (por ejemplo, para identificar cepas microbianas).
La huella genética basada en RFLP, aunque en gran parte obsoleta debido a las muchas ventajas del método PCR (Tabla 1), es todavía usada para clasificación de plantas y animales en investigación básica. Es particularmente útil cuando hay información insuficiente sobre el genoma de la especie – recordar que para el método de PCR, necesitamos regiones que varían ampliamente entre individuos y estén flanqueadas por regiones conservadas de secuencia conocida.
El aislamiento del ADN en la escuela da a los estudiantes un momento “guau” cuando se dan cuenta que están buscando en la información genética completa que codifica para un organismo – unas hebras de ADN como hilos de algodón que precipitan por alcohol. Es fácil de realizar en la escuela utilizando salivaw6 (o células epiteliales de kits comerciales disponibles), guisantes (Madden, 2006), tomates, cebollasw7 o timo de ternero (antes consultar las restricciones locales de usar timo de ternero en la escuela)w8.

La posterior PCR de un STR específico en ADN humano, por ejemplo D1S80 o TH01, puede ser realizada en la escuelaw6, usando kits comerciales disponibles de razonable preciow9 si es necesario. Si vuestra escuela no tiene acceso a un termociclador, el termociclado puede ser llevado a cabo en tres baños de agua, a pesar de que es tedioso y muy práctico.
Si este equipamiento no está disponible, hay kits que simulan y simplifican el proceso entero de huella genéticaw10. Estos kits contienen fragmentos de ADN que simulan la amplificación de diferentes alelos de un solo STR o VNTR (De hecho, son fragmentos de restricción de ADN del ADN de un plásmido o de una fago lambda.) El ADN requiere electroforesis y posterior revelado de modo que los estudiantes son capaces de comparar las secuencias “amplificadas” de ADN de una muestra de evidencia con los de varios sospechosos. Naturalmente, esto es muy diferente de detectar amplificando STR usando la secuenciación automatizada electroforética (y ni siquiera representan con exactitud la visualización de VNTR utilizando Southern blotting, como el ADN se tiñe directamente sobre el gel), pero sin embargo demuestra los principios del proceso analítico. Cuando se utilizan estos kit de simulación, los estudiantes deben ser conscientes de que los experimentos dan la impresión de que las diferencias entre los individuos pueden ser identificadas fácilmente, lo cual no es el caso.
A las autoras les gustaría agradecer a Wolfgang Nellen sus ideas sobre el artículo y por dejar las instrucciones de Science Bridge disponibles gratis.
Además, agradecer a Shelley Goodman por su asesoramiento en el uso de kits comerciales en la escuela.
En una entrevista con Science in School, Alec Jeffreys habla de su descubrimiento:
Hodge R, Wegener, A-L (2006) Alec Jeffreys interview: a pioneer on the frontier of human diversity. Science in School 3: 16-19. www.scienceinschool.org/2006/issue3/jeffreys
La afiliación a Science Bridge es necesaria para acceder a estas instrucciones, pero los lectores de este artículo las pueden pedir gratis a sara.mueller@sciencebridge.net
Para descubrir cómo aislar ADN a partir de tomates (instrucciones en Inglés), véase: http://ucbiotech.org/edu/edu_aids/TomatoDNA.html
Klug WS, et al. (2008) Concepts of Genetics 9th edition. San Francisco, CA, USA: Pearson. ISBN: 9780321524041
Goodwin W, Linacre A, Hadi S (2010) An Introduction to Forensic Genetics. Chichester, UK: Wiley-Blackwell. ISBN: 978-0470710197
Wallace-Müller K (2011) El juego del detective de ADN. Science in School 19. www.scienceinschool.org/2011/issue19/detective/spanish
Patterson L (2009) Nuevas piezas para entender el puzzle de las enfermedades genéticas. Science in School 13. www.scienceinschool.org/2009/issue13/insight/spanish
La idea de utilizar el ADN para identificar a un individuo en el mundo es alucinante. La huella genética de un individuo se basa en las secuencias que no son usadas en la codificación, pero ¿Cómo son estas huellas creadas? En este artículo se explica.
La habilidad para identificar a los criminales a partir de las bases de datos de ADN plantea preguntas importantes: ¿Es ético mantener el ADN humano en dichas bases de datos o se trata de una violación de los derechos humanos? ¿Debería haber una huella de ADN de cada persona, o simplemente aquellos que estén detenidos? ¿Cuánto tiempo debe el perfil de ADN mantenerse?
Los estudiantes tal vez deseen examinar cómo las huellas genéticas pueden ayudar a diagnosticar enfermedades genéticas, así como su aplicación en la lucha contra la caza furtiva y la extinción de las especies. Idealmente deben ser capaces de tratar la técnica por sí mismos, ya sea como un experimento real o simulado.
Shelley Goodman, Reino Unido
Huella gen é tic a : una historia forense (Word)
Huella gen é tic a : una historia forense (Pdf)
Download this article as a PDF