Evolution in Aktion: Pathogene Understand article
Pathogene, die die menschliche Gesundheit bedrohen unterliegen einer konstanten Evolution um unseren Abwehrmechanismen einen Schritt voraus zu bleiben. Wir können nun aber diese Veränderungen auf der genetischen Ebene verfolgen, sogar während sie noch ablaufen.
Eine der hauptsächlich wirkenden Kräfte der Evolution ist die natürliche Selektion – wobei individuelle Organismen mit einer bestimmten genetischen Ausstattung in einer gegebenen Umwelt mehr Nachkommen produzieren als andere, was in diesem Organismus zu Adaptationen führt, die es ihm erlauben zu überleben, während die anderen vergehen.
Science Photo Library/De Angelis, Maurizio
Wie von Darwin etabliert, trägt der Prozess der positiven natürlichen Selektion zur Diversität der rezenten Spezies bei, wobei Merkmale, welche die ‚Fitness’ eines Organismus erhöhen, in der Population dominant werden. Aber es ist eben auch ein Faktor in den Prozessen, die unser eigenes Überleben gefährden, nämlich im Rahmen der Geschwindigkeit von viraler Infektion und dem Auftreten antibiotischer Resistenz von bakteriellen Pathogenen. Allerdings können wir, dank genialer Laborexperimente (z.B. Bryk, 2017), und den Fortschritten in der Gensequenztechnologie, wie beispielsweise tragbare Gensequenzierungsmaschinen, diesen Prozess heute schnell und präzise nachvollziehen und zwar nicht nur im Labor, sondern praktisch überall auf der Welt. Das hat sich als hochgradig wertvoll für das Verständnis der Evolution krankmachenden Mikroorganismen herausgestellt.
In diesem Artikel konzentrieren wir uns auf zwei bemerkenswerte Studien die genau untersuchen, wie solche Pathogene sich konstant weiterentwickeln, die die Muster der genetischen Veränderungen und deren limitierende Faktoren entschlüsseln.
Antibiotikaresistenz im Scheinwerferlicht
Ein Musterbeispiel für die Evolution durch natürliche Selektion ist das Auftreten von Antibiotikaresistenz bei Bakterien. Die Verwendung von Antibiotika stellt einen Selektionsdruck auf eine Bakterienpopulation dar, welche sehr schnell mutieren und sich reproduzieren. Als Resultat verbleiben alle genetischen Varianten, die die Bakterien vor dem Antibiotikum schützen in der Population, während alle anderen Varianten verschwinden.
Der Forscher an der Harvard University und selbsternannte Bekämpfer der Antibiotikaresistenz Dr Michael Baym hat diesen Prozess anschaulich mit der Kamera festgehalten. Im Jahr 2015 baute Baym eine clevere Vorrichtung, um zu visualisieren, wie sich resistente Bakterien in einer Umgebung mit Antibiotika ausbreiten. Er benutzte eine riesige rechteckige Petrischale, 60 cm breit und 120 cm lang (zum Vergleich: eine Standard Petrischale misst 9 cm im Durchmesser), gefüllt mit Agar, einem gelatinösen Medium für das Bakterienwachstum, welches schwarz gefärbt wurde. Entscheidend war zudem, dass er das Medium mit steigenden Konzentrationen entlang der Petrischale tränkte (Abbildung 1), von Bereichen ohne Antibiotika an den Rändern bis hin zu 1000 Antibiotika-Einheiten in der Mitte. Diese Konzentration sollte ausreichen, jegliche Bakterien direkt abzutöten. Er stellte einen Scheinwerfer und eine Kamera über der Schale auf, sodass jede Bakterienkolonie als weißer Punkt vor dem schwarzen Hintergrund deutlich sichtbar war. Dann beimpfte er die Schale mit Escherichia coli (E. coli ) Bakterien an den Enden der Petrischale, wo keine Antibiotika vorhanden waren und wartete auf den Beginn des Prozesses (Abbildung 2).

Kishony Lab, Harvard Medical School and Technion – Israel Institute of Technology
Also, was passierte? Die E. coli konnten sich innerhalb des Oberfläche des Agars bewegen, wenn den Bakterien also die Nahrung in ihrer Umgebung ausgeht, wanderten sie in andere Stellen (Abbildung 3). Ihr Wachstum wurde allerdings durch das Antibiotikum in der Nachbarregion gehemmt, sodass lediglich diejenigen Bakterien mit Mutationen, die ein Überleben trotz Antibiotikakontakt erlaubten, sich weiter ausbreiten konnten. Die erste Region, die Antibiotika enthielt (in einer Konzentration von nur einer Einheit) wurde eingangs bei einzelnen Mutanten besiedelt. Diese vermehrten sich und ihre Nachkommen breiteten sich über die gesamte Region aus – bis sie selber auf die nächste Region mit einer höheren Antibiotikakonzentration trafen. An der Grenze stoppten die Bakterien solange, bis erneut neue Mutationen auftraten, die eine erhöhte Antibiotikatoleranz aufwiesen.
Dieser Prozess wurde solange an jeder Grenze wiederholt, bis, nach 11 Tagen des bakteriellen Wachstums, E. coli Bakterien die gesamte Oberfläche der riesigen Schale besiedelten, wobei die Bakterien in der Mitte der Schale eine Resistenz gegen Antibiotikaresistenzen entwickelt hatten, die 1000 mal höher waren als jene an den Rändern (Abbildung 4). Jeder Schritt wurde auf einem Video festgehalten, das mittlerweile Berühmtheit erlangt hatw1.

Kishony Lab, Harvard Medical School and Technion – Israel Institute of Technology

Kishony Lab, Harvard Medical School and Technion – Israel Institute of Technology

Kishony Lab, Harvard Medical School and Technion – Israel Institute of Technology
Während die Forscher die Ausbreitung der Bakterien von Einzelbild zu Einzelbild nachvollziehen konnten, waren sie auch in der Lage, Bakterien von kritischen Punkten zu entnehmen, an denen bestimmte Mutanten entstanden. Durch die Sequenzierung ihrer Genome, waren sie in der Lage, die Veränderungen in jedem evolutionären Schritt hin zur Resistenz nachzuweisen (Abbildung 5).
Es stellte sich heraus, dass viele der Mutationen viele Male auftraten, einige Wege zur Resistenz scheinen häufiger vorzukommen als andere. Zusätzlich traten Mutationen in Genen auf, die offensichtlich nichts mit dem Antibiotikum zu tun hatten, was einen entscheidenden Punkt auf dem Weg zur Resistenzentwicklung verdeutlicht: die Notwendigkeit der Anpassung an das Antibiotikum stellt eine Herausforderung an basale Stoffwechselprozesse dar. In Abwesenheit von Antibiotika wachsen resistente Bakterien oft langsamer als nicht resistente Vertreter. Die zusätzlichen Mutationen wurden gebraucht, um die stoffwechselphysiologische Mehrbelastung zu kompensieren, was ein weiterer Grund für die Wachstumspausen an den Grenzen zwischen zwei Bereichen sein könnte.
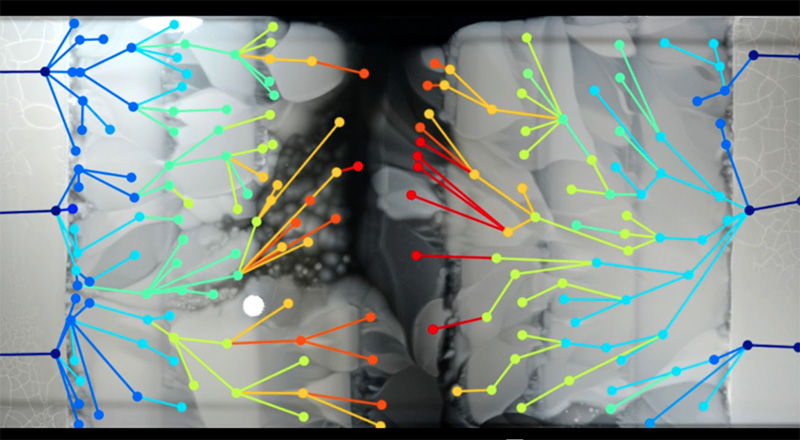
Kishony Lab, Harvard Medical School and Technion – Israel Institute of Technology
In einem weiteren Schritt dieses Experiments machten Baym und seine Kollegen eine weitere bahnbrechende Beobachtung. Wenn sie die Differenz zwischen der Antibiotikakonzentration in angrenzenden Regionen deutlich erhöhten, konnten keine Mutanten in der nächsten Region überleben und das Bakterienwachstum stoppte vollständig an der ersten Grenze. Dieses Phänomen kann mithilfe desselben Prinzips erklärt werden: Wenn der Selektionsdruck durch Antibiotika in der Umgebung sehr groß ist, ist es für die Bakterien nahezu unmöglich, überhaupt zu wachsen, weil die Mechanismen, die benötigt werden, die Herausforderung zu meistern, zu vieler Mutationen zur gleichen Zeit bedürfen – also sterben die Bakterien.
Die Ebolaepidemie
Ein weiteres Beispiel für die Evolution eines mikrobiellen Pathogens, das intensiv studiert wurde, ist eines der tödlichsten Pathogene für den Menschen: das Ebolavirus. Die Ebola-Epidemie von 2013-2015 war die längste und größte bislang mit 28 646 Fällen und 11 323 Toten bis einschließlich Juni 2016. Die Dauer der Epidemie und das Aufkommen der schnellen Gensequenzierungstechnologie im Feld erlaubte es Forschern die Evolution der Virus nachzuverfolgen während es neue Patienten infizierte.
Angeführt von den US-Genetikern Dr Pardis Sabeti (Harvard University), Dr Jeremy Luban (University of Massachusetts Medical School) and Dr Andrew Rambaut (University of Edinburgh) erstellten die Forscher einen Stammbaum des Virus während es sich über zwei Jahre über Westafrika ausbreitete (Park, 2015). Jeder Ast des Stammbaums repräsentiert ein neues Set an Mutationen die es dem Virus erlaubten besser als andere Stämme zu wachsen und sich zu vermehren. Durch den Vergleich der viralen Genome, gesammelt von Patienten von verschiedenen Ländern zu verschiedenen Zeiten, ermittelten die Forscher, dass die Hauptvariante des Virus aus Guinea stammte – und dass es sich erst ausbreiten konnte, nachdem es fünf neue Mutationen erlangt hatte. Als es sich 2014 von Guinea nach Sierra Leone ausbreitete, trat eine weitere Mutation auf und wurde in jener Region viel häufiger (Diehl et al., 2016)w2. Die neue Mutation wurde anfangs bei nur einem Patienten beobachtet, war jedoch so erfolgreich, dass 97% der mehr als 200 Ebola-Genome, die in der gesamten Studie untersucht wurden, Nachkommen dieses Mutanten waren. Dies ist eine bemerkenswerte Erfolgsgeschichte einer Rasse die vor gerade mal zwei Jahren auftauchte (Abbildung 6).
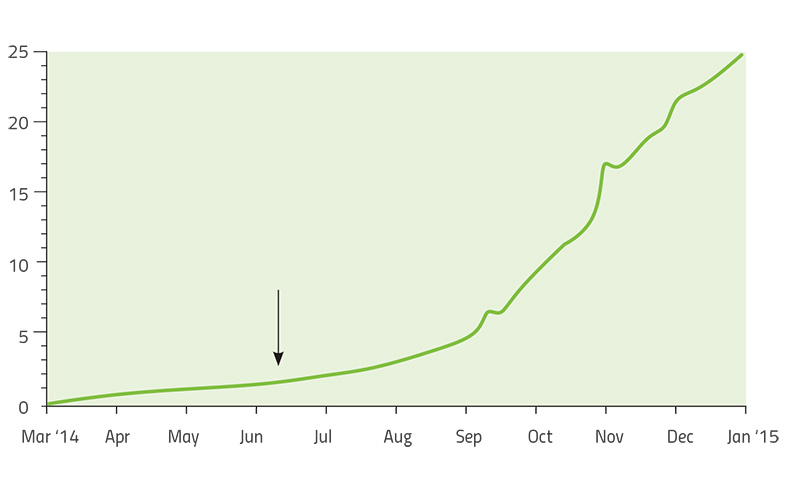
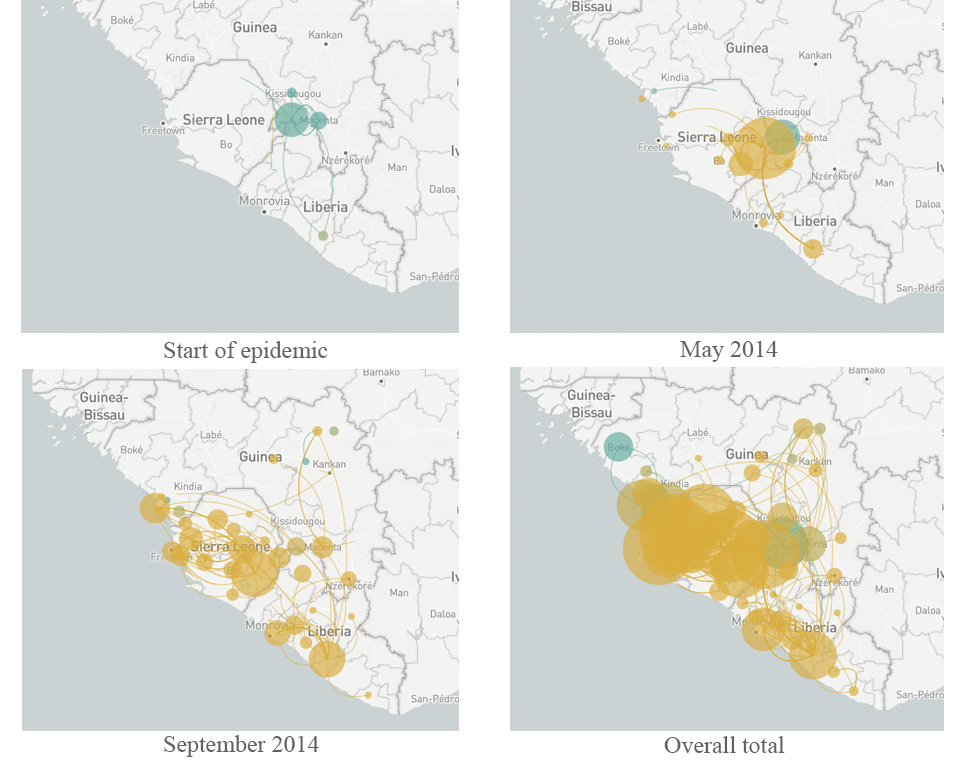
(Source: nextstrain.org)
Von noch größerer Bedeutung ist der Befund der Studie, dass sich einige Gene im Ebola-Genom stärker verändert hatten, als angenommen, darunter jene für die Proteinmoleküle auf der Außenseite des Virus. Diese viralen Proteine werden als Ziel für die menschlichen Antikörper angenommen, die eine Infektion bekämpfen. Plötzliche Veränderungen an diesen Proteinen hilft dem Virus also, die Neutralisation durch den Wirt zu vermeiden – ein Muster, das folglich durch die natürliche Selektion angetrieben wird. Diese Gene sind mittlerweile ein vielversprechender Ansatzpunkt für die Entwicklung von Maßnahmen gegen die Ausbreitung des Virus in möglichen zukünftigen Epidemien geworden. Mehr über die Mechanismen der Evolution herauszufinden hilft also den Forschern auch bei der Entwicklung von effektiven Strategien zur Bekämpfung von Pathogenen in der Zukunft.
References
- Bryk J (2017) Evolution in Echtzeit – das 67000 Generationen- Experiment. Science in School 41.
- Park D et al. (2015) Ebola virus epidemiology, transmission, and evolution during seven months in Sierra Leone. Cell 161 1516-1526. doi: 10.1016/j.cell.2015.06.007
- Diehl W E et al. (2016) Ebola virus glycoprotein with increased infectivity dominated the 2013–2016 epidemic. Cell 167 1088–1097. doi: 10.1016/j.cell.2016.10.014
Web References
- w1 – Schauen Sie das video von Michael Baym und seinen Kollegen an, das die Ausbreitung von E. coli in Regionen mit steigender Antibiotikakonzentration auf der ‘Megapetrischale’ zeigt.
- w2 – Schauen Sie sich die animation zur Ausbreitung des Ebola-Virus an und erfahren Sie, wie der neue Stamm, der im Juni 2014 auftauchte, diese beeinflusste.
Resources
- Besuchen Sie die Website von The Atlantic für einen frei zugänglichenArtikel zum Experiment von Baym und seinen Konsequenzen.
- Lesen Sie das wissenschaftliche Paper in Science über das Experiment von Baym:
- Baym M et al. (2016) Spatiotemporal microbial evolution on antibiotic landscapes. Science 53: 1147-1151. doi: 10.1126/science.aag0822
- Schauen Sie sich Pardis Sabetis TED Talk über den Kampf gegen Ebola an.
- Lesen Sie einen Blogeintrag über die Überwachungsarbeit zu Ebola mit Links zu Medienberichten zur Forschung und Technologie, die dahinterstehen.
- Ein Ebola-Ratgeber kann auf der Website der Ärzte ohne Grenzen gefunden warden.
Review
Eins der Hauptprobleme beim Unterrichten der Evolution ist die Zeitdimension. Weil das menschliche Leben im Vergleich zu den Zeiträumen in denen die großen evolutionären Prozesse stattgefunden haben, sehr kurz ist, stellt das Konzept der Veränderung über Zeiträume eine Schwierigkeit beim Verständnis der Evolution dar. Beispiele evolutionärer Prozesse, die in einer Zeitspanne ablaufen, die der menschlichen Wahrnehmung kompatibel ist, bietet Möglichkeiten, diese Probleme zu überwinden.
Das im Artikel beschrieben Experiment bietet eine guten Blick darauf, wie Zeit und Mutationen die Evolution beeinflussen. Lehrer können diesen Artikel verwenden, um Evolution zu unterrichten, indem gezeigt wird, dass Evolution in kurzen Zeiträumen stattfinden kann. Der Artikel ist auch hilfreich zum Verständnis der zentralen Rolle von Mutationen im evolutionären Prozess, als Quelle von Diversität auf die dann die natürliche Selektion wirkt. Und weil das Experiment ein bekanntes Phänomen umfasst – die Verwendung von Antibiotika – ist es leicht zu verstehen.
Panagiotis K Stasinakis, Biologielehrer, 4. High School of Zografou, Griechenland





