Usare i database biologici per insegnare l’evoluzione e la biochimica Teach article
Tradotto da Monica Menesini. Gli strumennti disponibili online possono essere usati per confrontare le sequenze proteiche e capire come si sono evoluti organismi diversi.
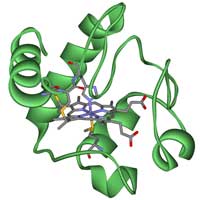
citocromo C
Immagine gentilmente
concessa da Klaus
Hoffmeier / Wikimedia
commons
Nel passato, gli scienziati conducevano le analisi evoluzionistiche confrontando le caratteristiche fisiche delle specie – cioè i fenotipi – rilevabili dai reperti fossili. Tutto è cambiato da quando è stato scoperto l’orologio molecolare.
Il concetto di orologio molecolare discende dall’osservazione che quanto più lungo è il tempo trascorso dalla divergenza di due specie rispetto all’antenato comune, tanto maggiori saranno le differenze tra i loro DNA e le loro proteine (per una revisione, vedere Bromham & Penny, 2003).
Confrontando geni o sequenze proteiche omologhe – in altre parole, le sequenze provenienti da due organismi con un antenato comune – possiamo misurare quanto tempo è trascorso da quando le due linee si sono separate. Per visualizzare questo processo si utilizza un albero filogenetico.
Per esaminare la similitudine tra due geni, bisogna conoscere le loro sequenze ed allinearle correttamente (Kozlowski, 2010). Ottenere queste sequenze non è più difficile come lo era una volta.
I vostri studenti probabilmente vi hanno detto che su Internet c’è tutto – questa volta hanno ragione. Ci sono molti esempi di database biologici liberamente accessibili su Internet e contenenti dati provenienti dalla vera ricerca; per questa attività utilizzeremo due risorse.
Il Centro Nazionale per l’Informazione Biotecnologica(NCBI)w1 di Bethesda, MD, USA, permette l’accesso ad informazioni biomediche e genomiche, mentre l’Istituto Europeo di Bioinformatica (EBI)w2, con sede a Hinxton, UK, fornisce dati liberamente accessibili provenienti da esperimenti biologici e conduce ricerca di base in biologia computazionale. Il database dell’NCBI vi fornirà le sequenze di ogni gene o proteina che sia stato sequenziato, dopodiché potrete usare gli strumenti messi a disposizione dall’EBI per allineare le sequenze ed esaminarle.
Attività
Quando si ricercano le relazioni evolutive fra organismi diversi, è importante scegliere attentamente il gene o la proteina da usare. Esistono ben noti geni omologhi che possono essere usati, come quelli che codificano per le proteine emoglobina e citocromo C, ed in questa attività useremo quest’ultimo. Il citocromo C é una piccola proteina eme che fa parte della catena di trasporto degli elettroni nei mitocondri. Tutti gli organismi aerobi si sono evoluti da un progenitore comune che utilizzava il citocromo C, quindi questa proteina è una buona scelta per il nostro scopo.w3.
Questa attività si svolge in tre fasi:
- ricerca della sequenza aminoacidica del citocromo C in organismi diversi;
- allineamento delle sequenze, e
- costruzione di un albero filogenetico.
Al termine vengono sottoposte alcune domande per guidare la ricerca delle relazioni evolutive.

ingrandirla
Ricerca delle sequenze proteiche
- Andare alla pagina web dell’NCBIw1.
- Nell’area di ricerca in alto, selezionare “protein” nel menù a tendina.
- Digitare il nome di una specie, ad esempio Homo sapiens, e cytochrome c.
- Cliccare il bottone search.
- Si aprirà una pagina che riporta i risultati della ricerca. Molti di essi sono la stessa sequenza proveniente da fonti diverse, ma altri possono essere sequenze parziali o appartenere a specie diverse o a proteine diverse. Scegliere con attenzione la proteina di interesse e cliccare sul link ‘FASTA’.
- Dalla pagina che si aprirà, copiare la sequenza di lettere maiuscole che si riferisce alla sequenza aminoacidica. Incollare le lettere in un documento Word, ricordando di specificare il nome dell’organismo dal quale proviene la sequenza.
- Ripetere il procedimento per un certo numero di organismi, a seconda di quello che volete ricercare con i vostri studenti. Si possono includere diversi primati per vedere come si è evoluto l’uomo, oppure organismi dei 5 regni per vedere come la vita in generale si è evoluta. In questa attività si utilizzeranno 3 animali, 2 piante, 2 alghe, un fungo ed un protozoo.
Allineamento delle sequenze
- Accedere al sito dell’European Bioinformatics Institute (EBI)w2 e cliccare su “Services”, quindi selezionare “proteins”.
- Cliccare su Clustal Omega. Copiare il testo salvato nel documento Word nel box denominato “STEP 1”.
- In STEP 2 scegliere un formato per il risultato dell’allineamento, ad esempio “Clustal/w numbers” che mostrerà la lunghezza di ciascuna sequenza. Infine, cliccare su “Submit” per completare la fase 3.
- L’allineamento delle sequenze multiple apparirà in una nuova finestra. La prima cosa da fare è cliccare per evidenziare i colori. Questa opzione attribuisce un colore a ciascun gruppo di aminoacidi, in modo da rendere più semplice la loro identificazione.
- Per analizzare l’allineamento tenete presente che : un asterisco (*) significa che le sequenze sono identiche in quella posizione; due punti (:) indicano sostituzioni conservative (stesso gruppo di colore); un punto (.) si riferisce a sostituzioni semi-conservative (forma simile). I colori raggruppano gli aminoacidi in base alle loro caratteristiche: il rosso si riferisce ad aminoacidi piccoli, idrofobici, aromatici; il blu a quelli acidi; il magenta a quelli basici; il verde agli aminoacidi basici con gruppo aminico, amidico, ossidrilico; il grigio a tutti gli altri.
- Cliccando su “Result Summary” si può visualizzare la percentuale di identità conservata tra i diversi organismi dopo l’allineamento. In questa matrice, potete ricavare la percentuale di identità tra due organismi per quanto concerne il citocromo C. Inoltre, se avete Java™ installato sul vostro computer, potete usare Jalview, un programma gratuito per la modifica, la visualizzazione e l’analisi dell’allineamento di sequenze multiple. Con Jalview potrete visualizzare la sequenza consenso del citocromo C e il livello di conservazione dei singoli aminoacidi.
Il software Clustal Omega ha molte altre opzioni che richiedono competenze matematiche più sofisticate. Se volete approfondire l’uso di Clustal Omega, potete leggere l’articolo di Sievers et al. (2011)
ingrandirla
Costruzione di un albero filogenetico
- Nella pagina dei risultati di Clustal Omega, cliccare su “Phylogenetic tree” in basso (dovete avere installato Java™ ).
- Potete ottenere un albero filogenetico oppure un cladogramma. Nel cladogramma, le lunghezze dei rami sono arbitrarie, mentre nell’albero filogenetico le lunghezze dei rami indicano il tempo necessario per l’evoluzione della proteina.
Per la discussione
ingrandirla
- Le molecole omologhe sono un esempio di evoluzione divergente. Come si può spiegare l’evoluzione divergente usando il citocromo C?
- Per gli allineamenti si possono utilizzare le sequenze di nucleotidi (geni) o di aminoacidi (proteine). Perché pensate sia più utile sfruttare le proteine invece del DNA per analizzare le relazioni evolutive?
- Negli alberi filogenetici, un “clado” è costituito da tutti gli organismi che hanno un antenato comune. Fate un esempio utilizzando il vostro cladogramma.
- Quali organismi hanno subito un evento di speciazione più di recente, secondo l’analisi filogenetica del citocromo C? Quanti sono in totale gli eventi di speciazione?
- Perché pensate che alcuni aminoacidi e non altri siano cambiati in seguito a mutazione? Pensate che gli aminoacidi conservati non siano cambiati perché i relativi codoni non hanno subito alcuna mutazione?
- Mettete in evidenza alcuni di questi aminoacidi conservati nel vostro allineamento. Scoprite la loro funzione su Internet.
Glossario
Cladogramma: un diagramma ramificato che mostra le relazioni evolutive tra specie, con lunghezze arbitrarie dei rami.
Sequenza consenso: Un set noto di sequenze conservate, o l’ordine calcolato degli aminoacidi trovati più di frequente in ogni posizione di un allineamento.
Aminoacidi conservati: una sequenza di aminoacidi in un polipeptide che è simile in diversi organismi.
FASTA: formato di testo per rappresentare sequenze nucleotidiche o peptidiche usando un codice a lettera singola. Una sequenza in FASTA inizia con una riga per la descrizione, seguita da righe di dati relativi alla sequenza. La riga di descrizione si distingue dalle altre per il segno maggiore di (>).
Proteina omologa: proteina condivisa tra diverse specie e derivante da un antenato comune.
Albero filogenetico: un diagramma ramificato che mostra le relazioni evolutive tra specie, nel quale la lunghezza dei rami indica la differenza tra due proteine o geni.
Evento di speciazione: il momento in cui da una specie ancestrale divergono due specie diverse.
Ringraziamenti
L’autore desidera ringraziare la sua collega María Isern per la revisione grammaticale dell’articolo.
References
- Bromhan L. Penny D. (2003) The modern molecular clock. Nature Reviews Genetics 5: 216-224
- Kozlowski C. (2010) Bioinformatica con penna e carta: costruire un albero filogenetico. Science in School 17.
- Sievers F. et al. (2011) Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Molecular Systems Biology 7: 539
- Questo articolo è liberamente disponibile sul sito Molecular Systems Biology.
Web References
- w1 – Il US National Center for Biotechnology Information fornisce accesso ad informazioni biomediche e genomiche.
- w2 – L’European Bioinformatics Institute fornisce dati liberamente disponibili derivanti da esperimenti di scienze della vita, svolge ricerca di base di biologia computazionale e offre programmi di aggiornamento, sostenendo i ricercatori nel mondo accademico e dell’industria.
- w3 – John Kimball ha scritto un libro di testo di biologia online intitolato ‘Taxonomy: Classifying Life’ che include un capitolo su ‘Phylogenetic trees’.
Resources
- Il sito ‘Understanding Evolution’ del Museo di Paleontologia dell’Università della California offre ottime informazioni su come si costruiscono e si interpretano gli alberi filogenetici.
- Per saperne di più sull’uso del citocromo C negli alberi filogenetici.
- Il sito Protein Data Bank in Europa appartiene all’ European Bioinformatics Institute e può essere usato per la ricerca e la visualizzazione della struttura 3D del citocromo C.
- Il sito Tree of Life website consente l’esplorazione interattiva e contiene I filmati della serie televisiva della BBC condotta da Sir David Attenborough.
Review
Gli insegnanti di biologia potrebbero usare questo articolo per collegare argomenti di biologia, storia della scienza, biochimica e genetica. Per utilizzare al meglio questo articolo, è importante che gli studenti conoscano i fondamentali della biochimica di DNA e proteine.
L’attività descritta nell’articolo è importante per motivare gli studenti a lavorare in autonomia nella ricerca reale utilizzando database scientifici. Nel laboratorio di classe, gli studenti possono essere guidati a lavorare in piccoli gruppi, confrontando sequenze di proteine, come il citocromo C, o di DNA per comprendere le differenze tra albero filogenetico e cladogramma. La bioinformatica è molto utile nella scuola secondaria per svolgere “content and language integrated learning” a diversi livelli, in un progetto interdisciplinare che coinvolga gli insegnanti di Inglese, Storia e Fisica. Questo articolo, che lega queste diverse materie, potrebbe anche fornire lo spunto per una discussione sul progresso e i limiti di tali ricerche.
Marina Minoli, Esperto di didattica Centro Universitario Agora, Italia






