Supporting materials
Pegada xenética :unha historia forense (Word)
Pegada xenética :unha historia forense (Pdf)
Download
Download this article as a PDF

Traducido por Jorge J. Pérez-Maceira. Nas series de detectives populares de televisión, a pegada xenética é xeralmente usada para identificar criminais. Sara Müller e Heike Göllner-Heibült botan unha mirada detrás das escenas.
A idea de distinguir persoas polas súas características xenéticas non é nova. O descubrimento en 1900 dos tipos sanguíneos ABO por Karl Landsteinerw1 foi o primeiro marcador xenético en ser usado en ciencia forense, complementado máis tarde cos grupos sanguíneos MN (1927) e o factor Rhesus (1937).
Incluso cando analizamos os tres grupos sanguíneos simultaneamente, aínda así, unha de cada dez persoas dá resultados idénticos; isto fai que as transfusións de sangue sexan posibles. Para fins forenses, con todo, é unha desventaxa, os resultados poden dicir que a mostra de sangue non vén do sospeitoso X, pero non poden indicar con algún nivel aceptable de certeza que vén do sospeitoso Y.
Os avances que foron realizados nos 70 e 80, coa análise de diferentes formas de encimas (isoenzimas) en glóbulos vermellos e en soro sanguíneo. A certeza que a mostra realmente viñese do sospeitoso dependía do número de proteínas analizadas (habitualmente catro); denomínase a dita certeza poder de discriminación. O poder de discriminación que estas técnicas combinadas ofreceu era aínda só de 1:10.000, mellor que o 1:10 da análise de grupos sanguíneos, pero aínda non o suficiente bo. Para conseguir un poder mellor, necesitabamos botar unha ollada máis de cerca na nosa composición xenética.
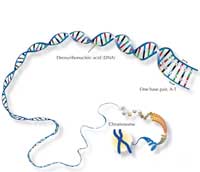
O xenoma humano consiste en 46 pares de cromosomas: 23 da nosa nai, 23 do noso pai. Polo tanto, temos dous de cada cromosoma (excepto – no caso dos homes – do cromosoma sexual) e así dúas copias de cada xene.
O compoñente principal dos cromosomas é o ácido desoxirribonucleico (ADN), o cal contén a información para construír as proteínas necesarias para a vida. Con todo, dos nosos 3 mil millóns de pares de bases (bp) (N.T.: Un billón no orixinal en inglés), só ao redor do 4% codifica para proteínas, o resto é a miúdo simplemente “de recheo” consistente en secuencias repetitivas organizadas en grupos. Si comparas o ADN de dous humanos, a maioría é idéntico, coa variabilidade atopada principalmente nestas secuencias repetitivas.
Diferentes persoas poden ter diferentes números de repeticións destas secuencias: unha persoa pode ter cinco repeticións nun locus de ADN específico (sitio); outra persoa pode ter sete. Utilizando mostras, por exemplo de sangue ou seme, podemos analizar as secuencias repetitivas en varios loci de ADN; esta análise denomínase pegada xenética (genetic fingerprint). Como as pegadas dactilares, as pegadas xenéticas poden usarse para distinguir individuos.
Aínda que o termino “pegada xenética” (ou perfil xenético (genetic profiling)) é comunmente usado, non todo o mundo é consciente de que en realidade abarca dúas técnicas moi diferentes, só una das cales utilízase comunmente na ciencia forense na actualidade.

O primeiro método de pegada xenética foi inventado en 1984 por Alec Jeffreysw2, quen utilizou secuencias de ADN repetido denominadas número variable de repeticións en tándem (variable number tandem repeats ou VNTR; por exemplo, a secuencia D1S80, (AGGACCACCAGGAAGG)n). Estas secuencias, de 10-100 bp por repetición, poden ser investigadas usando encimas de restrición, os cales traballan como tesoiras moleculares para cortar o ADN en secuencias definidas (secuencias de recoñecemento). No noso xenoma completo, unha secuencia de recoñecemento de 6bp terá lugar ao redor de 730.000 veces.
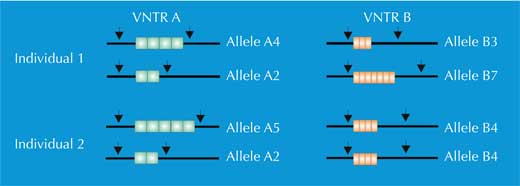
Isto significa que si cortas o xenoma cunha encima de restrición particular, conseguirás ao redor de 730.000 fragmentos de restrición de lonxitudes variables. E isto é onde as VNTR vólvense importantes: o número de repeticións nun grupo de VNTR particular pode varias entre individuos, o que significa que a lonxitude do correspondente fragmento de restrición variará entre individuos tamén (Figura 1). Denominamos a este fenómeno Polimorfismos de lonxitude de fragmentos de restrición (restriction fragment length polymorphism ou RFLP).
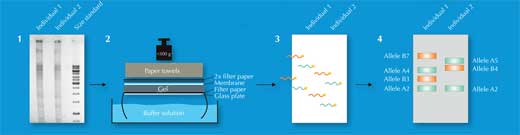
Dos 730.000 fragmentos de restrición, só algúns diferirán entre individuos – demasiado poucos para ser detectados a ollo. En cambio, os científicos utilizan unha técnica denominada Southern blotting, a cal deixa só as secuencias de interese para ser visualizadas. Para facer isto, separan os fragmentos de restrición segundo o seu tamaño con un xel de electroforese, utilizando unha corrente eléctrica para pasar as moléculas cargadas de ADN a través do xel. A distancia que viaxa esta determinada pola medida do fragmento (Figura 2, Paso 1). Logo, transfiren o ADN a unha membrana (Figura 2, Paso 2) e aplican unha sonda etiquetada radioactivamente que é complementaria a o (aos) VNTR(s) de interese. A sonda hibrida (engancha) coa secuencia coincidente (Figura 2, Paso 3) e a membrana colócase sobre unha película de radiografía, os científicos conseguen un imaxe das bandas etiquetas con radiactividade, cada cal representa unha lonxitude diferente de fragmento (Figura 2, Paso 4). Esta imaxe é a pegada xenética.
Así que, cantos VNTR necesitan ser comparados para distinguir de forma fiable entre individuos? Si os científicos escollen VNTRs con suficiente variación (por exemplo, D1S80, o cal pode ser repetido calquera entre 15 a 41 veces), eles só necesitan para comparar catro diferentes VNTR para ter un poder de discriminación de 1:1 millón – moito mellor que o 1:10 ofrecido polo tipo sanguíneo ABO.
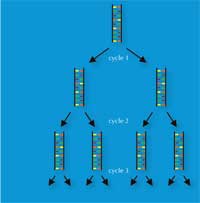
A invención de Kary Mullis, en 1983, da reacción en cadea da polimerasa (Polymerase Chain Reaction ou PCR) fíxolle gañar o premio Nobel en Químicaw3, w4. Esta invención, xunto co descubrimento a finais de 1980 dos polimorfismos de repeticións curtas en tándem (short tandem repeats ou STR) – secuencias repetitivas de 2-9bp, denominadas microsatélites – pavimentou o xeito para unha técnica de pegada xenética de alta velocidade que os científicos forenses utilizan hoxe.
A PCR habilita para que un locus de ADN de interese (por exemplo, o STR de 4bp denominado como D18S51, (AGAA)n), sexa amplificado exponencialmente, xerando mil millóns de copias dunha soa molécula de ADN en poucas horas (Figura 3). Para os científicos forenses, isto ten a vantaxe de facer a análise de ata mostras moi minúsculas – tan só 30 células (ver Táboa 1 para unha comparación coa pegada xenética baseada en RFLP).
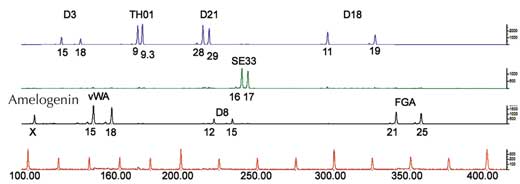
Para análise de PCR, necesitamos STR flanqueados por secuencias que sexan idénticas en todos os seres humanos (dicimos que estas secuencias están conservadas). Entón utilizamos primers – moléculas curtas que son complementarias á secuencia conservada do flanco (xenes 1134 e 1135 na Figura 4) – para iniciar a PCR. Unha vez o ADN foi amplificado, podémolo separar xa sexa por xel de electroforese (Figura 5) ou, nas ciencias forenses modernas, por secuenciación automatizada electroforética (Figura 6), e visualizalo como unha pegada xenética.

Temos dúas copias de cada cromosoma, así que tamén temos dúas copias de cada STR. Si, para cada copia do STR, alguén ten o mesmo número de repeticións (por exemplo, o mesmo alelo), a análise de PCR revela só unha medida do fragmento de ADN: a persoa é homocigota para este alelo STR (frechas verdes na Figura 5, correspondentes ao individuo 2 na Figura 4). Si os dous cromosomas levan alelos non idénticos para este STR, vemos dúas medidas do fragmento e dicimos que a persoa é heterocigota (frechas vermellas na Figura 5, correspondentes ao individuo 1 da Figura 4).
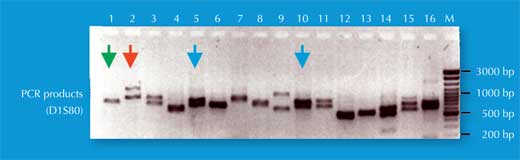
Si só analizamos un STR, a posibilidade de dúas persoas non relacionadas teñan a mesma pegada xenética baseada en PCR é alta – entre 1:2 e 1:100 (frechas azuis na Figura 5). Isto é porque os STR teñen menos alelos e máis baixa heterocigosidad que os VNTR utilizados en pegadas xenéticas baseadas en RFLP. Para superar esta desventaxa, analizamos múltiples STR simultaneamente, con 16 STR, como é común en casos forenses en Alemaña, podemos conseguir un poder de discriminación de 1:10 mil millóns (equivalente a unha persoa na poboación mundial; Figura 6).
| RFLP | PCR | |
|---|---|---|
|
Cantidade de ADN inicial |
30–50 µg |
Polo menos 200 pg (sobre 30 células) para un completo patrón STR |
|
Sensibilidade |
+ |
– |
|
Calidade do ADN requirida para a análise |
Complete genome |
No complete genome necessary; degradation products also sufficient because of the short sequences involved (total sequence length of an STR, including multiple repeats and flanking sequences, approximately 50– 500 bp) |
|
Tempo |
Días a semanas |
Horas |
|
Poder de discriminación por locus |
+++ |
+ |
|
Unidade de repetición |
10 bp a 100 bp |
2 bp a 9 bp (en casos forenses, principalmente 4bp) |
|
Detección automatizada |
Non posible |
Posible o procesamento de mostras de alto rendemento |
|
Número de loci validados (importante si os parentes están implicados) |
Número limitado |
Número grande |
|
Risco de contaminación |
+ |
+++ |
|
Requírense medidas de seguridade adicionais? |
Si (debido ás sondas radioactivas) |
Non (ningunha sonda radiactiva) |

Agora sabemos que é unha pegada xenética, pero, Cómo se utiliza? A pegada xenética baseada en PCR é amplamente aplicada nas investigacións forenses: permite á policía excluír ou identificar sospeitosos en base a material xenético como un folículo capilar, pel, seme, saliva ou sangue (véxase a historia que pode ser descargada a continuaciónw5). Unha pegada xenética soa, con todo, non é suficiente evidencia para unha condea, dado que parentes próximos poden ter pegadas moi similares (e os xemelgos homocigóticos normalmente téñenos idénticos).

E para complicar as investigacións forenses internacionais, malia que hai unha recomendación europea para analizar 16 STR, cada país pode decidir cales STR analizar, o cal fai as comparacións difíciles.
O método baseado en PCR é tamén utilizado en humanos para os test de paternidade, diagnóstico de moitas enfermidades xenéticas (por exemplo, a enfermidade de Huntingdon), identificación de vítimas de desastres, seguimento de árbores familiares, localización de persoas desaparecidas e investigación de figuras históricas (por exemplo, o último Zar de Rusia e a súa familia). Noutros organismos, pode usarse con propósitos de conservación (por exemplo, para analizar o marfil confiscado), en investigacións de fármacos (por exemplo, para analizar plantas de cannabis incautadas), para control alimentario ou de calidade de auga (por exemplo, para identificación de microbios contaminantes), en medicina (por exemplo, para detectar infeccións virais como VIH, hepatite ou gripe) e en investigacións de bioterrorismo (por exemplo, para identificar cepas microbianas).
A pegada xenética baseada en RFLP, aínda que en gran parte obsoleta debido ás moitas vantaxes do método PCR (Táboa 1), é aínda usada para clasificación de plantas e animais en investigación básica. É particularmente útil cando hai información insuficiente sobre o xenoma da especie – recordar que para o método de PCR, necesitamos rexións que varían amplamente entre individuos e estean flanqueadas por rexións conservadas de secuencia coñecida.
O illamento do ADN na escola dá aos estudantes un momento “guau” cando se dan conta que están buscando na información xenética completa que codifica para un organismo – unhas hebras de ADN como fíos de algodón que precipitan por alcohol. É fácil de realizar na escola utilizando salivaw6 (ou células epiteliais de kits comerciais dispoñibles), chícharos (Madden, 2006), tomates, cebolas (Madden, 2006), tomatoes, onionsw7 ou estafo de tenreira (antes consultar as restricións locais de usar estafo de tenreira na escola)w8.

A posterior PCR dun STR específico en ADN humano, por exemplo D1S80 ou TH01, pode ser realizada na escolaw6, usando kits comerciais dispoñibles de razoable prezow9 si é necesario. Si a vosa escola non ten acceso a un termociclador, o termociclado pode ser levado a cabo en tres baños de auga, malia que é tedioso e moi práctico.
Si este equipamento non está dispoñible, hai kits que simulan e simplifican o proceso enteiro de pegada xenéticaw10. Estes kits conteñen fragmentos de ADN que simulan a amplificación de diferentes alelos dun só STR ou VNTR (De feito, son fragmentos de restrición de ADN do ADN dun plásmido ou dunha fago lambda.) O ADN require electroforese e posterior revelado de modo que os estudantes son capaces de comparar as secuencias “amplificadas” de ADN dunha mostra de evidencia cos de varios sospeitosos. Naturalmente, isto é moi diferente de detectar amplificando STR usando a secuenciación automatizada electroforética (e nin sequera representan con exactitude a visualización de VNTR utilizando Southern blotting, como o ADN tínguese directamente sobre o xel), pero con todo demostra os principios do proceso analítico. Cando se utilizan estes kit de simulación, os estudantes deben ser conscientes de que os experimentos dan a impresión de que as diferenzas entre os individuos poden ser identificadas facilmente, o cal non é o caso.
As autoras gustaríalles agradecer a Wolfgang Nellen as súas ideas sobre o artigo e por deixar as instrucións de Science Bridge dispoñibles gratis.
Ademais, agradecer a Shelley Goodman polo seu asesoramento no uso de kits comerciais na escola.
Nunha entrevista con Science in School, Alec Jeffreys fala do seu descubrimento:
Hodge R, Wegener, A-L (2006) Alec Jeffreys interview: a pioneer on the frontier of human diversity. Science in School 3: 16-19. www.scienceinschool.org/2006/issue3/jeffreys
A afiliación a Science Bridge é necesaria para acceder a estas instrucións, pero os lectores deste artigo pódenas pedir gratis a sara.mueller@sciencebridge.net
Para descubrir como illar ADN a partir de tomates (instrucións en Inglés), véxase: http://ucbiotech.org/edu/edu_aids/TomatoDNA.html
Klug WS, et al. (2008) Concepts of Genetics 9th edition. San Francisco, CA, USA: Pearson. ISBN: 9780321524041
Goodwin W, Linacre A, Hadi S (2010) An Introduction to Forensic Genetics. Chichester, UK: Wiley-Blackwell. ISBN: 978-0470710197
Wallace-Müller K (2011) The DNA detective game. Science in School 19: 30-35. www.scienceinschool.org/2011/issue19/detective
Patterson L (2009) Getting a grip on genetic diseases. Science in School 13: 53-58. www.scienceinschool.org/2009/issue13/insight
A idea de utilizar o ADN para identificar a un individuo no mundo é alucinante. A pegada xenética dun individuo baséase nas secuencias que non son usadas na codificación, pero cómo son estas pegadas creadas? Neste artigo explícase.
A habilidade para identificar aos criminais a partir das bases de datos de ADN suscita preguntas importantes: É ético manter o ADN humano nas devanditas bases de datos ou se trata dunha violación dos dereitos humanos? Debería haber unha pegada de ADN de cada persoa, ou simplemente aqueles que estean detidos? Canto tempo debe o perfil de ADN manterse?
Os estudantes talvez desexen examinar como as pegadas xenéticas poden axudar a diagnosticar enfermidades xenéticas, así como a súa aplicación na loita contra a caza furtiva e a extinción das especies. Idealmente deben ser capaces de tratar a técnica por si mesmos, xa sexa como un experimento real ou simulado.
Shelley Goodman, Reino Unido
Pegada xenética :unha historia forense (Word)
Pegada xenética :unha historia forense (Pdf)
Download this article as a PDF