Supporting materials
Les empreintes génétiques : une histoire de médecine légale (Word)
Les empreintes génétiques : une histoire de médecine légale (Pdf)
Download
Download this article as a PDF

Traduit par Nathalie Bisson. Dans les séries télévisées populaires, les empreintes génétiques sont couramment utilisées pour identifier les criminels. Sara Müller et Heike Göllner-Heibült ont regardé derrière le rideau.
L’idée de distinguer les individus en utilisant leurs caractéristiques génétiques n’est pas nouvelle. Découvert en 1900 par Karl Landsteinerw1, les groupages sanguins ABO ont été les premiers marqueurs génétiques utilisés en médecine légale, complétés plus tard par les typages sanguins MN (1927) et la détermination des facteurs Rhésus (1937).
Cependant, même si nous recoupons l’ensemble de ces trois types de marqueurs sanguins, environ une personne sur dix donne les mêmes résultats ; c’est ce qui rend les transfusions sanguines possibles. En médecine légale, au contraire, ceci représente un désavantage : les résultats peuvent dire si l’échantillon de sang ne provient pas du suspect X, mais ils ne peuvent pas dire avec suffisamment de certitude qu’il vient du suspect Y.
Des progrès ont été réalisés dans les années 1970 et 80, avec l’analyse de différentes formes d’enzymes (isoenzymes) présentes dans les globules rouges et le sérum. La fiabilité des résultats dépendait du nombre d’enzymes analysées (habituellement quatre) ; Cette fiabilité est appelée pouvoir de discrimination. Ce pouvoir de discrimination n’était encore que de 1/1000, bien meilleur que celui de 1/10 basé sur les typages sanguins, mais encore insuffisant. Pour l’améliorer, il était nécessaire d’aller voir encore plus précisément dans nos caractéristiques génétiques.
Le génome humain consiste en 46 chromosomes : 23 de notre mère et 23 de notre père. Nous avons donc deux exemplaires de chaque chromosome (excepté- dans le cas des hommes- les chromosomes sexuels) et donc deux copies de chaque gène.
Le principal composant des chromosomes est l’acide désoxyribonucléique (ADN), qui renferme l’information permettant de fabriquer les protéines nécessaires à notre vie. Cependant, sur nos 3 millions de paires de bases (pb), seulement 4% code effectivement pour des protéines ; Le reste représente souvent seulement du « remplissage », et contient des groupes de séquences répétitives. Si l’on compare l’ADN de deux humains, la plus grande partie est identique, et la variabilité est surtout trouvée au niveau de ces séquences répétitives.
Des personnes différentes peuvent avoir un nombre différent de répétitions de ces séquences : une personne peut avoir cinq répétitions à un locus spécifique; une autre personne pourra en avoir sept. En utilisant des échantillons, souvent des cellules sanguines ou du sperme, nous pouvons analyser les séquences répétées à différents loci ; Ceci s’appelle faire une empreinte génétique. Comme les empreintes digitales, les empreintes génétiques peuvent être utilisées pour distinguer les individus.
Bien que le terme empreinte génétique (ou profil génétique) soit utilisé couramment, peu de monde comprend vraiment qu’il recouvre deux techniques très différentes utilisées toutes deux en médecine légale aujourd’hui.

La première méthode pour réaliser des empreintes génétiques a été inventée en 1984 par Alec Jeffreysw2, qui a utilisé des séquences d’ADN répétées appelées « nombre variable de répétitions en tandem » (VNTR; c’est à dire séquence D1S80, (AGGACCACCAGGAAGG)n). Ces séquences de 10 à 100 pb par répétition, peuvent être étudiées en utilisant des enzymes de restriction, qui sont des ciseaux moléculaires coupant l’ADN en des sites spécifiques (sites de restriction). Sur l’ensemble de notre génome, une séquence représentant un site de restriction de 6 pb peut apparaitre environ 730 000 fois.
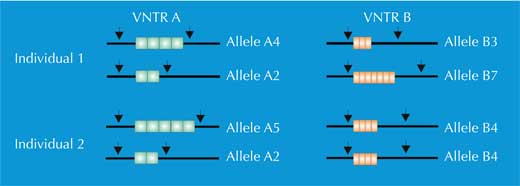
Cela signifie que si l’on coupe le génome avec une enzyme de restriction particulière, on peut obtenir environ 730 000 fragments de restriction de différentes longueurs. C’est à ce point que les VNTR deviennent importantes : le nombre de répétitions dans un groupe VNTR peut varier d’un individu à l’autre, ce qui signifie que la longueur du fragment de restriction correspondant va varier d’un individu à l’autre (figure 1). Ce phénomène est appelé polymorphisme de longueur des fragments de restriction (RFLP).
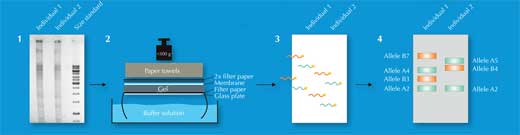
Sur les 730 000 fragments de restriction, seuls quelques-uns diffèrent entre deux individus- trop peu pour être repérés à l’œil. A la place, les scientifiques utilisent une technique appelée Southern blotting, qui permet de repérer seulement les séquences d’intérêt. Pour faire ceci, on sépare les fragments de restriction sur le critère de leur taille dans un gel d’électrophorèse, en utilisant un courant électrique qui permet aux fragments d’ADN, qui sont des molécules chargées, de se déplacer dans le gel. La distance parcourue est déterminée par la taille du fragment (figure 2, étape 1). Ensuite, ils transfèrent l’ADN sur une membrane (figure 2, étape 2) et l’incubent avec une sonde radioactive complémentaire des VNTR d’intérêt. La sonde s’hybride (se colle) aux séquences correspondantes (figure 2, étape 3), et en plaçant cette membrane sur un film radiographique, les scientifiques ont une image des bandes radioactives, qui pour chacune représentent une longueur différente de fragment (figure 2, étape 4). Cette image est l’empreinte génétique.
De combien de VNTR avons-nous besoin pour distinguer les individus entre eux? Si les scientifiques choisissent les VNTR qui possèdent beaucoup de variations (comme D1S80, qui peut être répété de 15 à 41 fois), ils ont besoin de comparer uniquement 4 VNTR pour avoir un pouvoir de discrimination de 1 / 1 million- ce qui est bien mieux que 1/10, offert par le typage des groupes sanguins ABO.
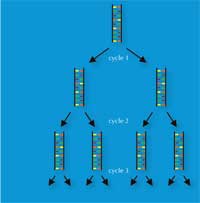
L’invention, en 1983, par Kary Mullis, de la réaction de polymérisation en chaine (PCR) lui a valu le prix Nobel de Chimiew3, w4. Cette invention, accompagnée à la fin des années 80 par la découverte des répétitions courtes en tandem (STRs)- séquences de 2 à 9 paires de bases (pb) répétées, aussi appelées microsatellites- ont ouvert la voie à une technique rapide de réalisation des empreintes génétiques, qui est utilisée aujourd’hui par les scientifiques de médecine légale.
La PCR permet d’amplifier de façon exponentielle un locus d’ADN d’intérêt (par exemple le STR de 4 pb connu sous le nom de D18S51, (AGAA)n), générant ainsi un million de copies d’un ADN simple brin en quelques heures. Pour la police scientifique, ceci a l’avantage de rendre possible l’analyse de très petits échantillons- contenant aussi peu que 30 cellules (voir le tableau 1 pour une comparaison avec les empreintes génétiques basées sur les RFLP).
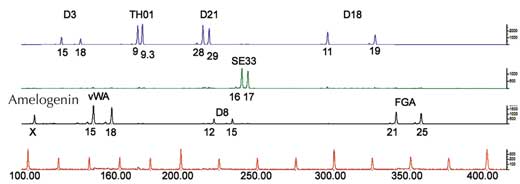
Pour une analyse par PCR, nous avons besoin de STRs encadrés par des séquences identiques chez tous les êtres humains (nous disons que ces séquences sont conservées). Ensuite, nous utilisons des amorces – courtes molécules complémentaires des séquences encadrantes conservées (gènes 1134 et 1135 sur la figure 4) – pour initier la PCR. Une fois que l’ADN a été amplifié, on peut le séparer soit par électrophorèse sur gel (Figure 5) ou, dans les laboratoires de police scientifique modernes, par la méthode de séquençage électrophorétique automatique (Figure 6), et visualiser ainsi les empreintes génétiques.

Nous avons deux copies de chaque chromosome, donc nous avons aussi deux copies de chaque STR. Si, pour chaque copie du STR, quelqu’un a le même nombre de répétitions (c’est-à-dire le même allèle), l’analyse par PCR révèlera seulement une taille de fragments d’ADN : la personne est homozygote pour l’allèle du STR (flèche verte dans la figure 5, correspondant à l’individu 2 de la figure 4). Si les deux chromosomes portent des allèles différents pour ce STR, nous verrons deux tailles de fragment et dirons que cette personne est hétérozygote (flèche rouge dans la figure 5, correspondant à l’individu 1 de la figure 4).
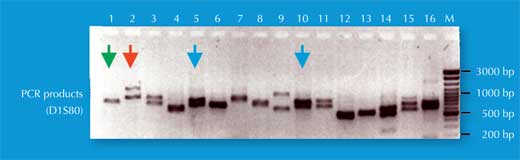
Si nous analysons un seul STR, les chances pour que deux personnes non apparentées aient la même empreinte génétique par PCR est élevée, entre 1:2 et 1:100 (flèches bleues dans la Figure 5). Ceci est lié au fait que les STRs ont moins d’allèles et moins de variabilité que les VNTRs utilisés dans les empreintes génétiques basées sur les RFLP. Pour s’affranchir de ce problème, nous devons analyser de nombreux STRs en parallèle ; Avec 16 STRs, comme pratiqué en routine dans les laboratoires de police scientifique en Allemagne, nous atteignons un pouvoir de discrimination de 1 /10 milliard (soit une personne dans la population mondiale ; figure 6).
| RFLP | PCR | |
|---|---|---|
|
Quantité d’ADN de départ |
30–50 µg |
Au moins 200 pg (environ 30 cellules) pour une empreinte complète par STR |
|
Sensibilité |
+ |
+++ |
|
Qualité de l’ADN requise pour l’analyse |
Génome complet |
Pas de génome complet nécessaire ; produits de dégradation suffisants aussi du fait des courtes séquences impliquées (la longueur totale d’un STR, incluant les répétitions multiples et les séquences encadrantes, est de 50 à 500 pb) |
|
Temps |
Jours à semaines |
Heures |
|
Pouvoir de discrimination par locus |
+++ |
+ |
|
Unité répétée |
10 bp à 100 bp |
2 bp à 9 bp (dans la plupart des cas traités par la médecine légale, 4 bp) |
|
Détection automatique |
Impossible |
Prise en charge d’un échantillon de grande capacité possible |
|
Nombre de locus valides (important si des individus parents sont impliqués) |
Nombre limité |
Nombre important |
|
Risque de contamination |
+ |
+++ |
|
D’autres mesures de sécurité sont-elles requises? |
Oui (à cause des sondes radioactives) |
Non (pas de sondes radioactives) |


Nous savons maintenant ce que sont les empreintes génétiques, mais comment sont elles utilisées? La méthode par PCR est applicable dans la plupart des cas de médecine légale : elle permet à la police d’exclure ou d’identifier des suspects sur la base de matériel génétique provenant de bulbes pileux, de peau, de sperme, de salive ou de sang (voir l’histoire à télécharger plus loinw5). Une empreinte génétique seule, cependant, n’est pas une preuve suffisante pour une condamnation, car des parents proches peuvent avoir des empreintes génétiques très similaires (et la même empreinte s’il s’agit de jumeaux monozygotes).
Et pour compliquer les enquêtes de police scientifique internationales, bien qu’il y ait une recommandation européenne préconisant d’analyser 16 STRs, chaque pays peut décider quels locus STR analyser, ce qui rend les comparaisons difficiles.
La technique par PCR est aussi utilisée, chez l’Homme, pour des tests de paternité, diagnostiquer de nombreuses maladies génétiques (par exemple la maladie de Huntington), identifier les victimes d’une catastrophe, réaliser des arbres généalogiques, rechercher des personnes disparues et faire des recherches sur des personnages historiques (par exemple le dernier tsar de Russie et sa famille). Chez d’autres organismes, elle peut être utilisée dans un objectif de conservation des espèces (par exemple pour analyser de l’ivoire confisqué), dans la recherche de drogues (par exemple pour analyser une saisie de plantes de cannabis), pour contrôler la qualité de l’eau ou de la nourriture (par exemple pour identifier des microbes contaminants), en médecine (par exemple pour détecter des infections par le HIV, l’hépatite ou le virus de la grippe), and dans les enquêtes concernant le bioterrorisme (pour identifier des souches de microbes).
Les empreintes génétiques par la méthode des RFLP, bien qu’obsolète compte tenu des nombreux avantages de la PCR (tableau 1), est encore utilise pour classer les plantes et les animaux en recherché fondamentale. Elle est particulièrement utile quand l’information sur les génomes des espèces est insuffisante- rappelez-vous que pour une analyse par PCR, nous avons besoin de régions qui varient beaucoup en fonction des individus et qui sont flanquées de régions conservées bien connues.
L’extraction d’AND à l’école procure un grand moment aux étudiants lorsqu’ils réalisent qu’ils ont accès à l’ensemble de l’information génétique qui code pour un organisme- quelques filaments cotonneux d’ADN précipités par l’alcool. Elle est facile à réaliser à l’école en utilisant de la salivew6 (ou des cellules épithéliales de kits commerciaux), des pois (Madden, 2006), des tomates, des oignonsw7 ou du thymus de veau (attention aux réglementations locales concernant son usage à l’école)w8.

Par la suite, l’analyse PCR d’un STR spécifique de l’ADN humain, par exemple D1S80 ou TH01, peut être réalisée à l’écolew6, en utilisant des kits commercialisésw9 de prix raisonnable si nécessaire. Si votre école n’a pas accès à un appareil à PCR, les cycles de températures peuvent être réalisés dans trois bains Marie, bien que cela soit fastidieux et demande beaucoup de manipulations.
Si ces équipements ne sont pas accessibles, il y a des kits qui en même temps simulent et simplifient le processus complet de réalisation d’empreintes génétiquesw10. Ces kits contiennent des fragments d’ADN qui simulent l’amplification de différents allèles d’un seul STR ou VNTR. (en fait, ce sont des fragments de restriction de plasmides ou de l’ADN de phage lambda). Il faut réaliser l’électrophorèse et la coloration des fragments d’ADN pour que les étudiants puissent comparer les séquences d’ADN « amplifiées » d’un échantillon présenté comme un indice, avec ceux de différents suspects. Bien sûr, cela est très différent de la détection des STRs amplifiés par la technique d’électrophorèse automatisée (and ne représente même pas réellement la visualisation des VNTRs après Southern blot, puisque l’ADN est visualisé directement dans le gel), mais cette manipulation démontre néanmoins le principe de cette analyse. Lorsqu’ils utiliseront ces kits, les étudiants devront être avertis que les différences entre les individus semblent être faciles à établir, ce qui n’est pas en réalité le cas.
Les auteurs voudraient remercier Wolfgang Nellen pour ses idées sur cet article et pour avoir donné accès gratuitement à Science Bridge.
Ils sont aussi reconnaissants à Shelley Goodman pour ses conseils concernant l’utilisation de kits commerciaux à l’école.
Dans une interview avec Science in School, Alec Jeffreys discute de ses découvertes:
Hodge R, Wegener, A-L (2006) Alec Jeffreys interview: a pioneer on the frontier of human diversity. Science in School 3: 16-19. www.scienceinschool.org/2006/issue3/jeffreys
Il faut normalement être membre de Science Bridge pour avoir accès à ces instructions, mais les lecteurs de cet article peuvent les réclamer gratuitement à sara.mueller@sciencebridge.net
Pour trouver comment isoler de l’AND de tomates (en anglais), voir: http://ucbiotech.org/edu/edu_aids/TomatoDNA.html
Klug WS, et al. (2008) Concepts of Genetics 9th edition. San Francisco, CA, USA: Pearson. ISBN: 9780321524041
Goodwin W, Linacre A, Hadi S (2010) An Introduction to Forensic Genetics. Chichester, UK: Wiley-Blackwell. ISBN: 978-0470710197
Wallace-Müller K (2011) Le jeu de détective génétique. Science in School 19. www.scienceinschool.org/2011/issue19/detective/french
Patterson L (2009) Maîtriser les maladies génétiques. Science in School 13. www.scienceinschool.org/2009/issue13/insight/french
L’idée d’utiliser l’ADN pour identifier un individu particulier sur Terre est très excitante. L’analyse d’empreintes génétiques des individus repose sur l’étude de séquences non codantes, mais comment sont-elles créées ? c’est ce que cet article explique. La possibilité d’identifier des individus sur la base de banques de données ADN soulève des questions importantes : est-il éthiquement acceptable de stocker de l’ADN humain dans de telles banques de données ou est-ce une atteinte aux droits de l’Homme? Devraient-elles renfermer une empreinte d’ADN de chaque personne ou seulement de ceux qui ont été arrêtés ? Combien de temps devrions-nous garder ces profils génétiques ?
Les étudiants pourraient aussi être intéressés à savoir comment les empreintes génétiques permettent de diagnostiquer des maladies génétiques, ainsi que leurs applications dans la lutte contre le braconnage ou l’extinction des espèces. Dans l’idéal, ils devraient pouvoir essayer la technique par eux-mêmes, dans une expérience réelle ou une simulation.
Shelley Goodman, Grande-Bretagne