Desvelando nuestro código genético Understand article
Traducido por Mónica González. ¿Qué función cumple el ADN basura? La comunidad científica ha investigado esta fracción del material genético, que podría ser clave en algunas enfermedades graves.

iStockphoto
El Proyecto Genoma Humano – secuenciación del genoma humano – desveló nuestro mapa genético (tres billones de bases). Pero la historia no termina ahí: descifrar cómo las células interpretan esta secuencia es esencial para entender el funcionamiento del genoma. Ese conocimiento se aplicaría, por ejemplo, a la investigación biomédica y la asistencia clínica.

automático de ADN empleado
en el Proyecto de Genoma
Humano. Cada pico en color
corresponde a una base
individual. El Proyecto de
Genoma Humano identificó 3
billones de bases. ENCODE
investiga el funcionamiento
del genoma. Haga clic sobre
la imagen para ampliarla.
Imagen cortesía de
Investigación del Genoma
Limited
El Proyecto concluyó que sólo el 2% del genoma codificaba para proteínas; el resto correspondía a ARN no codificantes, elementos reguladores e intrones. Parecía que una gran parte del genoma era inservible (ADN basura).
Más allá de la secuencia
Tras la secuenciación era momento de confirmar la existencia de ADN “basura”. En 2003 el consorcio ENCODE se constituye para caracterizar elementos no codificantes pero funcionales del genoma humano. El consorcio fue apoyado por el Instituto Nacional de Investigación del Genoma Humano en los EE.UU. y dirigido por el Instituto Europeo de Bioinformática (EBI; ver recuadro) de Reino Unido. La fase piloto del proyecto funcionó entre 2003 y 2007 e implicó una red científica internacional para optimizar y contrastar métodos experimentales y computacionales en el estudio de la fracción genómica considerada: 1%.
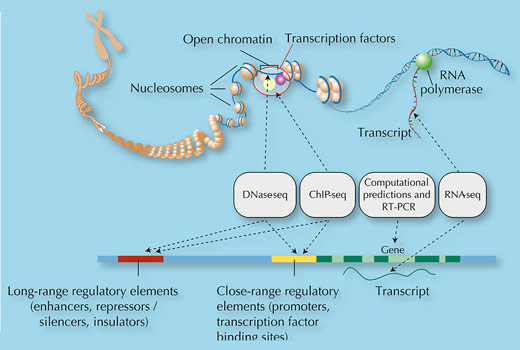
Imagen cortesía de Ian Dunham
analizó 147 tipos celulares
para comprender
especificidades de regulación
génica en distintos tejidos.
Este diagrama muestra 47 de
los 147 tipos. Las células
comparten el mismo genoma
pero el patrón de expresión
puede variar. Haga clic sobre
la imagen para ampliarla.
Imagen cortesía de Darryl Leja
Los resultados iniciales, publicados en junio de 2007 (Consorcio ENCODE Project, 2007), ofrecieron una primera visión de la actividad genómica. Datos combinados de microarrays (ver Koutsos et al., 2009) y experimentos de secuenciación mostraron que la mayoría del genoma se transcribe, incluidas regiones antes reconocidas como “ silenciosas” (figura 2). La función biológica de la mayoría de los transcritos era desconocida; algunos parecían implicados en la regulación génica. La interacción los distintos elementos del genoma resultaba más compleja de lo previsto: mayor diversidad de secuencias activas y menor proporción de genoma inutilizado.
Tras el éxito de la fase piloto el consorcio ENCODE amplió el alcance de su estudio: todo el genoma. Este progreso fue facilitado por innovación tecnológica en la metodología requerida.
El análisis avanzado permitió “cartografiar” las características del genoma , situando zonas de metilación (regiones tipo “shhhh) -indican silenciamiento de genes- u otras modificaciones que inciden en el empaquetamiento; también, “sitios de unión” para factores transcripcionales o enhancers (figura 3).
Diluvio de datos

imprimiesen el papel
llenarían 12 autobuses.
Imagen cortesía de
marcus_jb1973 / Flickr
En Septiembre de 2012, después de 5 años de experimentos y análisis con 442 investigadores de 32 institutos de investigación en el Reino Unido, EE.UU., España, Singapur y Japón, el proyecto ENCODE hizo públicos sus resultados. Se utilizaron cerca de 300 años de tiempo de ordenador para analizar 15 terabytes de datos (15 x 1012 bytes); toda la información es de acceso público. Si los datos se imprimiesen el papel llenaría 12 autobuses.
El proyecto ENCODE es un ejemplo de hito colectivo por contribución organizada de grupos de investigación en distintos países.
Dar vida a la secuencia
Fascinante: contra el pronóstico inicial el 80% de nuestro genoma es activo ¿Qué procesos ocurren? Cuestión por dilucidar, aunque un 9% del mismo –tal vez, mucho más- está implicado en la regulación de la expresión génica. El 80% del genoma activo contiene más de 70 000 regiones promotoras y casi 40 000 regiones potenciadoras.
Una visión global

de un ingeniero de sonido la
expresión génica se halla
bajo el control de más de 4
millones de interruptores
génicos.
Imagen cortesía de Stuart
Dallas Photography / Flickr
En conjunto, ENCODE identificó más de 4 millones de interruptores génicos dispersos por todo el genoma. Podemos imaginar el genoma como una mesa de mezclas de sonido, donde gran cantidad de interruptores activan y desactivan genes ¿Qué significa esto? Un ejemplo: un pequeño cambio en el interruptor génico CARD9 implica un incremento del 20% en el riesgo de desarrollar el síndrome de Crohn, enfermedad inflamatoria del intestino ¿Podríamos intervenir/modificar los interruptores para controlar la aparición de enfermedades? Tal vez, ahora podemos apreciar mejor el beneficio potencial de estas investigaciones que orientan hacia innovaciones biomédicas.
Los resultados ENCODE informan sobre la organización del genoma y las interacciones físicas que suceden en el mismo. En efecto: los interruptores génicos quedan próximos a los genes que regulan a pesar de la distancia lineal física de cientos de kilobases ¿Cómo se explica? El genoma no es una entidad lineal: se encuentra empaquetado permitiendo el acercamiento de zonas “linealmente” separadas.
Generando datos

permitirán una mejor
comprensión de la base
genética de la enfermedad.
Imagen cortesía de AlexRaths
/ iStockphoto
ENCODE ofrece un mapa detallado del genoma y sugiere nuevas líneas de investigación. Como Ian Dunham de EBI y autor principal del artículo ENCODE explica “En muchos casos, sabemos qué genes están implicados en una enfermedad pero ignoramos los interruptores. Por otro lado, la ubicación de los interruptores no parece relacionada con los genes “su” enfermedad” ENCODE señala pistas a seguir para dilucidar los mecanismos de patología molecular. A partir de ahí podrán plantearse nuevas estrategias terapéuticas.
Concluyendo, se conocían genes relacionados con enfermedades, ahora se confirma la existencia de interruptores implicados en su regulación (activación y desactivan). ENCODE ha sentado las bases para investigaciones de gran valor científico y social.
Más acerca de EBI
El Laboratorio Europeo de Biología Molecular (EMBL)w1 es una de las instituciones de investigación más importantes del mundo, dedicada a la investigación básica en las ciencias de la vida. EMBL es internacional, innovadora e interdisciplinar. Sus empleados procedentes de 60 naciones tienen formación en biología, física, química, informática y colaboran en proyectos de biología molecular.
EBIw2, con sede cerca de Cambridge, Reino Unido, forma parte del EMBL. Proporciona datos de experimentos de Biología e a la comunidad científica mundial y realiza investigación básica en biología computacional. EBI está comprometido con la formación de investigadores académicos y la industria para aprovechar al máximo los datos bioinformáticos.
EMBL es miembro de EIROforumw3, editor de Science in School.
References
- The ENCODE Project Consortium (2007) Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project. Nature 447: 799-816. doi: 10.1038/nature05874
-
Descarge el artículo gratis aquí, o suscríbase a Nature.
-
- The ENCODE Project Consortium (2012) An integrated encyclopedia of elements in the human genome. Nature 489: 57–74. doi: 10.1038/nature11247
-
Descarge el artículo gratis aquí, o suscríbase a Nature.
-
- Koutsos A, Manaia A, Willingale-Theune J (2009) Fishing for genes: DNA microarrays in the classroom. Science in School 12: 44-49.
Web References
- w1 – Aprenda más sobre EMBL.
- w2 – Aprenda más acerca de EBI.
- w3 – EIROforum es una organización internacional que reúne a ocho de las entidades de investigación científica más grande de Europa, combinando recursos, instalaciones y experiencia para potenciar la ciencia europa. Como parte de sus actividades de educación y divulgación, EIROforum publica Science in School.
Resources
- Nature website, the ENCODE Explorer te permite acceder a los datos de ENCODE y a un poster (20 MB) con un conjunto de datos.
- En un video online el editor de Nature Magdalena Skipper y Ewan Birney de EBI’s hablan del proyecto ENCODE.
- The Story of You: ENCODE and the Human Genome presentan ENCODE enformato comic.
- Ewan Birney de EBI, Tim Hubbard del Wellcome Trust Sanger Institute y Roderic Guigo del CRG presentan ENCODE en un video (subtitlado en español).
- Para introducir Bioinformática en el aula ¿por qué no prueba con estas actividades?
-
Kozlowski C (2010) La bioinformática con papel y lápiz: construcción de un árbol filogenético. Science in School 17.
-
Communication and Public Engagement team (2010) ¿Puedes descubrir una mutación cancerígena? Science in School 16.
-
- To learn more about bioinformatics, see:
-
Hayes E (2011) Svante Pääbo: un arqueólogo del genoma. Science in School 20.
-
Pathmanathan S, Hayes E (2007) Nicky Mulder, bioinformatician. Science in School 6: 75-77.
-
Institutions
Review
Este artículo explica uno de los últimos avances en genética humana: proyecto ENCODE y su proceso.
Cuando los estudiantes toman contacto con el código genético se sorprenden porque sólo el 2% de ADN humano codifica para proteínas; el resto podría ser “basura”. El proyecto ENCODE investigó la función de ADN no codificante: en realidad, después de todo, no es basura.
En la discusión del Proyecto del Genoma Humano con los estudiantes también se puede introducir ENCODE. Dar a los estudiantes información básica sobre regulación genética, enfermedades genéticas y sus tratamientos puede ser orientadora. El artículo puede suscitar un interés por la bioinformática; a continuación estaría indicada la revisión bibliográfica sobre el proyecto.
Namrata Garware, India





