De calzadas romanas, vagones de tren e inspectores: descubrimientos recientes en la investigación sobre el RNA Understand article
Traducido por I. Carrero, Universidad de Valladolid. El RNA es una biomolécula crucial de la que raramente se habla en detalle en los libros de texto. En el primer artículo de una serie, Russ Hodge describe recientes y excitantes investigaciones sobre el RNA.

Florian Raible buscando las
especies de evolución más
rápida y más lenta
Imagen cortesía de EMBL
Photolab
Imagina un romano en la antigüedad, de pie sobre una cumbre, observando el paisaje y planeando el trazado de una nueva calzada. Dos mil años más tarde todavía se ven sus carreteras cruzando las ciudades y los paisajes europeos, tan rectas como si hubieran sido trazadas con regla, desviándose únicamente ante los obstáculos más difíciles. En los años 50 y 60 del siglo XX, los científicos trazaron una especie de ruta biológica similar para describir las conexiones entre los diferentes tipos de moléculas en las células.
Imagina un romano en la antigüedad, de pie sobre una cumbre, observando el paisaje y planeando el trazado de una nueva calzada. Dos mil años más tarde todavía se ven sus carreteras cruzando las ciudades y los paisajes europeos, tan rectas como si hubieran sido trazadas con regla, desviándose únicamente ante los obstáculos más difíciles. En los años 50 y 60 del siglo XX, los científicos trazaron una especie de ruta biológica similar para describir las conexiones entre los diferentes tipos de moléculas en las células.
Su plano se conoce como el dogma central de la Biología molecular: “el DNA hace RNA que hace proteínas”, como garabateó James Watson en uno de sus cuadernos cuando él y Francis Crick empezaron su abordaje de la estructura del DNA. Con este dogma, los dos investigadores proponían una nueva relación entre las principales moléculas de la célula –un flujo de sentido único desde los ácidos nucleicos a las proteínas–. Inmediatamente tras su descubrimiento de la doble hélice, Crick ya daba conferencias en las que presentaba el dogma como un importante campo de investigación para la Biología molecular (Crick, 1970): los científicos deberían dedicarse a descubrir los mecanismos celulares exactos que subyacen bajo sus procesos.
Hacia los años 70, se empezaron a comprender las bases de cómo se forman los RNA y las proteínas, pero también se hizo obvio que una carretera de sentido único no podía describir la química de la célula. Al igual que muchas de las actuales rutas son bifurcaciones de las antiguas vías romanas, se han encontrado muchas desviaciones en la ruta entre genes y proteínas. Al principio estas parecían ser raras excepciones al dogma; con el tiempo se han reconocido como comunes y cruciales en la mayoría de los procesos biológicos de los organismos complejos. Algunos de los hallazgos son tan nuevos que aún no aparecen en los libros de texto y son poco familiares para los profesores. Este artículo es el primero de una serie para Science in School que tratará algunas de estas recientes investigaciones.
Algunos de los descubrimientos más excitantes se relacionan con el RNA, que en un momento dado se vio como poco más que un medio para producir proteínas. Sin embargo, en la última década las moléculas de RNA han pasado a ser vistas como agentes cruciales para el control de la información del genoma celular. La comprensión de cómo funciona el RNA ha llevado a descubrimientos sobre algunas enfermedades y a impactantes nuevos horizontes para la biotecnología, incluyendo los intentos para usar el RNA en terapias que contrarresten las consecuencias de tener genes defectuosos.
Ayuste (corte y empalme) alternativo
El RNA del dogma central es el RNA mensajero (mRNA), que sirve como molde para hacer proteínas. En eucariotas, los RNA normalmente se tienen que procesar antes de producir la forma de mensajero. La mayoría de los RNA recién formados son como largos trenes con muchos furgones (vagones de mercancías), cuya carga se tiene que elegir para que se ajuste a las necesidades de diferentes clientes. Los vagones vacíos se quitan y los anteriores y posteriores a ellos se unen entre sí. El equivalente celular de los vagones vacíos son los intrones, grandes regiones dentro de los genes que no codifican proteínas. Las secuencias de DNA que codifican proteínas se denominan exones. A menudo un exón contiene la receta para un módulo de una proteína. Pero incluso estos no siempre se utilizan. Un cliente puede no querer todas las mercancías de un tren, de manera que algunos de los furgones con carga se pueden eliminar junto con los vacíos. Y una célula puede no necesitar todos los módulos de una proteína concreta, así que varios exones se pueden combinar para producir formas diferentes.
Este proceso, denominado ayuste alternativo, fue descubierto de forma independiente en 1977 por Richard Roberts de Biolabs de Nueva Inglaterra y Phillip Sharp del Instituto de Tecnología de Massachusetts (Massachusetts Institute of Technology), ambos en EE. UU. (Berget et al., 2000). El hallazgo fue tan importante que mereció el premio Nobel de Fisiología y Medicina en 1993w1. En esa época se pensaba que el ayuste era poco frecuente; Sharp estimaba que lo más probable era que solo un 5% de los genes humanos sufriera el ayuste alternativo. El secuenciado completo del genoma humano ha revelado que un gen contiene de promedio 8,4 intrones, y todos ellos se tienen que eliminar a través del ayuste. Aunque algunos RNA humanos probablemente siempre se procesan de la misma manera, los científicos estiman ahora que al menos el 75% sigue el ayuste alternativo.

Imagen cortesía de EMBL Photolab
Los genes humanos tienen considerablemente más intrones que los de la mayoría del resto de organismos; la media para moscas y otros insectos está entre 2,4 y 5,4 intrones por gen. (Pese a esto, quien tiene el récord mundial actual para el ayuste alternativo es un gen de la mosca de la fruta denominado dscam, que puede generar 38016 proteínas distintas). Algunos investigadores creían que el alto número de intrones en humanos quería decir que los genes se iban haciendo más complejos con el tiempo. Sin embargo, un estudio reciente del grupo de Detlev Arendt y Peer Bork del Laboratorio de Biología Molecular Europeo (European Molecular Biology Laboratory, EMBL) en Heidelberg, Alemania, muestra casi con toda certeza que el antiguo ancestro común de insectos y vertebrados tenía genes más similares a los humanos, con más intrones (Raible et al., 2005). En el curso de la evolución, los genes de moscas y otras especies que se reproducen con rapidez se han ido simplificando.
También hay interesantes diferencias entre especies en lo que se refiere a la longitud total de los intrones comparada con la de los exones dentro de los genes. En los genes del gusano Caenorhabditis elegans y de otras muchas especies, los intrones y los exones contienen aproximadamente el mismo número de “letras” (nucleótidos). La situación en humanos es muy diferente: los intrones de un único gen tienen con frecuencia decenas de miles de nucleótidos y son, en promedio, cinco veces más largos que los exones. Esto puede tener consecuencias en la evolución de los genes humanos, como se reveló en un estudio realizado por Cristian Castillo-Davis de la Universidad de Harvard, y Eugene Koonin y Fyodor Kondrashov del Centro Nacional para la Información sobre Biotecnología (National Center for Biotechnology Information), todos en EE. UU. (Castillo-Davis et al., 2002). La transcripción es un proceso lento y energéticamente costoso: producir RNA desde un único gen con enormes intrones puede requerir varios minutos y miles de moléculas de ATP. Los autores encontraron que los intrones de los genes que se utilizan con mayor frecuencia son de media 14 veces más cortos que los de aquellos genes que no se utilizan casi nunca. Su conclusión: la selección natural ha acortado los intrones en los genes más usados, ahorrando así tiempo y energía.
Una vez que hubo evolucionado la maquinaria celular necesaria para llevar a cabo el ayuste alternativo, esta se pudo utilizar de diferentes formas. Mezclando y agrupando módulos se producen proteínas que se comportan de forma distinta. Esto ayuda a producir tipos de células diversos y tiene un papel muy destacado en el desarrollo de diferentes tejidos. El ayuste alternativo del RNA llamado Slo del oído de pollo mejora la capacidad auditiva del ave al generar células sensibles a diferentes frecuencias del sonido. En las moscas, hay tres proteínas críticas que se procesan de manera diferente en machos que en hembras y esto da lugar a importantes diferencias entre ambos sexos. Aunque las hembras tienen dos cromosomas X y los machos solo uno, las hembras no producen el doble de la cantidad de proteínas a partir de los genes de sus cromosomas, y esto se debe a las diferencias en estas proteínas. Diane Lipscombe y sus colaboradores en la Universidad de Brown (Rhode Island, EE. UU.) han encontrado que el ayuste alternativo es especialmente corriente en el cerebro de ratones y otros mamíferos (Lipscombe, 2005). Algunas de las moléculas procesadas son cruciales para la memoria y el aprendizaje.
El ayuste puede condicionar un amplio abanico de enfermedades. La mitad de los afectados por la neurofibromatosis, una enfermedad genética grave en la que se desarrollan tumores a lo largo de nervios y otros tejidos, presenta mutaciones que cambian el ayuste de los RNA producidos por el gen de la neurofibromina. Los pacientes con beta-talasemia tienen una anemia que es el resultado de que haya proteínas beta-globina no funcionales en sus glóbulos rojos; esta grave enfermedad es producida por un fallo en el ayuste del gen responsable. Otros ejemplos son cambios en los genes BRCA1 (ligado al cáncer de mama) y CFTR (que produce la fibrosis quística). Se estima que cerca del 50% de las mutaciones en exones relacionadas con enfermedades afectan a la forma en que se ayustan los RNA. Ciertos tumores y enfermedades neurodegenerativas a menudo están acompañados por RNA procesados de forma inusual que normalmente no se encuentran en tejidos sanos.
Control de calidad
En 1979, Regine Losson y François Lacroute del CNRS en Estrasburgo, Francia, descubrieron que las células tienen un sistema para inspeccionar los RNA y llevar a cabo controles de calidad (Losson & Lacroute, 1979). Unas tres décadas de investigación han mostrado que el sistema no es perfecto pero que permite proteger las células eucariotas de los efectos dañinos de la mayoría de las mutaciones. Los fallos en genes que cambian la forma, la estructura o las funciones de una proteína normalmente tienen efectos perjudiciales para la célula. Los errores en el ayuste también producen ese tipo de moléculas, de manera que el sistema de inspección –denominado degradación del mRNA mediada por mutaciones terminadoras (nonsense-mediated mRNA decay, NMD)– tiene que estar en alerta todo el tiempo.
La NMD se produce cuando un RNA entra en el citoplasma celular, pero los científicos la han relacionado con el ayuste, que tiene lugar antes de que el RNA deje el núcleo (Sun et al., 2000). La célula acopla un complejo de proteínas en los sitios donde haya habido eliminación de intrones, como cuando se marca un vagón para indicar que se han quitado otros a su lado. El complejo consta de al menos seis proteínas y se denomina complejo de unión al exón (exon junction complex, EJC). Este complejo se sitúa en los sitios de ayuste y su posición tiene una importante influencia en el destino del RNA.
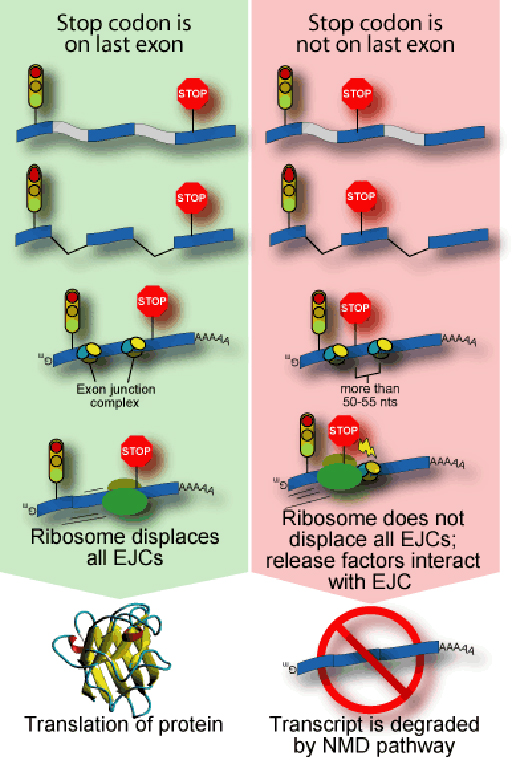
La traducción de un RNA para dar una proteína es llevada a cabo por una maquinaria molecular llamada ribosoma que se ancla sobre los RNA mensajeros, lee su mensaje y ensambla la cadena de aminoácidos que corresponda a la secuencia. Cualquier EJC que esté sobre la molécula simplemente se quita de en medio. Al final de la parte del RNA que codifica proteínas, el ribosoma encuentra una señal de tres letras denominada codón de parada y libera la proteína terminada. Las mutaciones a menudo alteran la secuencia de los RNA de manera que puede aparecer un codón de parada en medio de la molécula. Esto genera un código en el RNA que no tiene sentido para la célula (el ‘nonsense’ en la NMD) y permite que el EJC se quede sobre el RNA lo que promueve la acción de la NMD (ver figura).
Para evitar la producción de proteínas potencialmente perjudiciales, la NMD reconoce los EJC que se encuentran a más de 50 nucleótidos por detrás de un codón de parada. El proceso de traducción se interrumpe y otras moléculas vienen para retirar el RNA y degradarlo.
Pero algunos RNA se escapan de la NMD y producen proteínas dañinas que pueden causar enfermedades. E incluso cuando la NMD actúa, el resultado puede ser una enfermedad porque la NMD puede eliminar RNA dañado pero a pesar de ello necesario. En 1989, en el laboratorio de Lynne Maquat en el Instituto Conmemorativo del Dr. Roswell Park (Roswell Park Memorial Institute, Nueva York, EE. UU.) se demostró que la NMD contribuye a la beta-talasemia, la enfermedad genética más común en el mundo occidental. La beta-talasemia reduce la producción corporal de hemoglobina, que hace falta para transportar el oxígeno en la sangre. La enfermedad la padecen las personas que heredan una forma mutante del gen de la beta-globina; mediante la NMD se reconoce la mutación y el organismo degrada el RNA de la beta-globina –eliminando así una molécula importante (Lim et al., 1989). En este caso, un mecanismo cuyo fin es la seguridad produce daños en el organismo.
Hasta hace poco la NMD era considerada como poco más que un medio para interceptar RNA con errores; ahora se sabe que es una herramienta más general que la célula utiliza para controlar la cantidad y la calidad de ciertas moléculas. Esto sucede porque el proceso normal de ayuste alternativo algunas veces produce RNA con codones de terminación; por alguna razón, el proceso de cortar-y-pegar produce una zona de final de lectura en medio de un RNA. En 2004, R. Tyler Hillman, Richard Green y Steven Brenner de la Universidad de California, Berkeley, EE. UU., llevaron a cabo un análisis por ordenador que mostró que cerca de un tercio de las veces el ayuste alternativo coloca un codón de parada a más de 50 nucleótidos por delante de un sitio de ayuste. Esto activa la NMD, que elimina la mayoría de los RNA antes de que se traduzcan en proteínas (Hillman et al., 2004).
El mismo año, el grupo de Harry Dietz de la Facultad de Medicina de la Universidad John Hopkins (Maryland, EE. UU.) estudió este efecto en células de mamífero. Bloquearon la maquinaria NMD eliminando una proteína llamada Upf1, que es esencial para el proceso. Esto modificó el comportamiento de un elevado número de genes: cerca del 10% de los genes estudiados se hicieron más productivos, probablemente porque las formas generadas en el ayuste que normalmente habrían sido reconocidas y destruidas por la NMD se libraban de su eliminación (Mendell et al., 2004).
Esto significa que la NMD, como el ayuste alternativo, es una ruta opcional frecuentemente usada en la carretera entre genes y proteínas. Hay muchas más, algunas de las cuales se discutirán en un próximo ejemplar de Science in School. Si hay mucho suspense, aquí hay un acertijo para mantenerte ocupado: el color púrpura de algunas petunias se debe a un gen concreto de copia única. ¿Qué pasa con el color de esas petunias cuando se les añade una segunda copia del gen?
References
- Berget SM, Moore C, Sharp PA (2000). Spliced segments at the 5’ terminus of adenovirus 2 late mRNA. Reviews in Medical Virology 10: 355-371
- Castillo-Davis CI, Mekhedov SL, Hartl DL, Koonin EV, Kondrashov FA (2002) Selection for short introns in highly expressed genes. Nature Genetics 31: 415-418. doi:10.1038/ng940
- Crick F (1970) Central Dogma of Molecular Biology. Nature 227: 561-563
- Hillman RT, Green RE, Brenner SE (2004) An unappreciated role for RNA surveillance. Genome Biology 5: R8. doi:10.1186/gb-2004-5-2-r8
- Lim S, Mullins JJ, Chen CM, Gross KW, Maquat LE (1989) Novel metabolism of several beta zero-thalassemic beta-globin mRNAs in the erythroid tissues of transgenic mice. The EMBO Journal 8(9): 2613-2619
- Lipscombe D (2005) Neuronal proteins custom designed by alternative splicing. Current Opinion in Neurobiology 15: 358-363. doi:10.1016/j.conb.2005.04.002
- Losson R, Lacroute F (1979) Interference of nonsense mutations with eukaryotic messenger RNA stability. Proceedings of the National Academy of Sciences USA 76: 5134-5137
- Mendell JT, Sharifi NA, Meyers JL, Martinez-Murillo F, Dietz HC (2004) Nonsense surveillance regulates expression of diverse classes of mammalian transcripts and mutes genomic noise. Nature Genetics 36: 1073-1078. doi:10.1038/ng1429
- Raible F et al. (2005) Vertebrate-type intron-rich genes in the marine annelid. Platynereis dumerilii. Science 310: 1325-1326. doi:10.1126/science.1119089
- Sun X, Moriarty PM, Maquat LE (2000) Nonsense-mediated decay of glutathione peroxidase 1 mRNA in the cytoplasm depends on intron position. EMBO Journal 19: 4734-4744. doi:10.1093/emboj/19.17.4734
Web References
- w1 – El texto de la conferencia del nobel Philip Sharp acerca del ayuste alternativo, ‘Split Genes and RNA Splicing’, está disponible en línea.
- En la nota de prensa del anuncio del premio Nobel se da una breve panorámica del trabajo de Richard Roberts y Philip Sharp.
- Para más información acerca de este premio Nobel, incluyendo las biografías de sus ganadores, ver here.
Resources
- Muchos artículos de Crick están disponibles de forma libre en la página web de Nature.
Institutions
Review
Los avances en biotecnología de los últimos 40 años han tenido impacto en áreas como la agricultura, la ciencia de los alimentos y la medicina. Revertir nuestro conocimiento científico del dogma central de la Biología molecular en aplicaciones útiles, especialmente en la investigación sobre enfermedades y tratamientos médicos, requiere una comprensión más detallada de las funciones de las biomoléculas.
Esta serie presentará los avances y las nuevas teorías que son demasiado recientes como para aparecer en los libros de texto. También ilustrará los esfuerzos internacionales y universales por aprender más sobre cómo funcionan las moléculas biológicas.
Este artículo representa una excelente lectura auxiliar y algunas de sus partes se pueden usar para favorecer y examinar la comprensión por parte de los alumnos del dogma central, las propiedades del RNA o la síntesis de proteínas. En particular, el artículo sería aplicable a consideraciones sobre los usos de la biotecnología, la importancia de las investigaciones en curso y el reconocimiento del trabajo científico.
Marie Walsh, República de Irlanda





