Die Evolution des Menschen auf molekularer Ebene Understand article
Übersetzt von Johanna Scholbach. In seinem zweiten Artikel beschreibt Jarek Brykwie Wissenschaftler bis zu unseren Genen vordringen um die molekularen Grundlagen der menschlichen Evolution zu verstehen.

iStockphoto
Die DNA aller Individuen enthält zahlreiche Informationen über neue und vergangene evolutionäre Veränderungen. Sobald man Muster und Veränderungen in der DNA-Sequenz erkennt, kann man diese mit Sequenzen anderer Individuen vergleichen und herausfinden was mit den einzelnen Individuen geschehen ist. So ist es möglich zu entdecken, welche Gene oder Bestandteile des Genoms den einzelnen Spezies einen Vorteil gegenüber den anderen ermöglichten und ihnen somit ein besseres Überleben und Fortpflanzung garantierten.(die Erklärung der fettgedruckten Wörter findest du im Glossar).
In einem vorhergehnden Artikel (Bryk, 2010) habe ich schon ein paar Beispiele für solche günstigen Genveränderungen beim Menschen und anderen Organismen beschrieben. Zu zeigen welche genetischen Veränderungen nützlich gewesen sind, ist insbesondere beim Menschen relativ schwierig. Daher ist es eine umso größere Herausforderung die Mechanismen zu verstehen, die diese Veränderungen hervor gerufen und somit das Überleben und seine Fortpflanzungsfähigkeit verbessert haben.
In diesem Artikel möchte ich eine der Möglichkeiten vorstellen, die Wissenschaftler nutzen um zuerst die Regionen des Genoms zu identifizieren, die uns geholfen haben könnten zu überleben und uns fortzupflanzen. Erst danach finden sie heraus wie genau diese Abschnitte unsere Vorfahren mit Vorteilen ausgestattet haben.
Ein einfacher Weg mögliche vorteilhafte Regionen des Genoms zu identifizieren ist der Vergleich der DNA-Sequenzen vieler Individuen aus verschiedenen Pouplationen. An einem einfachen Beispiel erläutert heißt dies folgendes: wenn eine Population unter einem erhöhten Selektionsdruck stand (z.B. erhöhte UV-Strahlung in einem sonnigen Gebiet), der in der anderen Population nicht vorhanden war, dann sollte die jeweilige DNA-Sequenz für die entsprechende Anpassung (z.B. eine dunklere Hautfarbe) in den Populationen unterschiedlich sein.
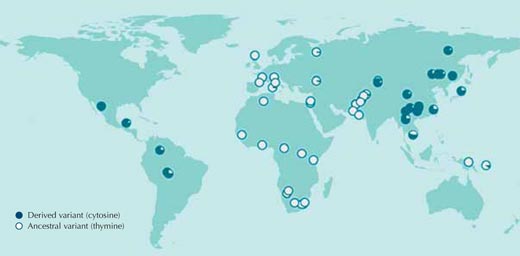
Angepasst mit der Erlaubnis von Macmillan Publishers Ltd: Nature, Sabeti et al. (2007), © 2007
Allerdings wissen wir in den allermeisten Fällen nicht welchem Selektionsdruck die Populationen in der Vergangenheit ausgesetzt waren oder welche Gensequenzen für die Anpassung verantwortlich sind. Draum fangen wir also nochmal von vorne an und vergleichen DNA-Sequenzen zwischen menschlichen Populationen ohne irgendeine Ahnung zu haben, was wir möglicherweise finden werden. Abbildung 1 zeigt einen solchen Vergleich für einzelene Nukleotide eines menschlichen Genoms.
Wenn Individuen unterschiedliche Nukleotide an einer bestimmten Position in der DNA-Sequenz aufweisen, spricht man von einem single nucleotide polymorphism(SNP, ausgesprochen “snip”). Von dieser Art von Variationen des menschlichen Genoms sind etwa drei Millionen in der für die Öffentlichkeit zugänglichen HapMap – Datenbankw1 katalogisiert. Die SNP-Skizze in Abbildung 1, rs3827760, kommt in zwei Varianten, sogenannten Allelen, vor: in diesem Fall ist es eine der beiden gezeigten Basen; entweder Thymin (T) oder Cytosin (C).
Jeder Kreis innerhalb der Abbildung steht für eine einzelne Population und veranschaulicht die Häufigkeit mit der die beiden Allele in der Population vorkommen.
Das Allel, welches Thymin enthält, kommt in allen afrikanischen und in den meisten europäischen Stichproben vor. Es fehlt jedoch fast vollständig bei Ostasiaten und Amerikanern. Dort hingegen ist die entsprechende Position in der Gensequenz am häufigsten mit Cytosin belegt (Sabeti et al., 2007, 2006; Xue et al., 2009).
Wenn wir diesen Vergleich mit all den anderen drei Millionen SNPs aus der HapMap machen würden, käme heraus, dass die Verteilung der rs3827760-Variante beim Menschen sehr ungewöhnlich ist. Diesem rs3827760 sollten wir also auf jeden Fall mehr Aufmerksamkeit schenken auch wenn die Verteilung noch lange nichts über den möglichen Nutzen der Varianten (den Anpassungswert) aussagt, geschweige denn ob es sich überhaupt um eine Anpassung handelt. Wir wissen nur, dass aus irgendeinem Grund Thymin, was unsere Vorfahren in Afrika an dieser Position trugen, durch Cytosin ersetzt wurde und sich diese Veränderung in ganz Ostasien und Amerika verbreitet hat. Den Zeitpunkt der Änderung können wir zwar schätzen, dennoch bleibt er sehr ungenau: irgendwann vor 1000 bis 70’000 Jahren hatten alle Menschen in Ostasien diese Cytosin-Variante.
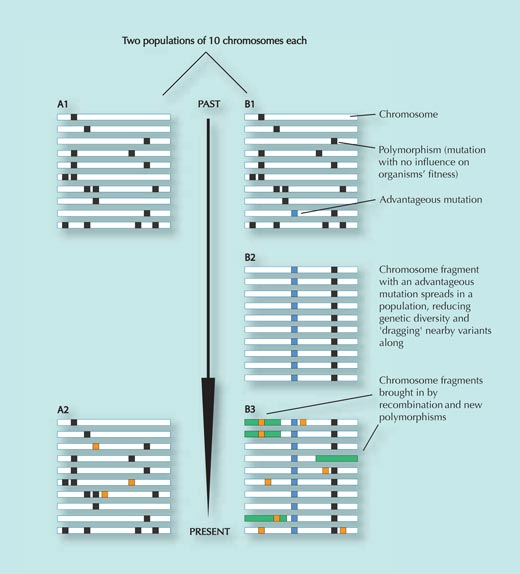
Population A stand nicht unter Selektionsdruck und hat sich nicht verändert. Es gibt ausschließlich ein paar zufällige Veränderungen, die aber für die Fitness keine Rolle spielen (orangene Quadrate in Feld A2; Varianten, die die Fitness reduzieren sind aus der Population entfernt wurden) – vergleiche Feld A1 und A2.
Population B zieht in eine neue Umgebung und ist dort, auf welchem Weg auch immer, neuem Selektionsdruck ausgesetzt. In dieser neuen Umgebung löst eine kleine genetische Veränderung (das blaue Quadrat in Feld B1) einen Vorteil aus für diejenigen, die es beseitzen und breitet sich schnell in der Population aus (Individuen, die die Veränderung tragen zeugen mehr Nachkommen). Die Gensequenzen die sich nah an der SNP befinden werden mit ihr mitgezogen (je näher die zwei Sequenzen liegen, desto geringer ist die Wahrscheinlichkeit, dass sie während der Rekombination, also wenn Teile der DNA zwischen mütterlichen und väterlichen Chromosoemen ausgetauscht werden, getrennt werden – siehe Abbildung 3).
Das Ergebnis dieser schnellen Ausbreitung der spezifischen DNA-Sequenz hat eine Verminderung der genetischen Variabilität in der Population zur Folge; die meisten Inidividuen werden die vorteilhafte SNP mit denen ihr naheliegenden Gen-Sequenzen (vergleiche mit Feld B1 und B2) besitzen.
Allerdings werden nach einiger Zeit neue genetische Veränderungen und die Rekombination dafür sorgen, dass neue Varianten entstehen (grüne Rechtecke und orangene Quadrate in Feld B3). Je länger also die Ausbreitung der ursprüunglich ausgewählten Genvariante zurück liegt, desto schwieriger ist es das Muster der reduzierten Variabilität (B2) zu finden, da es möglicherweise von anderen Veränderungen (B3) überdeckt wird
Mit freundlicher Genehmigung von Jarek Bryk
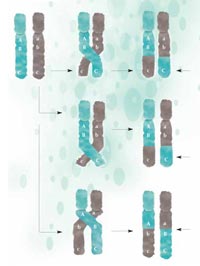
Rekombination werden Teile
der DNA zwischen
mütterlichen (grün) und
väterlichen (grau)
Chromosomen ausgetauscht
und in einer neuen
Zusammensetzung an die
nächste Generation weiter
gegeben. Je näher zwei
Regionen beieinander liegen,
desto unwahrscheinlicher ist
es, dass sie während der
Rekombination getrennt
werden: A und B liegen näher
zusammen als B und C,
desshalb werden sie höchst
wahrscheinlich nicht
getrennt. Zum Vergrößern
auf das Bild klicken
Mit freundlicher Genehmigung
von Nicola Graf
Wie können wir dann herausfinden ob dieses Muster zur posiviten Selektion (Cytosin hat Ostasiaten und Amerikanern einen Vorteil verliehen) beigetragen hat oder ob es einfach nur zufällig entstanden ist? Um zu erkennen ob die DNA-Veränderung (Thymin oder Cytosin) positiv selektiert wurde, schauen wir auf die DNA-Sequenzen die sich neben der SNP befinden. Wenn die DNA-Sequenzen, die rs3827760 umgeben in allen Populationen gleich sind, dann haben wir keinen Beweis dafür, dass sich die SNP auf die Fitness. des Organismus ausgewirkt hat. Wenn jedoch eine Population (z.B. die Ostasiaten) unter einem speziellen Selektionsdruck stand und sich rs3827760 in folge dessen verändert hat um sich an den Selektionsdruck anzupassen, dann unterscheiden sich die DNA-Sequenzen neben der SNP zwischen den Bevölkerungen. Um das zu verstehen, kannst du dir Abbildung 2 anschauen.
Schaut man sich jetzt die DNA-Sequenzen neben rs3827760 an und vergleicht diese, dann wird schnell klar, dass die Variabilität um das Cytosinmolekül in der ostasiatischen Bevölkerungsgruppe wesentlich geringer ist, als die Variabilität um das Thyminmolekül in der afrikanischen und der europäischen Population (die Amerikaner wurden nicht getestet). Das weißt darauf hin, dass die positive Selektion dafür verantwortlich war, dass die Cytosin-Variante sich in der ostasiatischen Bevölkerung ausgebreitet hat. Aber hatte es überhaupt irgendeine Auswirkung, dass diese spezielle SNP tatsächlich ausgewählt wurde?
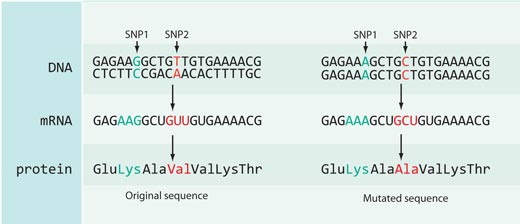
Nicht alle Änderungen der DNA-Sequenz wirken sich auch tatsächlich auf die Proteinsequenz aus: die meisten SNPs die in der HapMap registriet sind, liegen entweder in einem nichtkodierenden Abschnitt des Genoms (z.B. zwischen den Genen) oder sie liegen in dem kodierenden Bereich aber sie rufen keine Veränderung in der kodierten Proteinsequenz hervor (siehe Abbildung 4).
Mit freundlicher Genehmigung von Jarek Bryk
Bei unserem Beispiel dem rs3827760 haben wir gearde Glück: es liegt in dem kodierenden Bereich des Gens – und zwar am Ende eines Gens, das EDAR genannt wird. Es ist verantwortlich für die Entwicklung der Haarfollikel, Schweißdrüsen und Zähne. Außerdem hat die Umwandlung von Thymin in Cytosin in unserer DNA-Sequenz eine Veränderung der Proteinsequenz zur Folge: Afrikaner und Europäer (die die SNP-Variante mit Thymin besitzen) haben die Aminosäure Valin an der Position 370 des Proteins, wohingegen Ostasiaten und Amerikaner (mit der Base Cytosin) die Aminosäure Alanin besitzen. Dieser Teil des Proteins steht in Zusammenhang mit anderen Protein und Mutationen, die dafür bekannt sind ektodermale Dysplasien in Menschen und Mäusen hervor zurufen. Bei der ektodermalen Dysplasie handelt es sich um eine Gruppe von Erkrankungen, bei denen es zur Fehlentwicklung von Haaren, Zähnen und Schweißdrüsen kommt (siehe Abbildung 5).
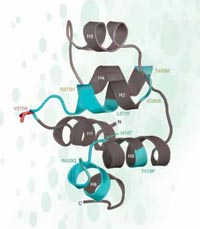
Struktur eines Teils des
EDAR-Proteins. Die
grün-gefärbten Mutationen
rufen ektodermale
Dyspalsien beim Menschen
hervor. Die vermutliche
ausgewählte SNP ist rot
markiert
Angepasst mit freundlicher
Genehmigung von Macmillan
Publishers Ltd: Nature, Sabeti
et al. (2007), © 2007
Um zu sehen ob die Veränderung der Proteinsequenz auch wirklich die Funktion des Proteins beeinflusst, führen wir ein paar Experimente durch mit denen wir biochemischen Signalwege untersuchen, in denen das EDAR-Protein vorkommt:es handelt sich hier um eine Vielzahl von Reaktionen, die in die Entwicklung der Haarfollikel, Schweißdrüsen und Zähne eingebunden sind. Als diese Tests durchgeführt wurden, fand man heraus, dass die Alanin-Variante des Proteins (die man bei Ostasiaten und Amerikanern fand und die durch die Cytosin-SNP-Variante kodiert wird) den Signalweg schneller ablaufen lies als die Valin-Variante (die man bei Afrikanern und Europäern fand und die durch die Thymin-SNP-Variante kodiert wird). Das macht sich vorallem in einem anschaulichen Beispiel bemerkbar: die Menschen mit der Alanin-Variante haben dickere Haare als die Menschen mit der Valin-Variante. Am Beipsiel der Maus lässt sich dieser Sachverhalt auch gut darstellen. Die Mäuse wurden genetisch manipuliert um eine erhöhte Aktivität des EDAR-Weges zu erreichen. Diese Mäuse hatten sowohl sichtbar dichteres Fell mit dickeren Haaren als auch größere Schweißdrüsen, als Mäuse mit einer normalen EDAR-Aktivität (Chunyan et al., 2008; Chang et al., 2009).
Alles in allem heißt das, dass die zwei SNP-Varianten (die entweder Thymin oder Cytosin enthalten) sowohl die Struktur als auch die Funktion des EDAR-Proteins beeinflussen und, dass dies auch zu physischen Unterschieden geführt hat. Und zwar zu Unterschieden in der Haardicke sowie möglicherweise in der Größe der Schweißdrüsen.
Man darf nich vergessen, dass die Unterschiede in der DNA-Sequenz, die wir bisher betrachtet haben dem Wunder eines großen natürlichen Experimentes gleichkommen und wir nur mutmaßen können unter welchem Selektionsdruck die Asiaten und Amerikaner gestanden haben. Wir wissen also nicht, was zur Ausbreitung des Cytosin-Allels geführt hat. Aber im Zusammenspiel mit Genomuntersuchungen, Experimenten und Tiermodellen sind wir in der Lage die funktionellen Unterschiede zwischen verschiedenen Populationen oder Spezies aufzudecken. So sind wir in der Lage die molekulare Grundlage vergangener Anpassungen unserer Vorfahren und anderer Organismen zu verstehen und zu belegen wie wir es geschafft haben uns an eine sich ständig verändernde Umwelt anzupassen.
Glossar
Anpassungswert: Ein Merkmal hat einen Anpassungswert, wenn es es dem Individuum ermöglicht in einer Umgebung zu überleben und sich besser fortzupflanzen als Indidivduen, die dieses Merkmal nicht ausprägen. Etwas wissenschaftlicher gesprochen heißt das: ein Merkmal hat einen Anpassungswert, wenn es die Fitness steigert.
Fitness: ein schwierig zu erklärender Begriff aus der Evolutionsbiologie und Populationsgenetik; er beschreibt die durchschnittliche Zahl der Nachkommen in einer Generation mit einem bestimmten Genotyp, verglichen mit einem anderen Genotyp in der selben Population.
Genom: in der Regel handelt es sich um die gesamte DNA, die sich im Zellkern befindet. Hierzu gehören nicht die mitochondriale oder Plastid-DNA. Wenn du es genauer wissen willst, dann schau auf die Webseite “What is a genome” von der US National Library of Medicinew2.
Positive Selektion: Die natürliche Selektion ist ein Mechanismus der Evolution und beschreibt das unterschiedliche Überleben und Fortpflanzen von Individuen in einer bestimmten Umgebung. Von positiver Selektion spricht man dann, wenn das Individuum aufgrund eines bestimmten Merkmals besser als andere Indiviuen in der Lage ist sich fortzupflanzen und zu überleben.
Selektionsdruck: Ein Merkmal der Umwelt (z.B. Temperatur, Vorhandensein von Parasiten, Angriffslustigkeit und Aggressivität der Mitglieder der selben Spezies), das unterschiedliches Überleben und Fortpflanzen der Individuen gewährleistet.
SNP: Abkürzung für “single nucleotide polymorphism”. Übersetzt heißt das soviel wie Veränderung eines Nukleotids der DNA und steht für die Veränderung einer Base in einer DNA-Sequenz, die bei anderen Individuen nicht vorkommt. SNP wird “snip”.
References
- Bryk J (2010) Natürliche Selektion auf molekularer Ebene. Science in School 14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE 4(10): e7591. doi:10.1371/journal.pone.0007591
- Dieser Artikel beschreibt den Phenotyp unterschiedlicher Drüsen von Mäusen bei denen der EDAR-Signalweg gehemmt wurde. Außerdem wird überlegt, welche Merkmale in der Entwicklunggsgeschichte des Menschen positiv selektiert worden sind. Der Artikel ist auf der Zeitungsseite: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form. Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- Dieser Artikel enthält detailierte Informationen zu den in-vitro Untersuchungen von EDAR und den transgenen Mäusen und beinhaltet sehr schöne Bilder und Photos.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle. Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage. Science312(5780): 1614-20. doi: 10.1126/science.1124309
- Es handelt sich hier um einen guten Überblick über die verschiedenen Methoden, die verwendet werden, um die positive Selektion auf genetischer Ebene zu betrachten.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations. Nature 449: 913-918. doi: 10.1038/nature06250
- Dieser Artikel kann gratis von Science in School hier, oder vonwww.nature.com/subscribe herunter geladen werden.
- Er beschreibt eine der vielen Möglichkeiten das gesamte Genom nach positiver Selektion ab zu suchen.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation. Genetics 183(3): 1065-77. doi:10.1534/genetics.109.107722
- Dieser Artikel enthält eine Diskussion über EDAR und vergleichbare Gene. Sie können ihn kostenlos von PubMed Central herunterladen:www.ncbi.nlm.nih.gov/pmc oder über den direkten Link:http://tinyurl.com/26xte2h verfügbar.
Web References
- w1 –Das ‘Hap Map Project’ ist ein Zusammenschluss von Wissenschaftlern und Leistungsträgern aus Kanada, China, Japan, Nigeria, Großbritanien und den USA. Sie beschäftigen sich mit der Erstellung einer Datenbank, die Wissenschaftlern dazu dienen soll die für Krankheiten verantwortlichen Gene zu finden und den Einfluss von Medikamenten auf die Gene zu testen. Siehe: www.hapmap.org
- w2 – Wenn du mehr über das Genom und das ‘Human Genome Project’ erfahren möchtest, kannst du dir ‘What is a genome’ auf der Website der US National Library of Medicine anschauen: http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
Vergiss dein gesamtes Wissen über das menschliche Genom, die genaue Funktion von großen Segmenten und wie und warum DNA-Sequenzen, die sich in Bevölkerungen verändert haben, lange unentdeckt blieben. Eine evolutionäre Anpassung hat beim Menschen natürlich stattgefunden, jedoch ist es sehr schwer diese Veränderungen zu zeigen. Dieser Artikel zeigt wie eine dieser Veränderungen entdeckt wurde. Experimente an genetisch veränderten Mäusen haben gezeigt, wie die Änderung einer einzelnen Base der DNA, zu einer Änderung der Aminosäuresequenz des Proteins und auch zur Veränderung der Funktion des Proteins führt. Das kann dann in einer Änderung des Phänotyps resultieren.
Im Schulunterricht kann dieser Artikel im Zusammenhang mit der Codesonne und der Degeneriertheit des genetischen Codes, der Proteinstruktur und -funktion und der Populationsgenetik verwendet werden. Er vermittelt auch Hintergrundwissen über menschliche Bevölkerungsgruppen und dient zur Vorstellung des Sanger-Instituts sowie des ‘Human Genome Mapping Project’.
Die Schüler können über evolutionäre Vorteile sprechen und dabei auf die verschiedenen Aspekte, die in dem Artikel beschrieben werden eingehen. Es können Selektion, Populationsgenetik und das Hardy-Weinberg-Gesetz disskutiert werden. Um diese Diskussion zu vervollständigen gibt es eine hervoragende Aufgabe in Ausgabe 6 von Science in School (Pongsophon et al., 2007).
Einige Verständnissfragen zum Text:
- Beschreibe mit deinen eigenen Worten, was eine SNP ist und erkläre es an einem Beispiel, das nicht im Text vorkommt.
- Erkläre die Signifikanz der SNPs.
- Für welche Aminosäure kodiert das Triplet GTT?
- Beschreibe die Veränderung die in Mäusen mit einem genetisch veränderten EDAR-Weg gefunden wurden und schlage Möglichkeiten vor, mit denen diese beobachteten Veränderung gemessen werden können.
Shelley Goodman, UK





