Wie man die Strategien von Influenzaviren austricksen kann Understand article
übersetzt von: Veronika Ebert, Höhere Bundeslehr- und versuchsanstalt für chemische Industrie, Wien. Influenzaviren zu bezwingen, kann viel bedeutender sein als nur ein lästiges Ärgernis zu beseitigen: dieser Krankheitserreger ist für die tödlichsten Pandemien der jüngeren Geschichte…
Mit freundlicher Genehmigung
von EMBL Photolab
Es wirkt wie ein permanenter Krieg – kaum hat man den neuesten Sicherheitsupdate für den eignen Computer herunter geladen, hat ein Hacker bereits eine neue Schwachstelle gefunden. Schon ist der Computer wieder gefährdet von einem Virus, von Spyware oder bösartigen Programmen attackiert zu werden, solange bis ein neuerlicher Sicherheitsupdate das Schlupfloch schließen kann.
Die Zellen in unserem Körper haben ähnliche Probleme: sie verfügen ebenfalls über zahlreiche Sicherheitssysteme, die vermeiden sollen, dass seine Stoffwechselmechanismen von einem Virus überfallen werden. Aber auch diese Systeme sind nicht perfekt, und viele Viren haben heimtückische Strategien entwickelt, um sie auszutricksen. Eine Gruppe von Wissenschafter/innen der FLUPOLw1 EU-Initiative versucht, diese Strategien zu verstehen um die Sicherheitsschranken des Körpers zu verbessern. Sie haben eine der Taktiken des Influenzavirus aufgeklärt. Dadurch könnte der Weg zur Entwicklung neuer Medikamente geebnet sein, die zur Bekämpfung zukünftiger Pandemien geeignet sind.
Influenza ist ein weltweites, schwerwiegendes Problem für Regierungen und Gesundheitsorganisationen. Obwohl Infektionen mit gängigen Virenstämme selten tödlich verlaufen, können sie für Ältere, Kranke oder immungeschwächte Personen fatale Folgen haben. Und noch viel schlimmer: laufend entstehen neue Stämme, die sich als viel gefährlicher als ihre Vorgänger herausstellen könnten. Die größte Gefahr geht von Vogelgrippeviren aus, die die Fähigkeit erlangen könnten, Menschen effizient zu infizieren. Viren, die diese Veränderung durchlaufen haben, erwiesen sich in der Vergangenheit als besonders todbringend. So stammte der Erreger der Grippepandemie (gr. pan: alle; demos: Volk; eine Epidemie einer Infektionserkrankung, die sich über große Regionen erstreckt, wie z.B. einen Kontinent, oder sogar die ganze Welt) von 1918 ebenfalls aus Vögeln. Bei ihrer Verbreitung um die ganze Welt wurden zwischen 50 und 100 Millionen Menschen getötet.
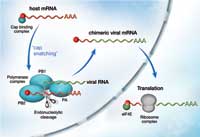
die Kappe der Wirts-mRNA mit
Hilfe des Proteins PB2, das Teil
des Polymerasekomplexes ist.
Wenn diese Kappe angefügt ist,
kann die virale RNA von Ribsomen
im Cytoplasma zu Proteinen
translatiert werden und weitere
Kopien des Virus werden
hergestellt. Zum Vergrößern auf
das Bild klicken
Mit freundlicher Genehmigung
von Petra Riedinger
In letzter Zeit hat ein Vogelgrippevirusstamm mit der Bezeichnung H5N1 zunehmende Besorgnis hervorgerufen (siehe auch Niekoop & Rienks, 2006). Dieser Stamm hat bereits einige hundert Menschen infiziert und getötet. Falls er einmal die Fähigkeit erwirbt, von Mensch zu Mensch übertragen zu werden, könnte eine neue tödliche Pandemie drohen. Es gibt zwar Grippeimpfstoffe, diese bieten in der Regel aber nur vor einem Virusstamm vollen Schutz, während sie gegenüber anderen Stämmen wenig effizient sind. Da es relative lange dauert, einen neuen Impfstoff zu erzeugen, könnte dieser zu spät kommen, um eine neue Pandemie zu verhindern. Neue Strategien der Verbreitung des Virus Einhalt zu gebieten, wären dringend erforderlich.
Wenn ein Virus eine Wirtszelle befällt, versucht es, möglichst viele neue Viruspartikel zu bilden, die ihrerseits weitere Zellen befallen können. Ein virales Enzym, eine Polymerase, die aus drei Untereinheiten aufgebaut ist, ist dafür hauptverantwortlich. Sie kopiert das Erbmaterial des Virus und steuert die Wirtszelle so, dass virale Proteine hergestellt werden. Der Fachbegriff für den Prozess dieser Steuerung heißt “CAP snatching” (engl. „snatch”: schnappen) siehe Bild unten. Das CAP ist ein zusätzliches kurzes RNA-Stück am Anfang jeder messenger RNA (mRNA). Es ist erforderlich, um die Proteinsynthesemaschinerie zu ihrem Startpunkt zu leiten, sodass ein neues Protein nach in mRNA enthaltenen Information synthetisiert werden kann. Die virale Polymerase entfernt dieses CAP von den Wirtszell-RNA-Molekülen: sie bindet an das CAP der Wirtszell-RNA´s, schneidet es ab und verbindet es mit einem viruseigenen RNA-Molekül. Bisher war unklar, wie die Polymerase dieses Ziel genau erreicht, und welche Untereinheit welche Funktion übernimmt. Da sich die Arbeitsweise viraler Polymerasen möglicherweise fundamental von jener ihrer menschlichen Pendants unterscheidet, wären solche Polymerasen ein idealer Angriffspunkt für die Blockade durch Medikamente, die die Verbreitung der Infektion im Körper eindämmen könnten. Die Gefahr von Nebenwirkungen wäre dementsprechend gering.
Mehr über die Polymerase zu erfahren, wäre auch wichtig, um zu verstehen, wie Influenuzaviren auf andere Arten überspringen können. Viele der Mutationen, die es Vogelinfluenzastämmen ermöglichen, sich an den Menschen anzupassen, liegen in jenem Gen, das für die Polymerase kodiert. Es wäre daher sehr wichtig herauszufinden, welche Auswirkungen diese Mutationen haben. Da diese Fragen nicht einfach zu beanworten sind, benötigt man ein breites Spektrum an Fachwissen. Um diese Herausforderung zu bewältigen, wurde die FLUPOL EU-Initiative gebildet: Stephen Cusack’s und Darren Hart’s Arbeitsgruppen an der EMBL Außenstelle in Grenoblew2, Frankreich und Kollegen/innen von der “Grenoble Unit of Virus Host Cell Interactionsw3, haben sich mit anderen europäischen Arbeitsgruppen in Madrid, Lyon, London und Marburg zusammen geschlossen. „Wir verfolgen einen multidisziplinäres Ansatz, dieses Enzym zu studieren, von der Bioinformatik, über Strukturuntersuchungen, bis zu Mausmodellen zur Untersuchung der Übertragung von Art zu Art“, sagt Stephen.
Aber die Polymerase gibt ihre Geheimnisse nicht so leicht preis. Lange war es unmöglich, ihre dreidimensionale Struktur aufzuklären. Dazu müssen die Forscher/innen die Untereinheiten der Polymerase in löslicher Form produzieren und anschließend kristallisieren. Die Struktur dieser Proteine kann anschließend durch Röntgenbestrahlung ermittelt werden. Dadurch kann die Arbeitsweise der Proteine besser verstanden werden. Eine Einleitung zum Thema „Kristallographie“ findet sich in dem Artikel von Dominique Cornuéjols in dieser Ausgabe. Auch im Unterricht ist es möglich, Proteine zu kristallisieren (Blattmann & Sticher 2009).
Lange Zeit gelang es niemandem, die erforderlichen Kristalle herzustellen. Wann immer die Forscher/innen dies versuchten, bekamen sie entweder unbrauchbare, unlösliche Klumpen, oder sie konnten überhaupt nicht genug Protein für eine Kristallisation herstellen. Die übliche Strategie, dieses Problem zu lösen, verlief frustrierend: die Proteine in ihre einzelnen Arbeitsstrukturen, sogenannte Domänen zu fragmentieren, in der Hoffnung, dass diese löslich sind. Diese Methode funktioniert nur, wenn man die einzelnen Domänen vorher identifizieren kann. Normalerweise können Domänen erkannt werden, wenn die Aminosäuresequenz des Proteins mit jener bekannter Proteine verglichen wird, deren Struktur bekannt ist. Da es für die Influenzapolymerase leider kein vergleichbares Protein gab, war dieser Weg nicht gangbar.

Mit freundlicher Genehmigung
von EMBL Photolab
Die Lösung fand sich in Form eines neuen, von Darren entwickelten automatisierten Systems, dem ESPRIT (Expression of Soluble Proteins by Random Incremental Truncation; „Herstellung von löslichen Proteinen durch zufällige, zunehmende Verkürzung“). Dieses System ermöglicht es den Wissenschafter/innen, alle möglichen Fragmente eines Proteins rasch und einfach auf lösliche Domänen hin zu durchsuchen. 2007 gelang es Darren und Stephen mit ESPRIT das erste lösliche Fragment von PB2, einer der drei Untereinheiten (PA, PB1, PB2), aus der die Influenzapolymerase zusammengebaut ist, zu gewinnen (Tarendeau et al., 2007). Die Aufklärung dieser Struktur ermöglichte es, zu verstehen, wie sich die PB2 Untereinheit in den Zellkern transportieren lässt, wo sie sich mit den beiden anderen Untereinheiten zur funktionstüchtigen Polymerase vereint wird.
Stephen and Darren interessierten sich besonders für einen anderen Abschnitt von PB2, der eine entscheidende Rolle für die Abtrennung des CAP´s spielt. Die PB2-Untereinheit bindet an das CAP der Wirtszell-mRNA, dann schneidet die PB1-Untereinheit der Polymerase das CAP ab und verbindet es mit dem Anfang der viralen mRNA. Die gecappte virale mRNA kann dann von der Wirtszellmaschinerie erkannt werden, virale Proteine können auf Kosten der wirtseigenen Proteine gebildet werden. Ohne CAP kann eine mRNA nicht in Protein übersetzt werden. Daher könnten Stoffe, die die Bindung von PB2 an CAP blockieren, die Virusvermehrung abstoppen.
Bis jetzt wusste man allerdings wenig darüber, wie PB2 arbeitet. „Obwohl jahrelang daran gearbeitet worden ist, wusste niemand genau, wo die CAP-bindende Domäne wirklich liegt”, sagt Stephen.
ESPRIT ermöglichte es Darren, Stephen und ihren Kollegen/innen den Abschnitt von PB2 zu identifizieren, der an das CAP bindet. Mit Hilfe der Kristallisationseinheit am EMBL in Grenoble und den intensiven Synchrotronröntgenstrahlen an der European Synchrotron Radiation Facility (ESRF)w4 (siehe auch Cornuéjols, 2009), gelang es ihnen, die Struktur der CAP-bindenden Domäne von PB2 bei der Bindung eines CAP aufzuklären. Es zeigte sich, dass diese Domäne eine bisher völlig unbekannte Struktur hat. „Sie hat eine völlig andere Faltung als andere Proteine”, sagt Stephen.
Aber obwohl diese Struktur völlig neu war, ist die Art, wie sie an das CAP bindet, vergleichbar mit jener anderer CAP-bindender Proteine: die zentrale Wechselwirkung ist ein Sandwich von zwei Aminosäuren von PB2, die das CAP von beiden Seiten umklammern (siehe Abbildung Seite 27). „Das ist ein schönes Beispiel für konvergente Evolution, bei der völlig unterschiedliche Proteine eine ähnliche Lösung gefunden haben”, sagt Darren.
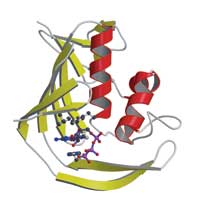
Influenzavirus PB2-Proteins
zeigt, wie das Virus das CAP-
Molekül seines Wirts entfernt.
PB2 bindet das CAP (markiert),
indem es dieses mit zwei
aromatischen Aminosäuren
umklammert. Zur Vergrößerung
des Bildes klicken.
Mit freundlicher Genehmigung
von Petra Riedinger
Um zu beweisen, dass das PB2-Fragment tatsächlich der böse CAP-Dieb ist, kreierten Darren und Stephen Fragmente, in denen jene Aminosäuren, die das CAP umklammern, mutiert sind. Tatsächlich banden die meisten dieser Fragmente das CAP nicht mehr. Außerdem konnten die FLUPOL Kooperationspartner am Centro Nacional de Biotecnologia in Madridw5, zeigen, dass die gleichen Mutationen die Fähigkeit der Polymerase, neue Viren zu bilden, blockierten. „Wir konnten die essentielle Rolle dieser Aminosäuren für das gesamte Virus zeigen”, sagt Stephen (Guilligay et al., 2008).
Nun können die Kenntnisse dieser Struktur verwendet werden, um relative einfache, kleine Moleküle zu designen, die die Aktivität von PB2 blockieren. „Wenn man die Abtrennung des CAP´s verhindern kann, tötet man das Virus”, sagt Stephen. Er und Darren bereiten sich jetzt vor, geeignete Stoffe zu finden, die genau das tun. Sie verwenden die Kenntnisse der dreidimensionalen Struktur aber nicht nur, um aktiv einen Inhibitor zu designen. Zusätzlich nützen sie ihre Möglichkeiten aus, um große Mengen an PB2-Fragment herzustellen, damit unzählige chemische Verbindungen auf ihrer Fähigkeit an PB2 zu binden getestet werden können – potentielle PB2-Inhibioren. “Wir sind die Ersten, die hinreichende Mengen dieses Fragments erzeugen können, um diesen radikalen Ansatz zur Identifikation eines kleinen inhibierenden Moleküls vornehmen zu können”, meint Darren.
Kürzlich haben Wissenschafter/innen am UVHCI und von Stephens Gruppe herausgefunden, dass ein Teil der PA-Untereinheit, der dritten Untereinheit der Polymerase, für die Entfernung des CAP von der Wirts-mRNA verantwortlich ist (Dias et al., 2009). Das war eine große Überraschung, weil alle dachten, dass die Schneideaktivität an einer anderen Stelle der Polymerase lokalisiert ist. Mit PA konnte ein weiterer viel versprechender Angriffspunkt für antivirale Agentien identifiziert werden.
Die Wissenschafter/innen produzierten Kristalle der ausschlaggebenden PA-Domäne und untersuchten sie mit den kräftigen Röntgenstrahlen des ESRF in Grenoble. Die dabei entstandenen hochauflösenden Bilder zeigen die einzelnen Aminosäuren des aktiven Zentrums, das für die Abtrennung des CAP´s von der mRNA verantwortlich ist. Ein schmaler Canyon in der Mitte der PA-Domäne schnappt sich die lange mRNA, und der Metallkomplex an den oberen Enden des Canyons schneidet das CAP ab. Weitere Experimente am ESRF erhärteten den Befund, dass diese Metallkomplexe Mangan enthalten. Das ist ein wichtiger Hinweis für die Entwicklung von antiviralen Stoffen, weil diese Metallionen bei der Entwicklung verbesserter Inhibitoren berücksichtigt werden müssen. Der Trick wird sein, die Kenntnis der Struktur der aktiven Regionen dieser Polymeraseuntereinheit, die entweder für die Ligandenbindung, oder für die enzymatische Aktivität verantwortlich sind, auszunutzen, um kleine, bindende Inhibitoren zu designen, die diese Stellen spezifisch blockieren, und dadurch die Virusvermehrung verhindern.
Diese Entdeckungen haben den Wissenschafter/innen ein nahezu vollständiges Bild des Mechanismus der CAP-Abtrennung vermittelt. Nun sind die Forscher/innen damit beschäftigt, mit Hilfe von ESPRIT den Rest der Influenzaviruspolymerase aufzuklären, um – hoffentlich bereits in naher Zukunft, neue Wege zu finden, auf die schmutzigen Tricks von Influenzaviren zu kontern.
References
- Blattmann B, Sticher P (2009) Growing crystals from protein. Science in School 11: 30-36. www.scienceinschool.org/2009/issue11/lysozyme
- Cornuéjols D (2009) Biological crystals: at the interface between physics, chemistry and biology. Science in School 11: 70-76. www.scienceinschool.org/2009/issue11/crystallography
- Dias A, Bouvier D, Crépin T, McCarthy AA, Hart DJ, Baudin F, Cusack S, Ruigrok RW (2009) The cap-snatching endonuclease of influenza virus polymerase resides in the PA subunit. Nature 458: 914-918. doi: 10.1038/nature07745. Laden Sie den Artikel hier kostenfrei herunter oder abonnieren Sie Nature today: www.nature.com/subscribe
- Guilligay D Tarendeau F, Resa-Infante P, Coloma R, Crepin T, Sehr P, Lewis J, Ruigrok RW, Ortin J, Hart DJ, Cusack S (2008) The structural basis for cap-binding by influenza virus polymerase subunit PB2. Nature Structural and Molecular Biology 15(5): 500-106. doi: 10.1038/nsmb.1421
- Niekoop L, Rienks F (2006) The ecologist’s view of bird flu. Science in School 3: 24-30. www.scienceinschool.org/2006/issue3/birdflu
- Tarendeau F, Boudet J, Guilligay D, Mas PJ, Bougault CM, Boulo S, Baudin F, Ruigrok RW, Daigle N, Ellenberg J, Cusack S, Simorre JP, Hart DJ (2007) Structure and nuclear import function of the C-terminal domain of influenza virus polymerase PB2 subunit. Nature Structural and Molecular Biology 14(3): 229-33. doi: 10.1038/nsmb1212
Web References
- w1 – Für weitere Informationen über die EU FLUPOL-Initiative siehe: www.flupol.eu
- w2 – Sie finden die Website der European Molecular Biology Laboratory (EMBL) Außenstelle in Grenoble unter: www.embl.fr
- w3 – Für weitere Informationen über die „Unit of Virus Host Cell Interactions“ in Grenoble siehe: www.uvhci.fr
- w4 – Für weitere Informationen über die European Synchrotron Radiation Facility (ESRF) in Grenoble, siehe: www.esrf.eu
- w5 – Für weitere Informationen über das Centro Nacional de Biotecnologia in Madrid siehe: www.cnb.uam.es





