Von römischen Straßen, Güterbahnhöfen und Kontrolleuren: neue Entdeckungen der RNA-Forschung Understand article
Übersetzt von Julia Heymann, MPIIB Berlin. RNA ist ein elementares biologisches Molekül, das in Lehrbüchern nur selten näher behandelt wird. Im ersten Artikel einer Serie berichtet Russ Hodge von spannender neuer Forschung an der RNA.

Florian Raible sehen nach,
wer sich am schnellsten oder
am langsamsten entwickelt
Freundlicherweise zur
Verfügung gestellt vom EMBL
Photolab
Stellen Sie sich einen antiken Römer vor, der auf einem Gipfel steht und über die Landschaft blickt, um den Verlauf einer neuen Straße zu planen. Zweitausend Jahre später sind diese Straßen immer noch zu erkennen. Sie durchqueren europäische Städte und Landschaften, als wären sie mit dem Lineal gezogen, nur den hartnäckigsten Hindernissen ausweichend.
In den fünfziger- und sechziger Jahren haben Wissenschaftler eine vergleichbare Route kartiert, als die Zusammenhänge zwischen verschiedenen Arten von Molekülen in der Zelle beschrieben wurden.
Ihr Entwurf ist als zentrales Dogma der Molekularbiologie bekannt: „von der DNA zur RNA zum Protein“, wie James Watson in eines seiner Notizbücher schrieb, als er und Francis Crick ihre Jagd auf die DNA-Struktur eröffneten. Neben diesem Leitsatz präsentierten die beiden eine neue Beziehung zwischen den Hauptmolekülen einer Zelle: einen gerichteten Informationsfluss von Nukleinsäuren zu Proteinen. Schon bald nach ihrer Entdeckung der Doppelhelix hielt Crick Vorlesungen, in denen er sein Dogma als das große Vorhaben der molekularbiologischen Forschung präsentierte (Crick, 1970): Wissenschaftler sollten die exakten zellulären Mechanismen enträtseln, die dieser Umwandlung zugrundelagen.
In den siebziger Jahren waren die Grundlagen der RNA- und Proteinsynthese bekannt, doch ofensichtlich konnte eine Einbahnstrasse nicht die die Chemie einer Zelle repräsentieren. So wie eine Handvoll neuer Seitenstrassen von der antiken römischen Strasse abzweigen, gibt es viele Abweichungen auf dem Weg vom Gen zum Protein. Zuerst schienen diese eher Ausnahmen des Dogmas zu sein. Inzwischen gelten sie als verbeitet und entscheidend in vielen biologischen Prozessen komplexer Organismen. Manche der Erkenntnisse sind so neu, dass sie noch nicht in Lehrbüchern vertreten und Lehrern vielleicht unbekannt sind. Dieser Bericht ist der erste einer Reihe für Science in School, die sich mit einem Teil dieser Forschung beschäftigt
Einige der spannendsten Erkenntnisse drehen sich um RNA, die einst nur als Zwischenstation bei der Proteinproduktion galt. Im Laufe der letzten Jahrzehnte aber entfaltete sie sich zu einem entscheidenden Kontrollfaktor der Geninformation einer Zelle. Das Verständnis der Verarbeitung von RNA führte zu Erkenntnissen über Krankheiten und leistungsstarken neuen Biotechnologien, wie den Einsatz von RNA in der Therapie gegen die Auswirkungen von Gendefekten.
Alternatives Splicing
Die im zentralen Dogma erwähnte RNA ist messenger RNA, die als Vorlage für die Proteinsynthese dient. In Eukaryoten müssen RNAs meist prozessiert werden, um in der Botenform vorzuliegen. Neu gebildete RNAs kann man sich als Güterzüge mit mehreren Waggons vorstellen, deren Ladung für verschiedene Kunden sortiert werden muss. Leere Waggons werden abgekoppelt und diejenigen davor und dahinter werden verbunden. Die leeren Waggons der Zelle heißen Introns, lange Abschnitte von Genen, die keine Proteine verschlüsseln. Kodierende Sequenzen hingegen nennt man Exons. Oft enthält ein Exon die Anleitung für nur einen Baustein des Proteins. Doch auch diese finden nicht immer Verwendung. Ein Kunde braucht vielleicht nicht alle Waren des Zuges, also werden einige volle Waggons zusammen mit den leeren entfernt. Und eine Zelle braucht manchmal nicht alle Teile eines bestimmten Proteins, deshalb werden verschiedene Exons kombiniert und so unterschiedliche Formen gebildet.
Dieser Prozess wird alternatives Spleißen genannt und wurde 1977 gleichzeitig von Richard Roberts bei New England Biolabs, USA und Phillip Sharp am Massachusetss Institute of Technology, USA, entdeckt (Berget et al., 2000). Dieser Fund war so bedeutsam, dass er ihnen 1993w1 den Nobelpreis für Physiologie oder Medizin einbrachte. Damals dachte man, Spleißen wäre selten; Sharp schätzte den Anteil von alternativ gespleißten Genen am menschlichen Genom auf nur 5%. Die Vervollständigung der menschlichen Genomsequenz ergab aber, dass das durchschnittliche Gen 8,4 Introns enthält, die alle während des Vorgangs erntfernt werden müssen. Auch wenn manche RNAs immer auf die gleiche Art und Weise prozessiert werden, durchlaufen geschätzte 75% alternatives Spleißen.

Abbildung freundlicherweise zur Verfügung gestellt vom EMBL Photolab
Menschliche Gene enthalten wesentlich mehr Introns als die der meisten anderern Organismen; der Durchschnitt liegt bei Fliegen und andere Insekten zwischen 2,4 und 5,4 Introns pro Gen (Auch wenn der momentane Weltrekordhalter im alternativen Spleißen ein Fruchtfliegen-Gen namens dscam ist, aus dem 38016 verschiedene Proteine entstehen können). Manche Wissenschaftler glauben, eine hohe Anzahl an Introns bedeutet, dass die Gene mit der Zeit immer komplexer geworden sind. Allerdings zeigt eine Studie der Gruppen von Detlev Ahrend und Peer Bork am Europäischen Laboratorium für Molekularbiologie (EMBL) in Heidelberg, dass der Vorfahre von Insekten und Wirbeltieren mit großer Sicherheit Gene mit mehr Introns, ähnlich denen des Menschen, besaß (Raible et al., 2005). Sie sind im Laufe der Evolution in Fliegen und anderen schnell reproduzierenden Spezies also stromlinienförmiger geworden.
Dazu gibt es interessante Unterschiede zwischen Arten hinsichtlich der Länge der Introns eines Gens, verglichen mit der der Exons. In den Genen des Wurms Caenorhabditis elegans und denen vieler anderer Arten umspannen Introns und Exons ungefähr die selbe Anzahl an “Buchstaben” (Nukleotiden). In Menschen ist das ganz anders: Introns in einem einzelnen Gen bestehen oft aus zehntausenden von Nukleotiden und sind im Mittel fünf mal so lang wie die Exons. Dies könnte Auswirkungen auf die Evolution menschlicher Gene gehabt haben, wie in einer Studie von Christian Castillo-Davis von der Harvard University, USA und Eugene Koonin und Fyodor Kondrashov vom National Center for Biotechnology Information, USA gezeigt wurde (Castillo-Davis et al., 2002). Die Transkription von RNA ist zeit- und energieaufwändig: die RNA eines einzelnen Gens mit riesigen Introns herzustellen kann mehrere Minuten dauern und tausende Moleküle an ATP in Anspruch nehmen. Die Autoren fanden heraus, dass die Introns häufig benutzter Gene durchschnittlich 14mal kürzer sind, als die von selten verwendeten. Ihre Schlussfolgerung: die natürliche Selektion hat Introns in den gebräuchlichsten Genen verkürzt, um Zeit und Energie zu sparen.
Sobald das alternative Spleißen entwickelt war, konnte es in der Zelle für viele Zwecke eingesetzt werden. Die unterschiedliche Kombination von Bausteinen führt zu Proteinen mit anderen Eigenschaften. Diese können verschiedene Zelltypen bilden und die Entwicklung verschiedener Gewebe steuern. Alternatives Spleißen einer RNA namens Slo im Hühnerohr verbessert das Gehör des Vogels durch erhöhte Sensitivität bezüglich einiger Schallfrequenzen. In Fliegen werden wichtige Proteine in Männchen und Weibchen unterschiedlich gespleißt, und so entstehen erhebliche Unterschiede zwischen beiden Geschlechtern. Obwohl Weibchen zwei X-Chromosomen besitzen und Männchen nur eines, bilden Weibchen aufgrund der Unterschiede der dort verschlüsselten Gene nicht auch die doppelte Proteinmenge. Diane Lipscombe und ihre Kollegen an der Brown University (Rhode Island, USA) haben herausgefunden, dass alternatives Spleißen im Hirn von Mäusen und anderen Säugetieren besonders weit verbreitet ist (Lipscombe, 2005). Manche der Moleküle haben mit Erinnerung und Lernen zu tun.
Das Spleißen spielt auch bei einigen Krankheiten eine Rolle. Die Hälfte der Neurofibromatose-Patienten – einer schweren genetischen Erkrankung, bei der sich Tumoren an Nerven und anderen Geweben bilden – weisen Mutationen auf, die das RNA-Spleißen des Neurofibromin-Gens ändern. Patienten mit Betathalassämie leiden an Anämie aufgrund eines nicht funktionsfähigen beta-Globin-Proteins der roten Blutkörperchen; diese ernsthafte Erkrankung wird ebenfalls durch falsches Spleißen des zugehörigen Gens hervorgerufen. Ein weiteres Beispiel sind Veränderungen im BRCA1-Gen (assoziiert mit Brustkrebs) und im CFTR-Gen (verursacht Cystische Fibrose). Geschätzte 50% der mit Krankheiten in Verbindung stehenden Mutationen in Exons haben mit der Art des Spleißens zu tun. Tumoren und neurodegenerative Erkrankungen werden oft von ungewöhnlich gespleißten RNAs begleitet, die sonst in gesundem Gewebe gefunden werden.
Qualitätskontrolle
1979 entdeckten Regine Losson und François Lacroute vom CRNS in Strasburg, Frankreich, dass die Zelle ein System besitzt, RNAs zu untersuchen und zu überprüfen (Losson & Lacroute, 1979). Fast drei Jahrzehnte Forschung haben ergeben, dass das System nicht perfekt ist, es aber doch schafft, eukaryotische Zellen vor gefährlichen Auswirkungen der meisten Mutationen zu schützen. Defekte in Genen die die Form, Struktur oder Funktion eines Proteins verändern, schaden der Zelle meistens. Auch Spleißfehler können solche Moleküle hervorbringen, also muss das Inspektionssystem nonsense-mediated mRNA decay (NMD) immer auf der Hut sein.
NMD findet statt, wenn eine RNA in das Cytoplasma einer Zelle eintritt. Forscher assoziierten es aber mit dem Spleißen, welches stattfindet, bevor die RNA den Kern verlässt (Sun et al., 2000). Die Zelle hängt ein Cluster von Proteinen an die Stellen, wo Introns entfernt wurden – so als ob Schilder an einen Waggon angebracht würden, um das Fehlen nachfolgender anzuzeigen. Das Cluster besteht aus mindestens sechs Proteinen und heißt exon junction complex (EJC). Dieser Komplex befindet sich an der Spleißstelle und seine Position hat großen Einfluss auf das Schicksal der RNA.
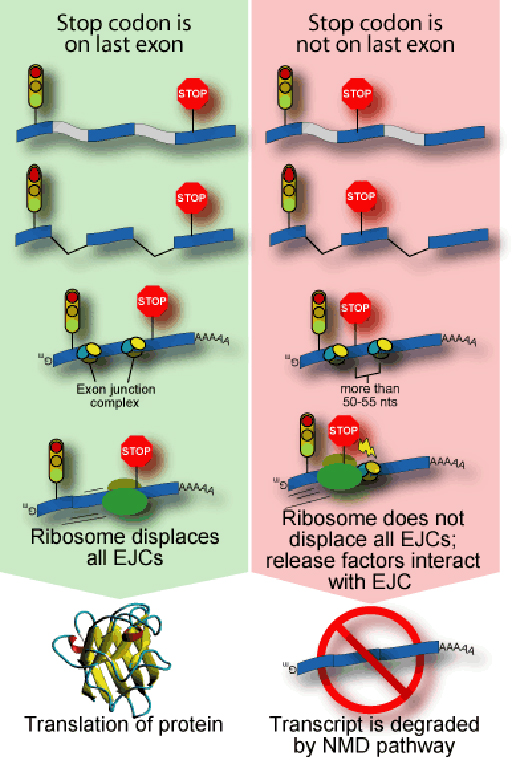
Die RNA-Translation zu einem Protein wird vom Ribosom ausgeführt, einer molekularen Maschinerie die an eine mRNA andockt, ihren Code liest und eine Kette aus Aminosäuren aufbaut, die ihrer Sequenz entsprechen. Alle auf ihr befindlichen EJCs werden einfach aus dem Weg geräumt. Am Ende des proteincodierenden Abschnitts der RNA begegnet dem Ribosom ein drei Buchstaben langes Signal, welches als Stopcodon bekannt ist, und sie gibt ihr fertiges Protein frei. Mutationen ändern oft den Leserahmen einer RNA, so dass ein Stopcodon irgendwo in der Mitte des Moleküls auftaucht. Dadurch entsteht ein Code, der für die Zelle keinen Sinn ergibt (der „Nonsens“ in NMD) und es kann ein EJC auf der RNA verbleiben, der NMD induziert (siehe Bild).
Um die Produktion von potentiell schädlichen Proteinen zu verhindern, erkennt NMD die EJCs, die sich mehr als 50 Nukleotide nach einem Stopcodon befinden. Der Translationsprozess wird dann unterbrochen, andere Moleküle schaffen die RNA weg und bauen sie ab.
Manche RNAs jedoch entkommen dem NMD und produzieren schädliche Proteine, die zu Krankheiten führen können. Auch wenn NMD funktioniert, kann am Ende eine Erkrankung entstehen, weil es vielleicht eine beschädigte aber notwendige RNA entfernt. 1989 zeigte Lynne Maquats Labor am Roswell Park Memorial Institute (New York, USA), dass NMD zur Betathalassämie beiträgt, der häufigsten genetischen Krankheit der westlichen Welt. Bei Betathalassämie ist die Hämoglobinproduktion im Körper reduziert. Dieses wird gebraucht, um Sauerstoff durch das Blut zu transportieren. Die Krankheit tritt bei Menschen auf, die eine mutierte Form des beta-Globin-Gens vererbt bekommen haben; NMD fängt die Mutation ab und der Körper baut die wichtige beta-Globin RNA ab (Lim et al., 1989). In diesem Fall greift ein eigentlicher Sicherheitsmechanismus in Wirklichkeit den Körper an.
Bisher wurde NMD für nicht mehr als ein Mittel gehalten, fehlerhafte RNAs abzufangen. Heutzutage wird es als ein gebräuchliches Werkzeug der Zelle betrachtet, die Qualität und Quantität bestimmer Moleküle zu überwachen. Es wird gebraucht, weil der Prozess des alternativen Spleißens manchmal RNAs mit Nonsens-Codons produziert. Aus irgendeinem Grund entsteht beim Ausschneiden und Einfügen ein Stück sinnloser Code in der Mitte einer RNA. Im Jahre 2004 unternahmen R. Tyler Hillman, Richard Green und Steven Brenner der University of California, Berkeley, USA eine Computeranalyse, die zeigte, dass alternatives Spleißen in circa einem Drittel der Fälle ein Stopcodon weiter als 50 Nukleotide vor eine Spleißstelle plaziert. Dadurch wird NMD aktiviert und die RNA wird eliminiert, bevor sie in Proteine umgeschrieben werden kann (Hillman et al., 2004).
Im selben Jahr untersuchte die Gruppe von Harry Dietz an der John Hopkins University School of Medicine (Maryland, USA) dies in Säugerzellen. Sie schalteten die NMD-Maschinerie ab, indem sie ein Protein namens Upf1 entfernten, das für den Prozess essentiell ist. Dies änderte das Verhalten vieler Gene: etwa 10% der betrachteten Gene wurden produktiver, wahrscheinlich weil gespleißte Formen fortbestanden, die normalerweise vom NMD erfasst und zerstört worden wären (Mendell et al., 2004).
Das bedeutet, NMD ist wie alternatives Spleißen eine häufig verwendete Seitengasse auf dem Weg zwischen Genen und Proteinen. Es gibt noch viele mehr, von denen manche in der kommenden Ausgabe von Science in School behandelt werden. Falls die Spannung überhand nehmen sollte, hier ein kleines Rätsel zur Beschäftigung: Die Farbe von Petunien beruht auf einer einzigen Kpioe eines bestimmten Gens. Was passiert mit ihr, wenn eine zweite Kopie dieses Gens dazukommt?
References
- Berget SM, Moore C, Sharp PA (2000). Spliced segments at the 5’ terminus of adenovirus 2 late mRNA. Reviews in Medical Virology 10: 355-371
- Castillo-Davis CI, Mekhedov SL, Hartl DL, Koonin EV, Kondrashov FA (2002) Selection for short introns in highly expressed genes. Nature Genetics 31: 415-418. doi:10.1038/ng940
- Crick F (1970) Central Dogma of Molecular Biology. Nature 227: 561-563
- Hillman RT, Green RE, Brenner SE (2004) An unappreciated role for RNA surveillance. Genome Biology 5: R8. doi:10.1186/gb-2004-5-2-r8
- Lim S, Mullins JJ, Chen CM, Gross KW, Maquat LE (1989) Novel metabolism of several beta zero-thalassemic beta-globin mRNAs in the erythroid tissues of transgenic mice. The EMBO Journal 8(9): 2613-2619
- Lipscombe D (2005) Neuronal proteins custom designed by alternative splicing. Current Opinion in Neurobiology 15: 358-363. doi:10.1016/j.conb.2005.04.002
- Losson R, Lacroute F (1979) Interference of nonsense mutations with eukaryotic messenger RNA stability. Proceedings of the National Academy of Sciences USA 76: 5134-5137
- Mendell JT, Sharifi NA, Meyers JL, Martinez-Murillo F, Dietz HC (2004) Nonsense surveillance regulates expression of diverse classes of mammalian transcripts and mutes genomic noise. Nature Genetics 36: 1073-1078. doi:10.1038/ng1429
- Raible F et al. (2005) Vertebrate-type intron-rich genes in the marine annelid. Platynereis dumerilii. Science 310: 1325-1326. doi:10.1126/science.1119089
- Sun X, Moriarty PM, Maquat LE (2000) Nonsense-mediated decay of glutathione peroxidase 1 mRNA in the cytoplasm depends on intron position. EMBO Journal 19: 4734-4744. doi:10.1093/emboj/19.17.47
Web References
- w1 – Eine Kopie von Philip Sharps Nobelvortrag über alternatives Spleißen „Split Genes and RNA Splicing“ ist online verfügbar
Resources
-
Viele Publikationen von Crick sind frei verfügbar auf der Nature-Hompage.
Institutions
Review
Die Fortschritte der Biotechnologie der letzten 40 Jahre haben sich auf Gebiete wie Landwirtschaft, Ernährungswissenschaft und Medizin ausgewirkt. Unser Wissen vom zentralen Dogma der Molekularbiologie in nützliche Anwendungen umzuwandeln, erfordert – besonders bei der Erforschung von Krankheiten und medizinischen Therapien – ein besseres Verständnis von der Funktionsweise der Moleküle des Lebens.
Diese Serie wird Entwicklungen und neue Theorien vorstellen, die vielleicht für Lehrbücher zu neu sind. Auch wird sie das internationale und universelle Streben erläutern, mehr über die Wirkungsweise der Moleküle des Lebens zu erfahren.
Dieser Artikel eignet sich sehr gut für das Anlesen von Grundlagen und Teile davon könnten verwendet werden, um das Verständnis vom zentralen Dogma, den Eigenschaften von RNA oder Proteinsynthese zu erweitern. Besonders geeignet ist der Artikel für Überlegungen zur Verwendung von Biotechnologie, dem Stellenwert aktueller Forschung und der Anerkennung wissenschaftlichen Bestrebens.
Marie Walsh, Republik Irland





