Supporting materials
Impressão digital genética: uma história forense (Word)
Impressão digital genética: uma história forense (Pdf)
Download
Download this article as a PDF

Traduzido por António Daniel Barbosa. Nas séries televisivas, a impressão digital genética é habitualmente usada para identificar criminosos. Sara Müller e Heike Göllner-Heibült espreitam os bastidores.
A ideia de distingui os indivíduos através das suas características genéticas não é nova. Descoberto em 1900 por Karl Landsteinerw1, o tipo sanguíneo ABO foi o primeiro marcador genético a ser usado nas ciências forenses, posteriormente complementado pelo grupo sanguíneo MN (1927) e pelo fator Rhesus (1937).
Contudo, mesmo quando analisamos os três sistemas sanguíneos simultaneamente, cerca de uma em cada dez pessoas origina resultados idênticos; razão pela qual é possível realizar transfusões sanguíneas. Porém, para os objetivos das ciências forenses, esta é uma desvantagem: os resultados podem dizer que a amostra de sangue não provém do Suspeito X, mas não pode dizer com um grau de certeza aceitável que provém do suspeito Y.
Avanços foram realizados nos anos 70 e 80 com a análise de diferentes formas de enzimas (isoenzimas) nos glóbulos vermelhos e soro sanguíneo. A certeza de que uma amostra provém definitivamente do suspeito depende do número de proteínas analisadas (habitualmente quatro); esta segurança é designada por poder de discriminação. O poder de discriminação que estas técnicas combinadas ofereciam era de apenas 1:1000, melhor do que a capacidade de 1:10 da análise dos grupos sanguíneos, mas ainda não suficientemente satisfatório. Para obter uma capacidade superior, foi necessário examinar mais de perto a nossa composição genética.
O genoma humano consiste em 46 pares de cromossomas: 23 da nossa mãe, 23 do nosso pai. Desta forma, possuímos dois de cada cromossoma (exceto – no caso dos homens – os cromossomas sexuais) e assim duas cópias de cada gene.
O principal componente dos cromossomas é o ácido desoxirribonucleico (DNA), que contem a informação para produzir as proteínas das quais necessitamos para viver. Porém, dos nossos 3 mil milhões pares de bases (pb), apenas cerca de 4% codifica efetivamente para proteínas; o resto é habitualmente apenas “enchimento” que consiste de sequências repetitivas organizadas em agregados. Se se comparar o DNA de dois seres humanos, a maioria será idêntico, com a variabilidade a ser detetada principalmente nestas sequências repetitivas.
Pessoas diferentes podem ter números diferentes de repetições destas sequências: uma pessoa pode ter cinco repetições num locus (local) de DNA específico; outra pessoa pode ter sete. Utilizando amostras, por exemplo de sangue ou sémen, pode-se analisar as sequências repetitivas em diversos loci de DNA; chama-se a esta análise uma impressão digital genética. Tal como as impressões digitais, as impressões digitais genéticas podem ser usadas para distinguir indivíduos.
Apesar do termo “impressão digital genética” (ou perfil genético) ser habitualmente usado, nem todos estão cientes de que na verdade esta corresponde a duas técnicas muito diferentes, apenas uma das quais é frequentemente usada nas ciências forenses atualmente.

O primeiro método de impressão digital genética foi inventado em 1984 por Alec Jeffreysw2, que usou sequências repetitivas de DNA conhecidas por repetições em série de número variável (VNTRs; por exemplo a sequência D1S80, (AGGACCACCAGGAAGG)n). Estas sequências, 10-100 pb por repetição, podem ser investigadas usando enzimas de restrição, que funcionam como tesouras moleculares que cortam o DNA em sequências definidas (sequências de reconhecimento). Em todo o nosso genoma, uma sequência de reconhecimento de 6 pb surgirá cerca de 730 000 vezes.
Isto significa que se se cortar o nosso genoma com uma enzima de restrição específica, obter-se-á cerca de 730 000 fragmentos de restrição de comprimentos variáveis. E é aqui que as VNTRs se tornam importantes: um número de repetições num agregado VNTR particular pode variar entre indivíduos, o que significa que o comprimento do fragmento de restrição correspondente irá variar também entre os indivíduos (Figura 1). Este fenómeno é designado por polimorfismos de comprimento de fragmentos de restrição (RFLP).
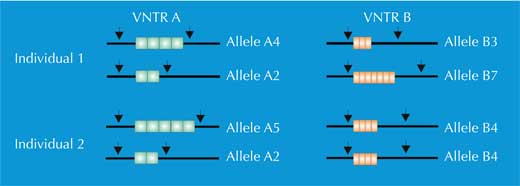
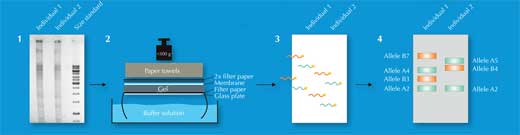
Dos 730 000 fragmentos de restrição, apenas alguns irão diferir entre os indivíduos – muito poucos para serem detetados visualmente. Por isso, os cientistas usam uma técnica chamada Southern blotting que permite que apenas as sequências de interesse sejam visualizadas. Para tal, os fragmentos de restrição são separados de acordo com o seu tamanho por electroforese em gel, usando uma corrente elétrica para mover as moléculas de DNA carregadas através do gel. A distância percorrida é determinada pelo tamanho do fragmento (Figura 2, Passo 1). De seguida, o DNA é transferido para uma membrana (Figura 2, Passo 2) e uma sonda radioativa complementar ao(s) VNTR(s) de interesse é adicionada. A sonda hibridiza (liga-se) às sequências complementares (Figura 2, Passo 3) e colocando a membrana num filme de raio-X os cientistas obtêm uma imagem das bandas marcadas radioactivamente, cada qual representando um fragmento de comprimento diferente (Figura 2, Passo 4). Esta imagem é a impressão digital genética.
Então, quantos VNTRs é necessário comparar para distinguir os indivíduos com segurança? Se os cientistas escolherem VNTRs com uma variabilidade suficiente (por exemplo D1S80, que pode ser repetido entre 15 a 41 vezes), apenas necessitam de comparar quatro VNTRs diferentes para obterem um poder de discriminação de 1:1 milhão – muito melhor do que o 1:10 oferecido pelo tipo sanguíneo ABO.
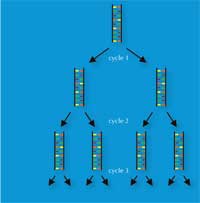
A invenção de Kary Mullis, em 1983, da reação em cadeia da polimerase (PCR) valeu-lhe o prémio Nobel da Químicaw3, w4. Esta invenção, juntamente com a descoberta nos finais da década de 80 de repetições pequenas em série (STRs) – sequências repetitivas de 2-9 pb, também designadas por microssatélites – abriu caminho para a técnica de impressão digital genética de alta velocidade que as ciências forenses utilizam atualmente.
Por PCR é possível amplificar exponencialmente um locus de DNA de interesse (por exemplo o STR de 4 pb conhecido como D18S51, (AGAA)n), produzindo milhões de cópias de uma única molécula de DNA em poucas horas (Figura 3). Para os cientistas forenses, esta técnica tem a vantagem de permitir a análise de amostras muito pequenas – bastam 30 células (ver Tabela 1 para uma comparação com a impressão digital genética por RFLP).
Na análise por PCR, precisamos de STRs flanqueadas por sequências que são idênticas em todos os seres humanos (estas sequências designam-se por conservadas). São então usados primers – pequenas moléculas que são complementares às sequências conservadas flanqueantes (genes 1134 e 1135 na Figura 4) – para iniciar o PCR. Uma vez amplificado o DNA, este pode ser separado quer por eletroforese em gel (Figura 5) ou, nas ciências forenses modernas, por sequenciação eletroforética automatizada (Figura 6), e ser visualizado como uma impressão digital genética.

Possuímos duas cópias de cada cromossoma, logo também temos duas cópias de cada STR. Se, para cada cópia do STR, alguém tem o mesmo número de repetições (isto é, o mesmo alelo), a análise de PCR mostra apenas um fragmento de DNA de um único tamanho: a pessoa é homozigótica para esse alelo do STR (seta verde na Figura 5, correspondente ao indivíduo 2 na Figura 4). Se os dois cromossomas possuem alelos não idênticos para esse STR, fragmentos com dois tamanhos distintos são observados e diz-se que a pessoa é heterozigótica (seta vermelha na Figura 5, correspondente ao indivíduo 1 na Figura 4).
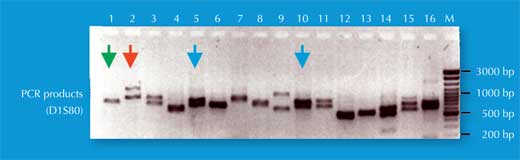
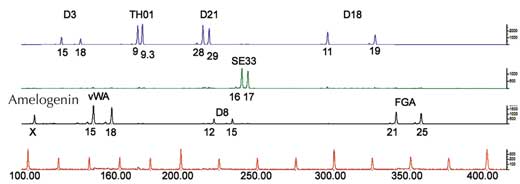
Se analisarmos apenas um STR, a probabilidade de duas pessoas sem parentesco terem a mesma impressão digital genética por PCR é elevada – entre 1:2 e 1:100 (setas azuis na Figura 5). Tal acontece porque os STRs têm menos alelos e menor heterozigosidade do que os VNTRs usados nas impressões digitais genéticas por RFLP. Para ultrapassar esta desvantagem, múltiplos STRs são analisados simultaneamente; com 16 STRs, como é comum nos casos tratados na Alemanha, é possível obter um poder de discriminação de 1:10 mil milhões (equivalente a uma pessoa na população mundial; Figura 6).
| RFLP | PCR | |
|---|---|---|
|
Quantidade de DNA inicial |
30–50 µg |
Pelo menos 200 pg (cerca de 30 células) para um padrão completo de STR |
|
Sensibilidade |
+ |
+++ |
|
Qualidade do DNA requerido para a análise |
Genoma completo |
Não é necessário o genoma completo; produtos de degradação são suficientes por implicar pequenas sequências (o comprimento total da sequência de um STR, incluindo repetições múltiplas e sequências flanqueantes, é aproximadamente de 50–500 pb) |
|
Tempo |
Dias a semanas |
Horas |
|
Poder de disciminação por locus |
+++ |
+ |
|
Unidade de repetição |
10 a 100 pb |
2 a 9 pb (nos casos forenses, maioritariamente 4 pb) |
|
Deteção automática |
Não é possível |
Possibilidade de processamento da amostra em larga escala |
|
Número de loci validados (importantes se familiares estiverem envolvidos) |
Número limitado |
Grande número |
|
Risco de contaminação |
+ |
+++ |
|
Medidas adicionais de segurança necessárias? |
Sim (devido à marcação radioativa) |
Não (não possui marcação radioativa) |

Sabe-se atualmente o que é a impressão digital genética, mas como usa-la? Impressões genéticas por PCR são amplamente usadas nas investigações forenses: permite à polícia excluir ou identificar suspeitos com base em material genético como folículos de cabelo, pele, sémen, saliva ou sangue (ver a história que pode ser descarregada em baixow5). No entanto, por si só, uma impressão digital genética não é uma evidência suficiente para uma acusação, uma vez que parentes próximos podem possuir impressões genéticas muito semelhantes (e gémeos monozigóticos terão normalmente impressões genéticas idênticas).

E para complicar as investigações forenses, apesar da recomendação Europeia para analisar 16 STRs, cada país pode decidir quais STRs analisar, o que torna as comparações difíceis.
O método à base de PCR é também usado nos seres humanos em testes de paternidade, diagnóstico de diversas doenças genéticas (por exemplo doença de Huntingdon), identificação de vítimas de acidentes, na elaboração de árvores genealógicas, procurar pessoas desaparecidas e investigar figuras históricas (por exemplo o último czar da Rússia e sua família). Em outros organismos pode ser usado para fins de conservação (por exemplo analisar marfim confiscado), investigações sobre droga (por exemplo na análise de plantas de canábis apreendidas), para controlar a qualidade alimentar e da água (por exemplo identificando micróbios contaminantes), na medicina (por exemplo para detetar infeções virais como o VIH, hepatite ou grite) e em investigações de bioterrorismo (por exemplo na identificação de estipes microbianas).
A impressão digital genética à base de RFLP, apesar de bastante obsoleta devido às imensas vantagens do método baseado em PCR (Tabela 1), é ainda usada na classificação de plantas e animais em investigações fundamentais.
O isolamento de DNA nas escolas dá aos alunos um momento “wow” quando se apercebem que estão a olhar para a informação genética completa que codifica para um organismo – umas poucas cadeias de DNA em forma de novelo de lã que são precipitadas com álcool. É fácil de realizar na escola usando salivaw6 (ou células epiteliais obtidas de kits comerciais), ervilhas (Madden, 2006), tomates, cebolasw7 ou timo de vitelo (verifique as restrições locais para a utilização de timo de vitelo nas escolas)w8.

Uma análise subsequente de um STR específico no DNA humano, por exemplo D1S80 ou TH01, pode ser realizada na escolaw6, utilizando kits comerciais disponíveis a um preço razoávelw9 se necessário. Se a sua escola não tiver acesso a um termociclador, o ciclo térmico pode ser realizado em três banhos de água, apesar de ser enfadonho e trabalhoso.
Se este equipamento não estiver disponível, existem kits que simulam e simplificam todo o processo de impressão digital genéticaw10. Estes kits contêm fragmentos de DNA que simulam a amplificação de diferentes alelos de um STR ou VNTR único. (De facto, são fragmentos de DNA por restrição de um plasmídeo ou de DNA de fago lamba.) O DNA requere eletroforese e subsequente coloração de forma a que os alunos sejam capazes de comparar as sequências de DNA “amplificadas” de uma amostra de uma prova com amostras de diversos suspeitos. É claro que isto é muito diferente de detetar STRs amplificados usando sequenciação eletroforética automatizada (e nem sequer representa de uma forma precisa a visualização de VNTRs usando Southern blotting, uma vez que o DNA é corado diretamente no gel), no entanto demonstra os princípios do processo analítico. Ao usar estes kits de simulação, os alunos devem ser alertados para o facto de a experiência dar a ideia que diferenças entre os indivíduos podem serem facilmente identificadas, o que não é o caso.
Os autores gostariam de agradecer a Wolfgang Nellen pelas suas ideias sobre o artigo e por permitir que as instruções da Science Bridge fossem disponibilizadas gratuitamente.
Estão também gratos a Shelley Goodman pelos seus conselhos sobre o uso de kits comerciais nas escolas.
Numa entrevista à Science in School, Alec Jeffreys fala sobre a sua pesquisa:
Hodge R, Wegener, A-L (2006) Alec Jeffreys interview: a pioneer on the frontier of human diversity. Science in School 3: 16-19. www.scienceinschool.org/2006/issue3/jeffreys
Ser associado da Science Bridge é normalmente necessário para aceder a estas instruções, mas os leitores deste artigo podem requere-las gratuitamente através do correio eletrónio sara.mueller@sciencebridge.net
Para saber como isolar DNA a partir de tomates (instruções em inglês), consultar: http://ucbiotech.org/edu/edu_aids/TomatoDNA.html
Klug WS, et al. (2008) Concepts of Genetics 9th edition. San Francisco, CA, USA: Pearson. ISBN: 9780321524041
Goodwin W, Linacre A, Hadi S (2010) An Introduction to Forensic Genetics. Chichester, UK: Wiley-Blackwell. ISBN: 978-0470710197
Wallace-Müller K (2011) O jogo detective de DNA. Science in School 19: 30-35. www.scienceinschool.org/2011/issue19/detective/portuguese
Patterson L (2009) Getting a grip on genetic diseases. Science in School 13: 53-58. www.scienceinschool.org/2009/issue13/insight
A ideia de usar o DNA para identificar um indivíduo é alucinante. A impressão digital genética de um indivíduo assenta nas sequências não codificantes, mas como são estas impressões genéticas criadas? Este artigo explica.
A capacidade de identificar criminosos a partir de bases de dados levanta questões importantes: será ético manter DNA humano em tais bases de dados ou será um violação dos direitos humanos? Deverá existir uma impressão digital genética de cada pessoa ou só dos que forem detidos? Durante quanto tempo deve ser mantido o perfil de DNA?
Os alunos poderão querer discutir como a impressão digital genética pode ajudar a diagnosticar doenças genéticas, bem como as suas aplicações na luta contra a caça furtiva e a extinção de espécies. Idealmente deverão ser capazes de testar a técnica por eles mesmos, quer numa experiência real ou simulada.
Shelley Goodman, Reino Unido