Sobre estradas romanas, parques de comboios e inspectores: descobertas recentes na investigação do RNA Understand article
Traduzido por Ana Luísa Carvalho. O RNA é uma molécula biológica de importância crucial, que raramente é mencionada em pormenor nos livros de texto. Neste primeiro de uma série de artigos, Russ Hodge descreve alguma da mais recente e excitante investigação sobre o RNA.

Florian Raible em busca das
espécies de evolução mais
rápida ou mais lenta
Imagem cortesia do Photolab
do EMBL
Imagine um romano dos tempos antigos, no alto de uma colina, observando a paisagem e planeando o percurso de uma nova estrada. Dois mil anos depois, as suas estradas são ainda visíveis, atravessando cidades e campos europeus, rectas como se desenhadas com uma régua, desviando-se apenas face a obstáculos mais persistentes. Nos anos 50 e 60, os cientistas mapearam um tipo semelhante de rotas em biologia ao descreverem as ligações entre diferentes tipos de moléculas na célula.
O seu trabalho é conhecido como o dogma central da biologia molecular: “O DNA faz RNA que faz as proteínas,” tal como James Watson escreveu nas suas notas quando ele e Francis Crick iniciaram a sua incursão na estrutura do DNA. Com este dogma, os dois cientistas proposeram uma nova relação entre os principais tipos de moléculas da célula – um fluxo de informação, num só sentido, dos ácidos nucleicos para as proteínas. Pouco depois da sua descoberta da dupla hélice, Crick apresentou este dogma como o grande projecto de investigação para a biologia molecular (Crick, 1970): os cientistas deveriam dedicar-se à descoberta dos mecanismos celulares responsáveis pelas transformações.
Nos anos 70, já eram conhecidos os fenómenos básicos da criação de RNA e proteínas, mas tornava-se óbvio que um mecanismo simples e unidireccional não era suficiente para explicar a química da célula. Tal como existem muitas estradas que ramificam a partir das antigas vias romanas, também foram encontrados mecanismos alternativos no trajecto entre genes e proteínas. No início, estas pareciam ser excepções raras do dogma central; entretanto, sabe-se que são fenómenos bastante comuns e cruciais em quase todos os processos biológicos dos organismos complexos. Algumas das descobertas são tão recentes que ainda não foram incluídas nos livros de texto e são desconhecidas por alguns professores. Este é o primeiro artigo de uma série da Science in School que irá apresentar alguma desta investigação recente.
Algumas das descobertas mais excitantes envolvem o RNA, considerado, em tempos, como apenas um meio para atingir o objectivo que é a síntese de proteínas. Na última década, estas moléculas começaram a ser encaradas como cruciais para o controlo celular da informação contida no genoma. A compreensão de como o RNA é utilizado pela célula levou à elucidação dos mecanismos de algumas doenças e a uma evolução extraordinária da biotecnologia, incluindo a tentativa de utilização de RNA em terapias que compensam os efeitos de genes defeituosos.
Splicing alternativo
O RNA a que se refere o dogma central é o RNA mensageiro, que serve de modelo para a síntese de proteínas. Nos eucariotas, os RNAs são sujeitos a processamento antes de terem a forma de mensageiro. Assim que são produzidos, os RNAs assemelham-se a longos comboios com muitas carruagens (vagões de carga), cuja carga tem que ser agrupada por forma a satisfazer as exigências dos diferentes clientes. As carruagens vazias são eliminadas, juntando-se a carruagem da frente com a carruagem de trás. Os equivalentes celulares destas carruagens vazias, são designados intrões, longas regiões dos genes que não codificam para qualquer proteína. As sequências de DNA que codificam para proteínas são designadas exões. Quase sempre, um exão contém a receita para um módulo de proteína. Mas, mesmo estes, nem sempre são utilizados. Um cliente pode não estar interessado em todas as carruagens de um comboio, pelo que alguns vagões contendo carga podem ser removidos juntamente com os vazios. E a célula pode não necessitar de todos os módulos de uma determinada proteína, pelo que vários exões podem ser combinados para produzirem formas diferentes da proteína.
Este processo, designado splicing alternativo, foi descoberto independentemente em 1977 por Richard Roberts dos New England Biolabs, nos EUA, e Phillip Sharp do Massachusetts Institute of Technology (Instituto de Tecnologia do Massachusetts, MIT), EUA (Berget et al., 2000). A descoberta foi tão importante que lhes valeu o Prémio Nobel em Fisiologia ou Medicina em 1993w1. Na altura, pensava-se que o splicing era um fenómeno raro; Sharp estimou que o processo de splicing alternativo ocorreria em apenas cerca de 5% dos genes humanos. A sequência completa do genoma humano revelou que, em média, um gene contém 8,4 intrões, sendo todos removidos durante o processo de splicing. Embora alguns RNAs humanos sejam submetidos ao mesmo processo de splicing, os cientistas calculam que cerca de 75% dos genes passa por splicing alternativo.

Imagem cortesia do Photolab do EMBL
Os genes humanos possuem um número de intrões consideravelmente superior à maioria dos outros organismos; moscas e outros insectos possuem cerca de 2,4 e 5,4 intrões por gene. (Mesmo assim, o actual recordista mundial do splicing alternativo é um gene da mosca-da-fruta designado dscam, que pode originar 38.016 proteínas distintas.) Alguns investigadores acreditavam que o elevado número de intrões humanos indicava um aumento da complexidade dos genes com o tempo. No entanto, um estudo recente dos grupos de investigação de Detlev Arendt e Peer Bork do Laboratório Europeu de Biologia Molecular (EMBL, European Molecular Biology Laboratory) em Heidelberg, na Alemanha, mostra que um ancestral comum de insectos e vertebrados possuía seguramente genes com muitos intrões, à semelhança dos genes humanos (Raible et al., 2005). Ao longo da evolução, os genes tornaram-se mais resistentes a alterações, nas moscas e outras espécies de reprodução rápida.
Também existem diferenças interessantes entre espécies no que respeita ao comprimento total dos intrões comparados com os exões. Nos genes do verme Caenorhabditis elegans e muitas outras espécies, os intrões e os exões contêm cerca do mesmo número de ‘letras’ (nucleótidos). O que ocorre nos humanos é muito diferente: os intrões de um só gene contêm frequentemente dezenas de milhares de nucleótidos e possuem, em média, cinco vezes o comprimento dos exões. Isto pode exercer algum impacto na evolução dos genes humanos, tal como revelado num estudo de Cristian Castillo-Davis da Universidade de Harvard, nos EUA, e Eugene Koonin e Fyodor Kondrashov do Centro Nacional para a Informação em Biotecnologia (National Center for Biotechnology Information), nos EUA (Castillo-Davis et al., 2002). A transcrição do RNA é um processo lento e consumidor de energia: produzir RNA a partir de um só gene contendo intrões enormes pode exigir vários minutos e milhares de moléculas de ATP. Os autores descobriram que os intrões de genes frequentemente usados, são em média 14 vezes mais curtos que os intrões dos genes que são raramente usados. Conclusão: a natureza tem vindo a encurtar os intrões dos genes mais comuns, poupando tempo e energia.
Com a evolução da maquinaria celular necessária para o splicing alternativo, este pôde ser usado de muitas formas diferentes. Misturando e correspondendo módulos produz proteínas com comportamentos diferentes. Estas ajudam a criar diferentes tipos de células e influenciam o desenvolvimento de diferentes tecidos. O splicing alternativo de um RNA designado Slo no ouvido da galinha, melhora a audição da ave, conferindo às células sensibilidade a diferentes frequências de som. Nas moscas, três proteínas importantes sofrem splicings diferentes dependendo do organismo ser fêmea ou macho, originando diferenças importantes entre ambos os sexos. Embora as fêmeas possuam dois cromossomas X e os machos apenas um, as fêmeas não produzem o dobro da quantidade de proteína dos genes daquele cromossoma, e isto deve-se às diferenças entre estas proteínas. Diane Lipscombe e colegas da Universidade Brown (Rhode Island, EUA) descobriram que o splicing alternativo é significativamente comum no cérebro de ratinhos e outros mamíferos (Lipscombe, 2005). Algumas das moléculas são essenciais para os processos de memória e aprendizagem.
O fenómeno de splicing é determinante numa variedade de doenças. Metade das pessoas que sofrem de neurofibromatose, uma doença genética grave na qual vários tumores acompanham o desenvolvimento dos nervos e outros tecidos, apresentam mutações que alteram o splicing dos RNAs produzidos pelo gene neurofibromin. Os pacientes com beta-talassémia sofrem de anemia resultante da presença, nos glóbulos vermelhos, de proteínas beta-globinas não funcionais; esta doença grave é causada por erros de splicing do gene responsável. Outros exemplos são alterações do gene BRCA1 (associado ao cancro mamário) e do gene CFTR (responsável pela fibrose cística). Estima-se que cerca de 50 % das mutações nos exões, associadas a doença, afectem a forma como se processa o mecanismo de splicing. Os tumores e as doenças neurodegenerativas estão muitas vezes associados a splicing invulgar dos RNAs, que não se verifica nos tecidos saudáveis.
Controlo de qualidade
Em 1979, Regine Losson e François Lacroute do CNRS em Estrasburgo, França, descobriram que a célula possui um sistema de inspecção e controlo da qualidade dos RNAs (Losson & Lacroute, 1979). Quase três décadas de investigação demonstraram que o sistema não é perfeito, mas consegue proteger as células eucariotas dos efeitos perigosos da maioria das mutações. Os defeitos genéticos que alteram a forma, estrutura ou funções de uma proteína, são habitualmente prejudiciais à célula. Erros de splicing podem produzir esse tipo de moléculas, logo o sistema de inspecção – designado nonsense-mediated mRNA decay (NMD) – tem que estar permanentemente alerta.
O NMD ocorre quando o RNA entra no citoplasma da célula, mas os cientistas associam-no ao fenómeno de splicing, que ocorre antes de o RNA sair do núcleo (Sun et al., 2000). A célula agrega várias proteínas nos locais onde ocorreu remoção de intrões, como se sinalizasse uma composição onde várias carruagens estão ausentes. Este agregado consiste de pelo menos seis proteínas e é designado complexo de junção de exões (exon junction complex, EJC). Este complexo forma-se nos locais de splicing e a sua localização tem grande importância sobre o destino do RNA.
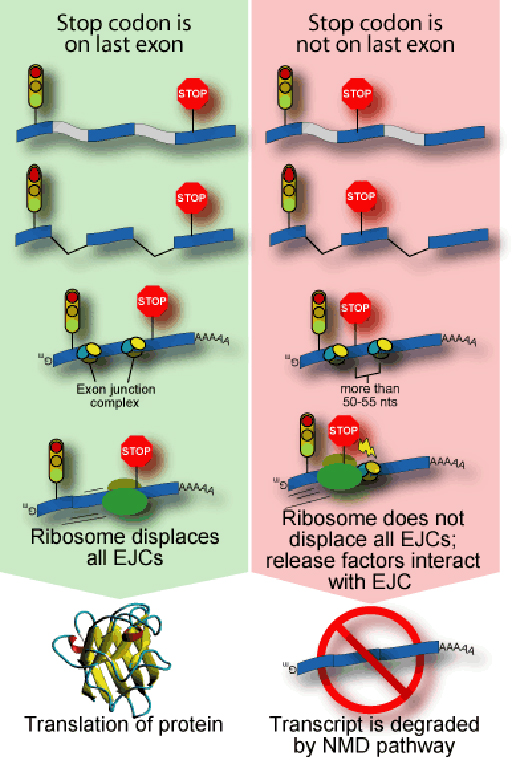
A tradução de um RNA em proteína ocorre numa máquina molecular designada ribossoma que se liga ao RNA mensageiro, lê o seu código e constrói uma cadeia de amino-ácidos correspondente à sequência. Qualquer complexo EJC associado ao RNA é simplesmente desviado do caminho. Chegando ao fim da sequência codificante do RNA, o ribossoma encontra um sinal, um codão stop, de três letras e liberta a proteína acabada de sintetizar. As mutações alteram frequentemente a sequência do RNA, de tal forma que um codão stop pode surgir a meio da molécula. Isto origina um código que não faz sentido para a célula, marcando o RNA com um EJC, promovendo a acção do NMD (ver figura).
Para prevenir a produção de proteínas potencialmente perigosas, o NMD reconhece EJCs que podem ser encontrados a mais de 50 nucleótidos a jusante de um codão stop. O processo de tradução é interrompido e outras moléculas surgem que levam o RNA para degradação.
No entanto, alguns RNAs escapam ao NMD e produzem proteínas prejudiciais que provocam doenças. Mesmo quando o NMD funciona, o resultado pode ainda ser uma doença, pois o NMD pode remover um RNA defeituoso, mas mesmo assim essencial. Em 1989, o grupo de investigação de Lynne Maquat no Instituto Roswell Park Memorial (Nova Iorque, EUA) mostrou que o NMD contribui para a beta-talassémia, a doença genética mais comum no mundo ocidental. A beta-talassémia reduz a produção de hemoglobina, essencial para o transporte de oxigénio no sangue. A doença surge nas pessoas que herdam uma forma mutante do gene da beta-globina; o NMD identifica a mutação e o organismo degrada o RNA beta-globina – removendo uma molécula essencial (Lim et al., 1989). Neste caso, um mecanismo de defesa intencional prejudica o organismo.
Até há pouco tempo, o NMD foi considerado como sendo pouco mais do que uma forma de localizar RNAs com erros; hoje, sabe-se que é uma ferramenta da célula para controlar a quantidade e a qualidade de certas moléculas. Isto acontece porque o processo normal de splicing alternativo produz, por vezes, RNAs com codões sem sentido; por alguma razão, a operação de corte e colagem produz código sem sentido a meio de um RNA. Em 2004, R. Tyler Hillman, Richard Green e Steven Brenner da Universidade da Califórnia, Berkeley, EUA, efectuaram uma análise computacional que mostrou que, com uma frequência significativa (um terço do tempo), o splicing alternativo coloca um sinal de stop a mais de 50 nucleótidos de um local de splicing. Isto activa o NMD que elimina a maior parte do RNA antes de ser transformado em proteínas (Hillman et al., 2004).
No mesmo ano, o grupo de Harry Dietz do John Hopkins University School of Medicine (Maryland, EUA) estudaram este efeito nas células de mamíferos. Inactivaram a maquinaria do NMD por remoção de uma proteína designada Upf1, essencial para este processo. Isto alterou o comportamento de um enorme número de genes: cerca de 10% dos genes estudados tornaram-se mais produtivos, provavelmente porque as formas processadas escapavam ao NMD e não eram destruídas (Mendell et al., 2004).
Isto significa que o NMD, tal como o splicing alternativo, é um caminho alternativo à estrada que liga os genes às proteínas. Existem muitos mais, que discutiremos noutros números da Science in School. Se a curiosidade é muita, aqui fica um enigma: a cor púrpura das petúnias deve-se a uma única cópia de um determinado gene. O que acontece à cor das petúnias se adicionarmos à flor uma segunda cópia desse mesmo gene?
References
- Berget SM, Moore C, Sharp PA (2000). Spliced segments at the 5’ terminus of adenovirus 2 late mRNA. Reviews in Medical Virology 10: 355-371
- Castillo-Davis CI, Mekhedov SL, Hartl DL, Koonin EV, Kondrashov FA (2002) Selection for short introns in highly expressed genes. Nature Genetics 31: 415-418. doi:10.1038/ng940
- Crick F (1970) Central Dogma of Molecular Biology. Nature 227: 561-563
- Hillman RT, Green RE, Brenner SE (2004) An unappreciated role for RNA surveillance. Genome Biology 5: R8. doi:10.1186/gb-2004-5-2-r8
- Lim S, Mullins JJ, Chen CM, Gross KW, Maquat LE (1989) Novel metabolism of several beta zero-thalassemic beta-globin mRNAs in the erythroid tissues of transgenic mice. The EMBO Journal 8(9): 2613-2619
- Lipscombe D (2005) Neuronal proteins custom designed by alternative splicing. Current Opinion in Neurobiology 15: 358-363. doi:10.1016/j.conb.2005.04.002
- Losson R, Lacroute F (1979) Interference of nonsense mutations with eukaryotic messenger RNA stability. Proceedings of the National Academy of Sciences USA 76: 5134-5137
- Mendell JT, Sharifi NA, Meyers JL, Martinez-Murillo F, Dietz HC (2004) Nonsense surveillance regulates expression of diverse classes of mammalian transcripts and mutes genomic noise. Nature Genetics 36: 1073-1078. doi:10.1038/ng1429
- Raible F et al. (2005) Vertebrate-type intron-rich genes in the marine annelid. Platynereis dumerilii. Science 310: 1325-1326. doi:10.1126/science.1119089
- Sun X, Moriarty PM, Maquat LE (2000) Nonsense-mediated decay of glutathione peroxidase 1 mRNA in the cytoplasm depends on intron position. EMBO Journal 19: 4734-4744. doi:10.1093/emboj/19.17.473
Web References
- w1 – Uma cópia da conferência Nobel de Philip Sharp sobre splicing alternativo, ‘Split Genes and RNA Splicing’, encontra-se disponível online.
- Uma breve revisão sobre o trabalho de Richard Roberts e Philip Sharp encontra-se na comunicação à imprensa aquando da atribuição do Prémio Nabel.
- Para mais informação acerca do Prémio Nobel, incluindo biografias dos laureados, ver http://nobelprize.org/
Resources
- Muitos artigos de Crick estão disponíveis no sítio da revista Nature.
Institutions
Review
Os avanços na biotecnologia dos últimos 40 anos tiveram impacto em áreas como a agricultura, a ciência alimentar e a medicina. Canalizar o nosso conhecimento científico para aplicações de utilidade, especialmente na investigação de doenças e tratamentos médicos, requer um conhecimento detalhado das funções das moléculas da vida.
Esta série de artigos reunirá avanços e novas teorias que podem ser demasiado recentes para estarem presentes nos livros de texto. Também ilustrarão o esforço internacional e universal de aprendizagem de como funcionam as moléculas da vida.
Este artigo providencia uma leitura de apoio excelente e partes dele podem ser empregues para aumentar a compreensão dos estudantes sobre o dogma central, as propriedades do RNA ou a síntese proteica. Mais especificamente, este artigo pode ser aplicado nas considerações sobre o uso da biotecnologia, a importância da investigação actual e no reconhecimento do esforço científico.
Marie Walsh, República da Irlanda





