Cristaux biologiques : à l’interface entre physique, chimie et biologie Understand article
Traduit par Dominique Cornuéjols. Dominique Cornuéjols, de l’Installation Européenne de Rayonnement Synchrotron (ESRF), nous dévoile le monde de la cristallographie.

autorisation de BlackJack3D/iStockphoto
« Cristal » n’est un mot qui vient naturellement à l’esprit lorsqu’on pense à la biologie. Les cristaux sont avant tout des magnifiques représentants du monde minéral. Les pierres précieuses, ces « étoiles qui éclairent le monde souterrain », nous ont fascinés depuis toujours, et la plus célèbre de toutes, le diamant, est devenu symbole à la fois de dureté et d’éternité.
Au contraire, la plupart des tissus biologiques sont mous, et chacun sait que la vie n’est pas éternelle. Cependant, il est possible d’isoler des molécules du vivant – des protéines par exemple – et d’en obtenir des cristaux en laboratoire. L’étude de biocristaux que l’on a fait croître artificiellement est à l’origine d’une discipline en plein développement, la cristallographie macromoléculaire.
Cristaux et structure
L’étude scientifique des cristaux minéraux – la cristallographie – a débuté à la fin du 17e siècle. Au début, cette science consistait essentiellement à décrire et à mesurer les faces et les angles des différentes structures cristallines afin de les classifier en fonction de leurs caractéristiques géométriques. Assez vite, certains cristallographes ont suggéré que la géométrie observée à l’échelle macroscopique pourrait s’expliquer par l’arrangement régulier de très petites particules (en fait des atomes, des molécules ou des ions), invisibles à l’œil nu et même au microscope. La théorie atomistique étant encore embryonnaire, ce modèle a été largement débattu tout au long des 18e et 19e siècles, sans que les scientifiques arrivent à une conclusion définitive.
Une série de percées remarquables dans le domaine de la physique ont révolutionné la façon dont nous voyons la matière aujourd’hui:
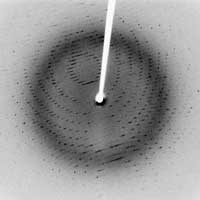
rayons X d’un cristal de protéase
3ClproSARS
Image reproduite avec l’aimable
autorisation de Jeff Dahl; source
de l’image: Wikimedia Commons
- En 1895, W.C. Röntgen découvre les rayons X. Ces rayons ont la possibilité extraordinaire de pénétrer dans les objets et les corps, révélant ainsi leurs caractéristiques internes. Les rayons X trouvent une application immédiate en imagerie médicale, mais leur nature (en tant qu’ondes électromagnétiques) est restée une énigme pendant 17 ans.
- En 1912, Laue découvre la diffraction des rayons X, en ayant l’idée d’illuminer un cristal par un faisceau de rayons X. Dans un cristal, les atomes (ou les molécules) sont positionnés selon un ordre rigoureux, parfaitement répétitif. Lorsque le cristal est exposé aux rayons X, chaque atome de la structure rediffuse une infime partie des rayons X. Du fait de l’arrangement régulier des atomes dans la structure, les rayons diffusés interfèrent de façon constructive dans certaines directions de l’espace : c’est la diffraction. Les faisceaux diffractés sont enregistrés sur un détecteur sous forme de points, et l’image qui en résulte est appelée un diagramme de diffraction. L’expérience de Laue a démontré deux choses : d’abord que les cristaux sont faits d’un arrangement régulier d’atomes, ensuite que les rayons X sont bien des ondes électromagnétiques, avec une longueur d’onde très courte, de l’ordre du dixième de nanomètre, c’est-à-dire la distance typique entre deux atomes
- En 1913, les Bragg (père et fils) établissent une relation entre la longueur d’onde (λ) des rayons X, la distance (d) entre deux plans d’atomes dans le cristal, et enfin l’angle d’incidence (θ ) des rayons X : 2 d sinθ = n λ, où n est un nombre entier. C’est la loi de Bragg (voir Hughes, 2007, pour une explication plus approfondie de la loi de Bragg). Cette importante découverte a marqué le début de la cristallographie par rayons X, c’est-à-dire la possibilité de déterminer la structure d’un cristal en l’exposant aux rayons X. La première structure « résolue » à l’échelle atomique a été celle du sel de cuisine, en 1914, suivie de près par celle du diamant. Encouragés par ces débuts prometteurs, les scientifiques ont alors utilisé la technique extrêmement puissante de la diffraction des rayons X pour comprendre et clarifier la structure atomique et moléculaire de toutes sortes de cristaux. « C’était comme la découverte d’un champ aurifère, où les pépites n’attendaient que d’être ramassées », comme le reconnurent les Bragg eux-mêmes.
Dans le même temps, les premiers cristaux biologiques avaient été obtenus en laboratoire, rendant donc possible l’étude de molécules biologiques par les rayons X. La première protéine à donner une image de diffraction, dès 1930, fut une enzyme, la pepsine. Peu de temps après, des scientifiques réussirent à isoler un virus, à le cristalliser et à montrer qu’il ne perdait rien de son activité biologique pendant ce processus : le virus de la mosaïque du tabac était toujours infectieux après cristallisation. La cristallographie macromoléculaire pouvait s’épanouir!
D’une façon un peu surprenante, pas un seul biologiste ne fut impliqué dans les débuts de cette recherche sur les structures moléculaires complexes. Elle fut entièrement menée par des chimistes, physiciens et cristallographes, reflétant ainsi le fait que, pendant la première moitié du 20e siècle, l’intérêt de nombreux scientifiques d’autres disciplines s’était porté sur la biologie. L’un des meilleurs exemples de cette tendance est le livre Qu’est-ce que la vie? (What is Life?), écrit en 1944 par Erwin Schrödinger, un physicien très connu dans le domaine de la mécanique quantique. La biologie moléculaire, qui est apparue dans les années 40 à la jonction de la biochimie et de la génétique, a été – depuis le début – très interdisciplinaire. Il est évident que cette discipline naissante a énormément bénéficié des nouveaux outils inventés par les physiciens. A un niveau plus conceptuel, l’idée que la vie puisse être expliquée par de simples mécanismes physico-chimiques a été très controversée, et beaucoup pensaient que la complexité du monde vivant ne pouvait se réduire à des interactions entre biomolécules. Aujourd’hui, la biologie moléculaire « structurale » est reconnue comme une des branches principales de la biologie et continue à se développer à un rythme soutenu.
Elle repose largement sur la cristallographie macromoléculaire. En effet, chaque macromolécule (protéine) possède son propre nuage électronique qui diffracte les rayons X. La forme et la composition atomique du nuage électronique déterminent la façon dont les rayons X sont diffractésw2 – c’est le signal de la molécule. Tous les petits signaux minuscules obtenus par chacune des molécules dans le cristal s’additionnent pour donner un signal mesurable.
L’image de diffraction finale, résultat des signaux pris à différents angles de rotation du cristal, est traitée mathématiquement (cette opération est appelée transformée de Fourier) pour obtenir la « carte de densité électronique » qui représente le nuage d’électrons de la protéine. Grâce aux logiciels de modélisation et aux techniques de raffinement, la séquence des acides aminés de la protéine est mise en correspondance avec le nuage d’électrons pour déterminer l’arrangement tridimensionnel des atomes de la protéine, ce qu’on appelle la structure.
Mais pourquoi la structure tridimensionnelle est-elle si importante pour l’étude des protéines et des autres molécules biologiques?
Structure et fonction
Nos mains et nos yeux, comme les autres caractéristiques anatomiques des plantes et des animaux, ont vu leur forme évoluer dans le temps pour répondre aux besoins de la vie. D’une façon similaire, la structure microscopique de chaque organelle subcellulaire ou macromolécule biologique est liée à sa fonction. Des molécules aux formes adaptées ont pour tâche d’activer ou de désactiver certains gènes, de catalyser les mécanismes chimiques complexes du vivant, de protéger les cellules contre les invasions ou encore de donner le signal pour initier la division cellulaire et en contrôler le développement.
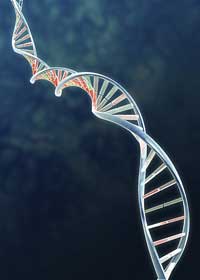
Image reproduite avec l’aimable
autorisation geopaul/ iStockphoto
La relation entre structure moléculaire et fonction est particulièrement évidente dans l’exemple de l’ADN. La structure en double hélice de l’ADN, avec ses paires de bases, est à la fois simple et belle. Elle rend aussi la génétique immédiatement compréhensible en termes chimiques. Les gènes, ces facteurs jusque là mystérieusement impliqués dans la transmission des traits génétiques, étaient en réalité des fragments de molécules d’ADN. Or on pouvait extraire l’ADN d’une solution en faisant tourner dans celle-ci l’extrémité d’une tige en verre (voir Madden, 2006, pour un protocole de purification d’ADN en classe), et étudier les fibres ainsi obtenues grâce à la diffraction des rayons X. La détermination de la remarquable – mais en même temps très simple – structure de l’ADN a marqué d’une pierre blanche la biologie structurale.
Par contraste, l’étude des structures de protéines n’a pas produit une explication limpide et universelle de la structure des protéines ni, par conséquent, de leurs fonctions. A ce jour, et bien que connaissant la structure d’environ 45000 protéines différentes, nous sommes encore incapables d’établir un ensemble de règles générales qui nous permettraient de prédire la structure tridimensionnelle d’une protéine à partir de la séquence des acides aminés de sa chaîne polypeptidique. Les protéines remplissent une gamme de fonctions biologiques infiniment plus large que ne le fait l’ADN, et la diversité fonctionnelle a dicté la diversité structurale.
Un obstacle majeur en génomique structurale : la cristallisation des protéines
Par comparaison avec la génétique moléculaire, les progrès en structures de protéines ont été extrêmement lents, en grande partie par la simple difficulté technique d’obtenir des cristaux de protéines de taille suffisamment grande pour être utilisés dans les analyses cristallographiques, et qui diffractent suffisamment bien pour permettre de déterminer la structure en haute résolution (résolution atomique). De plus, bien que ressemblant par certains côtés à d’autres cristaux, comme par exemple les cristaux de sel de cuisine, les cristaux de protéine sont beaucoup plus petits et généralement extrêmement fragiles.
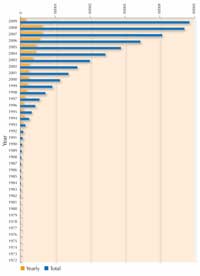
protéines résolues par rayons X.
Note : les structures répertoriées
varient avec le temps, car certaines
deviennent obsolètes et sont retirées
de la base de données. Cliquer sur
l’image pour agrandir
La cristallisation des protéines nécessite souvent des successions d’essais et d’erreurs, sans théorie prédictive. Certaines protéines cristallisent instantanément, d’autres refusent obstinément de produire des cristaux exploitables ; certains chercheurs semblent avoir la « main verte », comme de bons jardiniers, et peuvent obtenir des cristaux alors que d’autres échouent. De ce fait, la cristallisation des protéines a parfois été perçue comme un art plutôt que comme une science.
Pour chaque nouvelle protéine, les scientifiques doivent passer au crible un très grand nombre de paramètres afin de trouver les conditions particulières qui vont permettre la cristallisation. Ces paramètres sont, par exemple, la concentration en protéine, la température, le pH, ainsi que la concentration d’un vaste choix d’agents de précipitation pouvant être utilisés avec différents sels. Pour essayer vous-même de trouver les meilleures conditions pour cristalliser une protéine en classe, et avoir vos résultats analysés par diffraction des rayons X dans un vrai laboratoire, voir Blattmann & Sticher (2009) dans ce numéro. Parce que la cristallisation des protéines est tellement problématique, les protéines les plus étudiées jusqu’à maintenant ont été celles que l’on peut cristalliser facilement et qui peuvent être produites en grandes quantités, plutôt que celles ayant le plus grand intérêt biologique. Cependant, d’importants progrès ont été faits depuis une dizaine d’années, comme le montre le nombre croissant de structures de protéines résolues par cristallographie rayons X depuis 1983. Cette croissance spectaculaire est due à l’amélioration des techniques dans trois domaines : la préparation des cristaux, la cristallographie par rayons X et le développement logiciel.
Voyage d’une protéine d’un laboratoire à l’autre
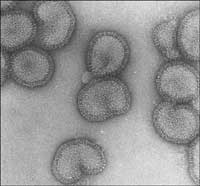
microscopie électronique
Image reproduite avec l’aimable
autorisation de Rob Ruigrok/UVHCI
our illustrer la façon dont la structure d’une protéine est résolue aujourd’hui en utilisant l’instrumentation la plus avancée, nous allons voir comment des scientifiques ont identifié la structure d’une des protéines clés du virus de la grippe, la polymérase. Une équipe de scientifiques du Laboratoire européen de biologie moléculaire (EMBL, antenne de Grenoblew3, France) et de l’Unité de l’interaction virus / cellule-hôtew4 ont étudié cette protéine impliquée dans le mécanisme utilisé par le virus pour prendre le contrôle de certaines fonctions essentielles des cellules humaines infectées (voir Ainsworth, 2009, dans ce numéro, pour les détails et les résultats de cette étude). Pour ce projet, les scientifiques ont bénéficié du Partenariat pour la biologie structuralew5 (PSB), une plateforme collaborative servant à résoudre des structures de molécules biologiques présentant un grand intérêt médical.
Clonage et expression (à l’UVHCI, EMBL et PSB)
Après que la protéine a été sélectionnée pour être étudiée, le gène correspondant a été amplifié, c’est-à-dire cloné dans un système particulier lui permettant d’être exprimé. Cette technique est excellente pour produire de grandes quantités de la protéine en utilisant un système hôte, généralement une bactérie.
Purification (au PSB) et contrôle de qualité (à l’IBS)
Les cellules bactériennes ont été ensuite « récoltées » par centrifugation, et les débris de cellules ainsi que des contaminants possibles (acides aminés par exemple) ont été retirés. La protéine a été ensuite soumise à un processus de purification en plusieurs étapes, lent mais essentiel, car 95% de pureté est souhaitable pour une bonne cristallisation. La qualité de la protéine a été vérifiée à l’Institut de biologie structurale Jean-Pierre Ebelw6 (IBS) par spectrométrie de masse (pour une courte introduction à la spectrométrie de masse, voir Wilson & Haslam, 2009, dans ce numéro), et un séquenceur a été utilisé pour s’assurer que la protéine purifiée était bien celle recherchée : la polymérase du virus de la grippe.
Cristallisation (au PSB)
Les scientifiques ont essayé de cristalliser la protéine par la technique de criblage multifactoriel. En d’autres termes, ils ont exposé différentes concentrations de la protéine à différents agents de cristallisation, tampons, températures, etc. Cette méthode (connue sous le nom de « microbatch », voir image ci-dessous) a été élaborée pour obtenir le maximum d’informations sur la protéine que l’on souhaite cristalliser, tout en dépensant une quantité minimale de solution de protéine.
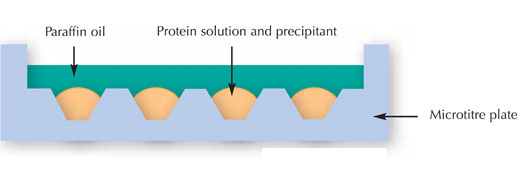
Image reproduite avec l’aimable autorisation de Nicola Graf
Diffraction des rayons X et collection de données sur une ligne de lumière synchrotron (à l’ESRF)

Image reproduite avec l’aimable
autorisation de l’ESRF

sur une ligne de lumière de l’ESRF
Image reproduite avec l’aimable
autorisation de l’ESRF
Après avoir obtenu des cristaux de polymérase, les scientifiques les ont cryogénisés à l’azote liquide et les ont ensuite transportés à l’Installation européenne de rayonnement synchrotron (ESRF)w1. Là, un cristal a été fixé sur une tête goniométrique – opération généralement robotisée – et exposé aux rayons X du synchrotron, qui ont pour particularité d’être extrêmement intenses. Un goniomètre est un instrument qui permet la rotation d’un objet (tel qu’un cristal) avec une grande précision angulaire.
La tête goniométrique fait tourner le cristal dans le faisceau de rayons X afin d’obtenir le maximum de « réflexions », ou faisceaux diffractés. Ayant ainsi obtenu d’énormes quantités de données en peu de temps, ce qui est l’avantage des sources de lumière synchrotron, les scientifiques ont pu résoudre la structure de la protéine de façon automatique, grâce à un ensemble d’outils logiciels développés spécialement dans ce but.
Modélisation, correspondance, raffinement et validation (à l’UVHCI et EMBL)
Basé sur les données expérimentales, un modèle atomique de la polymérase a été construit et comparé avec la carte de densité électronique. Le modèle a été ensuite raffiné itérativement afin de correspondre au mieux aux données observées, et ceci grâce aux représentations graphiques interactives et aux logiciels puissants mis aujourd’hui à la disposition des chercheurs. Après validation du modèle, la structure a finalement été publiée et déposée dans la Banque de données des protéinesw7 accessible publiquement.
L’ESRF : une des sources de rayons X les plus intenses au monde
L’ESRF est un bon exemple de grand équipement scientifique fonctionnant jour et nuit pour servir des milliers d’utilisateurs venant du monde entier. Un « utilisateur » est un scientifique, généralement faisant partie d’une équipe plus importante, ayant besoin occasionnellement d’un outil puissant pour obtenir des informations sur un échantillon de matière qu’il souhaite étudier (un morceau de matériau, un cristal de protéine, un fossile ou une réaction catalytique, par exemple). La plupart des utilisateurs font le voyage à Grenoble une ou deux fois par an pour enregistrer des données à l’ESRF.

rayonnement synchrotron
Image reproduite avec l’aimable
autorisation de P. Ginter/ESRF
En tant que source de troisième génération, l’ESRF produit des rayons X extrêmement intenses, appelés « rayonnement synchrotron ». Ces rayons X sont émis par des électrons de haute énergie (6 GeV) qui circulent dans un grand « anneau de stockage » long de 844 m. Les rayons X synchrotron sont très collimatés, un peu comme des rayons lasers (on dit que des rayons de lumière sont « collimatés » lorsqu’ils sont presque parallèles).
Les rayons X sont dirigés vers les lignes de lumière qui sont disposées tout autour de l’anneau de stockage, dans le hall d’expériences. Chacune des 42 lignes de lumière de l’ESRF est spécialisée dans une technique spécifique ou un type de recherche. Pour environ 10 lignes, cette spécialité est la cristallographie macromoléculaire (l’étude des cristaux de protéines). Les lignes de lumière à l’ESRF deviennent de plus en plus automatisées, ce qui les rend plus accessibles, et ceci même à grande distance. Il est maintenant possible pour les utilisateurs de mener leur expérience à bien sans quitter physiquement leur laboratoire d’origine. Les cristaux sont envoyés par courrier plutôt que transportés par les scientifiques, quand ceux-ci viennent à l’ESRF, car les contraintes de sécurité rendent difficile le transport d’échantillons biologiques sensibles.
Le rayonnement synchrotron compte pour 80% des structures de macromolécules déposées dans la Banque de données des protéinesw7 (en 1995, , seulement 17% de ces structures provenaient de données synchrotron, voir image ci-dessus). L’ESRF produit environ 20% du total des structures déposées.
References
- Ainsworth C (2009) Outmanoeuvering influenza’s tricks. Science in School 11: 25-29. www.scienceinschool.org/2009/issue11/influenza
- Blattmann B, Sticher P (2009) Growing crystals from protein. Science in School 11: 30-36. www.scienceinschool.org/2009/issue11/lysozyme
- Hughes D (2007) Taking the stress out of engineering. Science in School 5: 61-65. www.scienceinschool.org/2007/issue5/stress
- Madden D (2006) Discovering DNA. Science in School 1: 34-36. www.scienceinschool.org/2006/issue1/discoveringdna
- Schrödinger E (1944) What is Life? Cambridge, UK: Cambridge University Press
- Wilson A, Haslam S (2009) Sugary insights into worm parasite infections. Science in School 11: 20-24. www.scienceinschool.org/2009/issue11/schistosomiasis
Web References
- w1 – Pour plus d’informations sur l’ESRF, voir www.esrf.eu
- w2 – Plus d’informations sur la théorie de la cristallisation des protéines et la diffraction des rayons X peuvent être trouvées ici : www-structmed.cimr.cam.ac.uk/Course
- w3 – Pour en savoir plus sur l’antenne grenobloise de l’EMBL, voir: www.embl.fr
- w4 – Pour en savoir plus sur l’Unité de l’interaction virus / cellule-hôte, voir : www.uvhci.fr
- w5 – Le Partenariat pour la biologie structurale (PSB) est une collaboration entre l’EMBL, l’IBS, l’ESRF et l’Institut Laue-Langevin (leader mondial des sources de neutrons). Le PSB rassemble sur le même site les techniques les plus avancées en une plateforme intégrée pour la biologie structurale : clonage et expression, production de cristaux, caractérisation physique-chimique-biochimique, cristallographie par rayons X et neutrons, résonance magnétique nucléaire (RMN), microscopie électronique et tomographie, diffusion aux petits angles en rayons X et neutrons, spectroscopie de masse et microscopie optique avancée. Voir : www.psb-grenoble.eu
- w6 – Pour en savoir plus sur l’IBS, voir : www.ibs.fr
- w7 – Pour le site web de la Banque de données des protéines, voir: www.rcsb.org/pdb
Resources
- Abad-Zapatero C (2002) Crystals and Life: A Personal Journey. La Jolla, CA, USA: International University Line. ISBN: 9780972077408
- Blow D (2002) Outline of Crystallography for Biologists. Oxford, UK: Oxford University Press
- Branden C, Tooze J (1991) Introduction to Protein Structure. New York, NY, USA: Garland
- Michette A, Pfauntsch S (1996) X-rays: the first hundred years. Chichester, UK: John Wiley & Sons
- Wood EA (1972) Crystals – A Handbook for School Teachers. Chester, UK: International Union of Crystallography
- Pour un site web avec des expériences de croissance cristalline, voir: www.sciencecompany.com/sci-exper
- Pour le site web de l’Union internationale de cristallographie, voir: www.iucr.org





