Supporting materials
La bioinformática con papel y lápiz: construcción de un árbol filogenético (Word)
Download
Download this article as a PDF

Traducido por Sonia Fernández Alba. La bioinformática es generalmente llevada a cabo con un ordenador potente. Con la ayuda de Cleopatra Kozlowski, puedes investigar nuestra ascendencia primate- utilizando nada más que un papel y lápiz.
Como resultado de los recientes avances tecnológicos es relativamente rápido y fácil determinar una secuencia de ADN o de proteínas. Estas secuencias de por sí, por supuesto que nos dicen muy poco: GAATCCA, como por ejemplo. Necesitamos conocer el significado de esas secuencias. ¿Qué proteínas están codificadas por esa secuencia de ADN? ¿Codifica la secuencia de hecho una proteína? ¿Qué efecto tiene un pequeño cambio en la secuencia de ADN sobre la estructura de la proteína codificada? ¿Qué función tiene esa proteína en la célula? Y por supuesto, ¿qué puede decirnos nuestra secuencia de ADN sobre nuestra historia evolutiva?
Éstas y otras cuestiones biológicas importantes pueden ser abordadas con la bioinformática: esencialmente, mediante la comparación de ADN o secuencias de proteínas – por ejemplo, a través de la comparación de secuencias recién descubiertas con secuencias de las cuales ya tenemos mucha información (¿quizás tengan una función similar?) o comparando secuencias similares en diferentes especies.
La bioinformática se desarrolla por supuesto con la ayuda de un ordenador potente generalmente. Sin embargo, es muy fácil dejar que el ordenador haga todo el trabajo sin el conocimiento de los principios subyacentes implicados. Por esta razón, estas actividades están diseñadas para ser realizadas sobre el papel, para así conseguir que los estudiantes entiendan cómo funciona el análisis bioinformático.
Este artículo incluye un grupo de cuatro actividades. Las dos actividades preliminares (“hallazgo de genes” y “mutaciones”) y la actividad final (“ADN móvil”) pueden ser descargadas desde la página web del Laboratorio Europeo de Aprendizaje para las Ciencias de la Vida (ELLS)w1. Todas las tablas necesarias para que los estudiantes completen esta actividad, junto con el procedimiento paso a paso y las respuestas a las preguntas de comprensión, pueden ser descargadas desde la página web Science in Schoolw2.
La acumulación de mutaciones provoca que las secuencias de ADN cambien a través de generaciones. La siguiente actividad demuestra como esto puede ser utilizado para deducir las relaciones evolutivas entre organismos. Se tarda unos 90 min y no requiere más que un lápiz y las tablas, las cuales pueden ser descargadas desde la página web Science in Schoolw2.
Piensa en como clasificarías diversos animales. Tradicionalmente, las diferencias físicas entre organismos fueron utilizadas para deducir las relaciones evolutivas entre ellos, por ejemplo, si un organismo tiene una columna vertebral, o si tiene alas. Sin embargo, esto puede causar problemas. Por ejemplo, aves, murciélagos e insectos tienen todos ellos alas, pero ¿están estrechamente relacionados? ¿Cómo calculas el tiempo que ha pasado desde que los organismos divergieron de un ancestro común?
Sabemos por estudios de secuenciación de ADN que las mutaciones ocurren aleatoriamente a un ritmo muy lento y son transmitidas de padres a hijos. De este modo, si asumes que todos los organismos tienen un ancestro común, puedes utilizar las diferencias en secuencias homólogas para medir el tiempo que ha pasado desde que los organismos divergieron. En otras palabras, cuanto más tiempo haya pasado desde que dos especies divergieron de un ancestro común, más diferentes serán sus secuencias de ADN.
Las secuencias homólogas se definen como aquellas secuencias en dos organismos que tienen un origen común. En realidad no tenemos pruebas de que dos secuencias son homólogas (no estuvimos allí para observar el cambio del ADN con el tiempo), pero si son suficientemente similares, a menudo asumimos que son “homólogas”. Para conocer la similitud de dos secuencias, necesitas alinearlas correctamente (pero esto no forma parte de esta actividad).
Hay que tener en cuenta que las diferentes regiones del ADN – regiones codificantes y no codificantes – evolucionan a diferentes velocidades. En general, las regiones codificantes evolucionan más lentamente, ya que una mutación que provoca un cambio en una proteína es generalmente más costoso para el organismo – es menos probable sobrevivir y dejar descendencia. Esto es analizado en la actividad “ADN móvil”.
Para ilustrar el concepto de homología, puedes utilizar el ejemplo de filología – el estudio de la evolución de las lenguas. De hecho, hay muchos paralelismos entre los métodos usados para estudiar la evolución de las lenguas y la de los organismos.
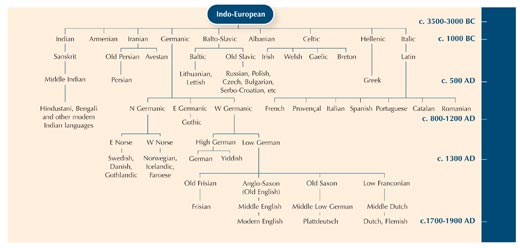
El uso de las diferencias entre fragmentos de secuencias de ADN es como comparar una palabra que significa lo mismo en diferentes idiomas, para ver lo estrechamente que están emparentadas.
| Armenio | gatz |
| Vasco | katu |
| Holandés | kat |
| Inglés | cat |
| Estonio | kass |
| Finés | kissa |
| Islándico | kottur |
| Italiano | gatto |
| Noruego | katt |
| Polaco | kot |
| Portugués | gato |
| Ruso | kot |
| Español | gato |
| Sueco | katt |
Puedes ver que las palabras “gato” en italiano, español y portugués son casi iguales: gatto, gato y gato. Tanto en sueco como en noruego, la palabra es “katt”, pero puedes ver que en finés es diferente: “kissa”. Aunque Finlandia es un país nórdico como Suecia y Noruega, la palabra finés “cat” es más parecida a la palabra en estonio “kass”. De hecho, las dos lenguas están estrechamente relacionadas. Así que puedes aprender un poco sobre las relaciones lingüísticas mediante el estudio de cómo las palabras han cambiado con el paso del tiempo.

En esta actividad, construiremos un árbol filogenético utilizando cinco secuencias homólogas de ADN de primates. Debido a que las secuencias han sido inventadas, no podemos deducir ninguna real estimación de la distancia genética; para crear un significativo árbol filogenético a partir de datos reales se requeriría secuencias mucho más largas. No obstante, las secuencias ficticias (en Tabla 2) han sido elegidas para dar una imagen bastante aproximada de las relaciones en los primates.
Nota: todas las tablas necesarias para que los estudiantes completen esta actividad pueden ser descargadas desde la página web Science in Schoolw2.
| Primate | Secuencia |
|---|---|
| Neandertal (n) | TGGTCCTGCAGTCCTCTCCTGGCGCCCCGGGCGCGAGCGGTTGTCC |
| Humano (h) | TGGTCCTGCTGTCCTCTCCTGGCGCCCTGGGCGCGAGCGGATGTCC |
| Chimpancé (c) | TGATCCTGCAGTCCTCTTCTGGCGCCCTGGGCGCGTGCGGTTGTCC |
| Gorila (g) | TGGACCTGCAGTCATCTTCTGCCCGCCCGAGCGCTTGCCGATGTCC |
| Orangután (o) | ACAACCTGCACTCCTATTCTGCCGAGCCGGGCGCGTGGCAAAGTCC |
| Neandertal | TGGTCCTGCAGTCCTCTCCTGGCGCCCCGGGCGCGAGCGGTTGTCC |
|---|---|
| Humano | TGGTCCTGCTGTCCTCTCCTGGCGCCCTGGGCGCGAGCGGATGTCC |
| Chimpancé | TGATCCTGCAGTCCTCTTCTGGCGCCCTGGGCGCGTGCGGTTGTCC |
|---|---|
| Gorila | TGGACCTGCAGTCATCTTCTGCCCGCCCGAGCGCTTGCCGATGTCC |
Las tablas de comparación para todos los pares de especies y la tabla completa de diferencias entre las secuencias (Tabla 4) pueden ser descargadas desde la página web Science in Schoolw2.
| Neandertal | Humano | Chimpancé | Gorila | Orangután | |
|---|---|---|---|---|---|
| Neandertal | 0 | 3 | |||
| Humano | 3 | 0 | |||
| Chimpancé | 0 | 11 | |||
| Gorila | 11 | 0 | |||
| Orangután |
El número de nucleótidos diferentes entre dos secuencias dividido por el número total de nucleótidos en cada secuencia (en este caso, 46) da la distancia proporcional entre dos secuencias.
La “secuencia promedio” de dos especies se asume que es el ancestro de ambos. En este ejercicio, no calculamos directamente la secuencia promedio, por ejemplo de neandertales y humanos, pero sí la distancia evoulitva entre el ancestro de neandertal / humano y todos los otros primates en el grupo.
| Neandertal / humano | Chimpancé | Gorila | Orangután | |
|---|---|---|---|---|
| Neandertal / humano | 0 | (4+5)/2 = 4.5 | (11+12)/2=11.5 | |
| Chimpancé | (4+5)/2 = 4.5 | 0 | ||
| Gorila | (11+12)/2=11.5 | 0 | ||
| Orangután |
Usando la Tabla 5, puedes empezar a construir el árbol evolutivo.
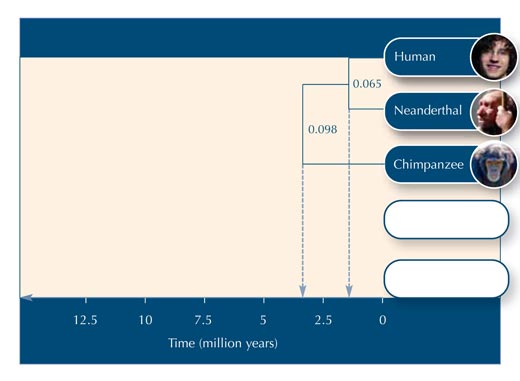
Vamos a suponer que se necesitarían 20 millones de años para que cada uno de los nucleótidos de esta particular secuencia de ADN cambien. De este modo, para que la secuencia de ADN cambie un 0.065, se necesitarían 0.065*20 millones = 1.3 millones de años. La rama debe medir por lo tanto, 1.3 millones de años en la escala de tiempo (ver Figura 2).
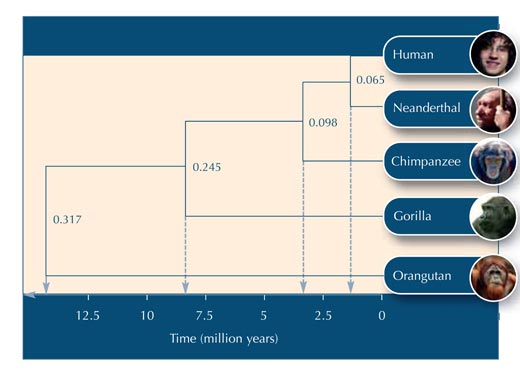
A continuación hay algunas preguntas que podrías utilizar para comprobar si tus alumnos comprendieron la actividad. Las respuestas pueden ser descargadas desde la página weg de Science in Schoolw2.
Esta actividad fue realizada en colaboración especial entre el Laboratorio Europeo de Aprendizaje para las Ciencias de la Vida (ELLS)w1 y miembros del E-STAR Laboratorio Europeo de Biología Molecular para el desarrollo de recursos didácticos para escuelas. Cleopatra Kozlowski fue respaldada mediante una beca E-STAR financiada por la Comisión Europea en el marco de su programa 6 (beca Marie Curie de acogida para formación de jóvenes investigadores), bajo el número de contrato MEST-CT-2004-504640.
Hodge R (2006) A new tree of life. Science in School 2: 17-19.
Cuando pensamos en la bioinformática, nos imaginamos probablemente enormes ordenadores y máquinas de secuenciación, pero los métodos de esta nueva ciencia pueden ser presentados por medio de actividades sencillas en el aula que son llevadas a cabo con papel y lápiz, como Cleopatra Kozlowski hace en este artículo.
El autor nos desafía con la construcción del árbol genealógico de humanos y otros primates que se basa en las diferencias genéticas entre secuencias cortas (falso) de ADN. La actividad propuesta puede ser (y de forma divertida) aprovechada en las escuelas de secundaria para abordar algunos temas complicados de biología como es el uso de los relojes moleculares en el estudio de la evolución.
El artículo está dirigido a los profesores de ciencias, quienes encontrarán ejercicios útiles de comprensión al final del texto; los estudiantes pueden también usar las preguntas para profundizar en su comprensión del tema. Las citadas webs de referencias proporcionan más información y recursos.
Giulia Realdon, Italia
La bioinformática con papel y lápiz: construcción de un árbol filogenético (Word)
Download this article as a PDF