Systembiologie im Klassenzimmer? Understand article
Übersetzt von Svantje Braun. Die Systembiologie ist eines der am schnellsten wachsenden Fachgebiete der Biowissenschaften. Aber worum handelt es sich dabei? Und gibt es dafür einen Platz im Klassenzimmer? Les Grivell von der European Molecular Biology Organization (EMBO) in Heidelberg,…
Wenn man den Begriff ‘systems biology’ (englisch für: Systembiologie) in das Suchfeld der wichtigsten biomedizinischen Literaturdatenbank PubMedw1 eingibt, und dabei die Ergebnisse auf die Zeit vor dem Jahr 2000 beschränkt, wird die Suchanfrage nur eine Handvoll Treffer liefern. Macht man dasselbe für das Jahr 2008, erhält man Links zu mehreren Hundert Veröffentlichungen, die sich mit diesem sehr schnell wachsenden Gebiet der biologischen Forschung befassen. Machen Sie die gleiche Suchanfrage bei Google, und vergleichen Sie das Ergebnis (ca. 36,9 Millionen Treffer) mit dem für Gebiete wie Hochenergiephysik (etwa 11,4 Millionen Treffer) oder Luft- und Raumfahrttechnik (um 2,5 Millionen Treffer). Angenommen, die Anzahl an Treffern spiegelt in jedem Fall die laufenden Aktivitäten in dem jeweiligen Gebiet wider, dann ist die Systembiologie wirklich ein ziemlich aktiver Neuling im Geschäft.
Aber was genau ist Systembiologie?

einem alten Elektronenröhren-Radio
Mit freundlicher Genehmigung von
Ermin Gutenberger/iStockphoto
Zyniker könnten erwidern, dass ‘Systembiologie’ nur ein Modewort ist – ein weiterer Weg für Forscher, um neue Finanzierungsquellen anzuzapfen. Physiologen könnten sagen, dass es nichts Neues ist; sondern einfach nur eine Hightech-Variante von dem, was sie schon immer getan haben oder versucht haben zu tun. Die Systembiologen selbst würden sagen, dass es eine grundlegend neue Denkweise in der Biologie ist. Anstatt die Eigenschaften isolierter Bestandteile einer Zelle oder eines Organismus zu erforschen, wie es Biochemiker und Molekularbiologen seit vielen Jahren tun, betrachten Systembiologen das Gesamtsystem.
Lassen Sie uns im Vergleich ein Radio anschauen: Ein Radio wandelt elektromagnetische Wellen in Schallwellen um, aber wir wissen nicht genau, wie. Während die Molekularbiologie das Radio bisher nur in seine Einzelteile zerlegt hat, um diese genauer zu bestimmen, versucht die Systembiologie nun zu verstehen, wie die einzelnen Teile zusammenarbeiten, um eine Funktion zu bewirken.
Betrachten wir zum Beispiel die Fähigkeit von Bakterien, eine Nährstoffquelle aufzuspüren, und in ihre Richtung zu schwimmen (Chemotaxis). Der Molekularbiologe wird versuchen, die einzelnen Komponenten der Chemotaxis-Maschinerie zu charakterisieren, und deren Funktion zu entschlüsseln – indem er die Komponenten selbst sowie die dafür codierenden Gene aufreinigt, und die Auswirkung von Mutationen auf jede Komponente untersucht. Im Gegensatz dazu möchte der Systembiologe verstehen, wie die hoch komplizierten Schwimm- und Taumel-Verhaltensmuster der Zellenw2 kontrolliert werden – indem er sich die Einflüsse und Wechselwirkungen möglichst vieler Komponenten des Systems anschaut. Systembiologen erforschen so alles vom ersten Kontakt der Nährstoffe mit dem entsprechenden Rezeptor im Bakterium, über den gesamten Signaltransduktionsweg, bis zum Mechanismus, der die Rotation des bakteriellen Antriebsmotors – des Flagellums – kontrolliert.
lange, dünne Strukturen,
sogenannte Flagellen, um sich
vorwärts zu treiben. Diese Flagellen
bilden Bündel aus, die entgegen
dem Uhrzeigersinn rotieren, und
so eine Drehkraft erzeugen, die das
Bakterium im Uhrzeigersinn rotieren
lässt
Mit freundlicher Genehmigung von Nicolle
Rager Fuller, National Science Foundation;
Bildquelle: Wikimedia Commons
Laut Leroy Hood, dem Gründer des weltweit ersten Instituts speziell für Systembiologiew3 in Seattle, USA, kann die Systembiologie anhand sechs wesentlicher Merkmale definiert werden:
- Umfassende Messungen: Wissenschaftler messen die dynamischen Veränderungen aller Gene, mRNAs und Proteine, anstatt nur einzelne Gene, mRNAs oder Proteine zu betrachten.
- Eine Integration verschiedener Datentypen: Informationen über DNA, RNA, Proteine und deren Interaktionen werden computergestützt und mathematisch zusammengefasst.
- Dynamische statt statische Messungen: über verschiedene Bereiche wie Entwicklung, Physiologie, Krankheit und Umwelt hinweg.
- Die Forschung wird getrieben von Entdeckungen und Hypothesen, nicht nur von einem von beiden.
- Die durchgeführten Messungen sind quantitativ, nicht nur qualitativ (man möchte wissen, wie viel mehr eines Proteins unter bestimmten Bedingungen produziert wird, nicht nur, dass mehr Protein vorhanden ist).
- Ein interaktiver Ablauf der Datenerzeugung: Daten → Modell → Vorhersage → Bestätigung → Modifizierung → Daten.
Auf der molekularen Ebene macht sich die Systembiologie häufig Hochdurchsatz-Technologien zunutze, wie umfangreiche DNA Sequenzierungen und RNA-, Protein-, sowie Metabolit-Analysen ganzer Zellen oder Gewebe. So werden umfassende Datensätze zusammengetragen, die das untersuchte System beschreiben. Veränderungen in der Genexpression oder in Stoffwechselprodukten, zeitlich oder als Antwort auf genetische Mutationen und/oder Reize aus der Umgebung, dienen dann der Erstellung computergestützter Modelle. Diese können Verhaltensweisen vorhersagen, und somit zu einem besseren Verständnis der zugrundeliegenden molekularen Prinzipien und Strategien beitragen.
Dieser Schwerpunkt auf dem System als Ganzes ist geradezu bezeichnend, denn er kennzeichnet die Umkehr eines stark reduktionistischen Ansatzes in der Forschung, der in den frühesten biochemischen Untersuchungen an isolierten Enzymen Ende des 19. und in der ersten Hälfte des 20. Jahrhunderts seinen Anfang fand (Cornish-Bowden & Cárdenas, 2005). Die reduktionistische Herangehensweise war zu dieser Zeit notwendig: Ohne die individuelle Aufreinigung und Untersuchung der Eigenschaften und Verhaltensweisen einzelner Zellkomponenten hätte man wenig Fortschritte machen können. Es wird jedoch immer deutlicher, dass das Verhalten einer einzelnen Zelle oder von ganzen Zellpopulationen auf eine Verstrickung unterschiedlichster Wechselwirkungen zurückzuführen ist, die sowohl höher geordneten Organisationsebenen zugrunde liegt, als auch auf einzelne Moleküle und deren Komplexe in Zellen rückkoppelt.
Sebastian Kaulitzki/iStockphoto
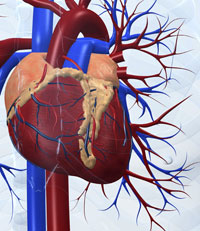
Ein gutes Beispiel für diese Art der Komplexität sind die systembiologischen Untersuchungen, die zu ersten Modellen eines menschlichen Organs führten: das virtuelle Herz (Englisch: „The virtual heart“w4 (Noble, 2007). Biophysikalische und biochemische Untersuchungen spezifischer Ionenkanäle in Herzzellen führten hierbei zunächst zu Modellen für das Verhalten einzelner Zellen, dann zu verknüpften Modellen für zwei- und dreidimensionale Gewebsausschnitte in Herzvorhof und Herzkammer. Schließlich konnten die elektrischen und mechanischen Verhaltensweisen des schlagenden Herzens als gesamtes Organ simuliert werden, in dem jede Zelle eine spezifische Rolle im Einklang mit ihren Nachbarn spielt.
Die Ergebnisse der bereits erwähnten Suchanfragen bei PubMed könnten den Eindruck erwecken, dass die Systembiologie zwischen 1999 und 2000 plötzlich auftauchte, und sich rasch entwickelte. Tatsächlich gibt es die Systembiologie in der einen oder anderen Form aber schon viel länger, unter verschiedenen Bezeichnungen. Man könnte die vorhin zitierten Physiologen – durchaus berechtigt – als die Vorreiter in diesem Bereich betrachten, denn die Physiologie ist definiert als ‚die Studie lebender Organismen und ihrer Bestandteile‘. Somit ist die Physiologie wie auch die Systembiologie selbst von Natur aus integrativ (Strange, 2005).
Und was lehrt uns die Systembiologie? Letztendlich ermöglicht diese Art der Forschung natürlich ein Verständnis für das jeweilige System, sei es ein relativ einfaches Netzwerk wechselwirkender Moleküle, eine Zelle, ein Gewebe oder ein Organ.
Auf der Ebene einer einzelnen Zelle analysieren Cheong and Levchenko (2008) die neuesten Daten, die zum NF-kappaB Signalweg gesammelt wurden. Diese molekulare Signalkaskade ist in vielen Zellen während einer entzündlichen Reaktion auf eine Infektion aktiviert. Zu den einzelnen Komponenten des Systems wurde in den vergangenen Jahrzehnten eine Fülle von Daten gesammelt. Systembiologen fassen diese Daten nun zusammen und erstellen quantitative Computermodelle des gesamten Signalweges. Anschließend wird in weiteren Experimenten überprüft, ob die Vorhersagen des Modells richtig sind. Ergebnisse dieser Analysen lieferten einen bemerkenswerten Einblick in die zugrundeliegenden, hoch komplizierten molekularen Schaltkreise, die Zellen zum Aufspüren und Bekämpfen einer Infektion verwenden. Letztendlich wird uns dieses Wissen helfen, zu verstehen, warum einzelne Personen unterschiedliche Entzündungsreaktionen aufweisen, und effektivere Behandlungsstrategien zu entwickeln.
Um Stoffwechselvorgänge einschließlich der Darmflora im Zusammenhang des gesamten menschlichen Organismus zu untersuchen, schlägt Nicholson (2006) einen systematischen Ansatz vor. Zelluläre Stoffwechselprozesse sind ein ziemliches Glücksspiel, in dem Stoffwechselprodukte oder Wirkstoffmoleküle mit Enzymen und anderen Molekülen zufällig wechselwirken. Solche Wechselwirkungen können viele Folgen haben, es könnte sogar zu Zellschädigungen kommen. Und hier betrachten wir zunächst nur eine einzige Zelle – stellen Sie sich vor, wie kompliziert es sein muss, den Stoffwechselzustand eines gesamten menschlichen Organismus vorherzusagen! Nur die Beobachtung und mathematische Modellierung des Systems als Ganzes kann uns Hoffnung machen, die Komplexität solcher Reaktionen zu verstehen, und Therapien zu entwickeln, die genau auf den Systemzustand einer bestimmten Person zugeschnitten sind.
Sollte Systembiologie in den Lehrplan von Schulen aufgenommen werden? Meiner Meinung nach – ja. Hiermit will ich nicht sagen, dass Schüler Zugang zu Einrichtungen brauchen, in denen Hochdurchsatz-Verfahren wie Microarrays oder Proteomik-Analysen durchgeführt werden. Vielmehr finde ich es wichtig, dass sie mit den Grundlagen der Systembiologie vertraut gemacht werden, und vor allem, dass sie die Einschränkungen der reduktionistischen Ansätze erkennen, die in der biologischen Forschung lange vorherrschend waren.
Angefangen mit Fragen wie „Was ist ein Gen?”, „Wieviele Gene braucht man um einen minimalen, eigenständigen Organismus zu schaffen?“ und „Wie kann ich eine biologische Uhr bauen?“, könnten Schüler an typische Konzepte von Systemen herangeführt werden. Ein Beispiel ist die Nichtlinearität biologischer Systeme, ein weitgefasster, aber wichtiger Begriff: Viele Stoffwechsel- und Signalwege sind zyklisch, nichtlinear aufgebaut. Es gibt negative und positive Rückkopplungsschleifen innerhalb einer Zelle, die sich meistens gegenseitig auf verschiedenen Ebenen beeinflussen. Außerdem ist das Verhältnis zwischen dem Input in einen biologischen Prozess und dessen Ergebnis oft nichtlinear. Es ist also ziemlich schwierig aus dem Stand heraus vorherzusagen, was passieren würde, wenn eine Komponente eines Signalweges geringfügig mehr oder weniger aktiv wäre, und wie dies alle anderen Komponenten beeinflussen würde.
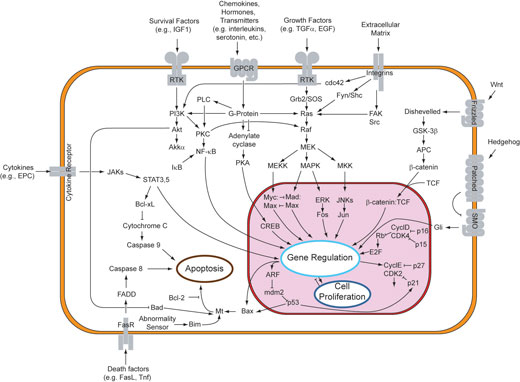
Public domain Bild; Bildquelle: Wikimedia Commons
Ein weiterer wichtiger Begriff ist Modularität: Biologische Systeme sind zwar komplex, man kann sie aber als Netzwerk kleinerer, einfacherer Einheiten (Module) betrachten, die jeweils bestimmte Funktionen ausüben. Andere zentrale Merkmale eines Systems sind Robustheit (Aufrechterhaltung der Funktion trotz genetischer oder umweltbedingter Störungen) sowie Evolvierbarkeit (die Möglichkeit zur Veränderung).
Der international Genetically Engineered Machine Wettbewerb(iGEM)w5 fordert Hochschulstudenten heraus, einige dieser Systembiologie Grundsätze in die Tat umzusetzen, indem standardisierte biologische Bauteile entworfen und angewendet werden. Die Liste der Bauteilew6 ist auf einer faszinierenden Internetseite zusammengetragen, die beweist, dass Systembiologie und synthetische Biologie Spaß machen! Neben der verblüffenden BacteriO’Clockw7 – einem einfachen Reagenzglas mit veränderten Bakterien, die ihre Farbe abhängig von der Tageszeit ändern (Team Paris) – sind dies u.a. die aktuellen Projekte der iGEM Teams: die Entwicklung einer Lactobacillus Art um Joghurt herzustellen, der die Zähne reinigtw8 (Team MIT); ein bakterieller Biosensor, der direkt in einen elektrischen Schaltkreis eingebaut werden kannw9 (Team Harvard); und eine E. coli Zelle die leuchtet, wenn sie krankheitserregende Bakterien im Trinkwasser aufspürt (Team Sheffield).
Schließlich muss folgenden Generationen bewusst gemacht werden, dass es eine enorme Vielfalt von Möglichkeiten gibt, spannende Fragestellungen aus der lebendigen Welt anzugehen. Viele davon werden von großer Bedeutung für die Gesellschaft sein. Systembiologie benötigt Systembiologen, und es bedarf der Zusammenarbeit von Wissenschaftlern aus den Disziplinen Physik, Informatik und Biologie, um dieses Forschungsgebiet so voranzutreiben, dass die Gesellschaft als Ganze davon profitieren kann.
Danksagung
Ein großer Dank gilt Dr Thomas Lemberger (EMBO), sowohl für seine Anmerkungen zu diesem Artikel, als auch für vielfältige andere Diskussionen über Systembiologie.
References
- Cheong R, Levchenko A (2008) Wires in the soup: quantitative models of cell signalling. Trends in Cell Biology 18: 112-118.
- Cornish-Bowden A, Cárdenas ML (2005) Systems biology may work when we learn to understand the parts in the terms of the whole. Biochemical Society Transactions 33: 516-519.
- Nicholson JK (2006) Global systems biology, personalized medicine and molecular epidemiology. Molecular Systems Biology 2: 52.
- Noble (2007) From the Hodgkin-Huxley axon to the virtual heart. Journal of Physiology 580(1):15-22.
- Strange K (2005) The end of “naïve reductionism”: rise of systems biology or renaissance of physiology? American Journal of Physiology 288: 968-974.
Web References
- w1 – Hier kann man auf PubMed zugreifen: www.ncbi.nlm.nih.gov/pubmed
- w2 – Hier gibt es Filme zum Bewegungsvermögen (Motilität) von Bakterien: www.microbiologybytes.com/video/motility.html
- w3 – Für weitere Informationen zum Institut für Systembiologie, siehe: www.systemsbiology.org
- w4 – Unter diesem Link gibt es ein Video, in dem Denis Noble die Grundlagen der Systembiologie anhand des virtuellen Herzens erklärt: http://videolectures.net/eccs07_noble_psb
- Finden Sie mehr zum Thema ‚virtuelles Herz‘ heraus, in: Noble D (2008) The Music of Life: Biology Beyond Genes. Oxford, Großbritannien: Oxford University Press. ISBN: 9780199228362
- Schauen Sie sich einmal die Website zum virtuellen Herz der Cornell Universität an (nicht die Version von Denis Noble, die online nicht verfügbar ist, aber trotzdem eine sehr gute): http://thevirtualheart.org
- w5 – Um mehr über den iGEM Wettbewerb zu erfahren, siehe: http://2008.igem.org/Main_Page
- w6 – Hier findet man die Liste der Bauteile (BioBricks), die im iGEM Wettbewerb eingesetzt werden; http://partsregistry.org/Main_Page
- w7 – Für eine Erläuterung und ein Video der BacteriO’Clock, siehe: http://2008.igem.org/Team:Paris
- w8 – Hier gibt es mehr Informationen zum Joghurt, der Zähne reinigt (‘biogurt’) – entwickelt vom iGEM Team MIT: http://2008.igem.org/Team:MIT
- w9 – Um mehr über das iGEM Projekt des Team Harvard – ‘bactricity’ – herauszufinden, siehe: http://2008.igem.org/Team:Harvard
Resources
- In einem aufschlussreichen historischen Überblick über die Entwicklung der Systembiologie erklären Westerhoff und Palsson (2004), wie anfängliche Konzepte der molekularen und zellulären Selbstorganisation auf die Modellierung und quantitative Analyse von Stoffwechselnetzwerken ausgeweitet wurden. Diese frühen Ansätze hatten noch einen kleinen Umfang, waren aber ein wichtiger Auftakt zur Entwicklung der heutigen Systembiologie.
- Westerhoff HV, Palsson BO (2004) The evolution of molecular biology into systems biology. Nature Biotechnology 22: 1249-1252.Schauen Sie einmal in den Blog ‚Was ist Systembiologie?’ unter http://blog-msb.embo.org/blog/2007/07/what_is_systems_biology_3.html
- Für einen Überblick über eine eher reduktionistische Methode , die dennoch auf dem neuesten Stand ist – die Proteinkristallographie – siehe:Cornuéjols D (2009) Biological crystals: at the interface between physics, chemistry and biology. Science in School 11: 70-76. www.scienceinschool.org/2009/issue11/crystallography
Review
Die Physiologie betrachtet die Funktionsweise biologischer Systeme. In diesem Artikel wird beschrieben, wie durch die Betrachtung molekularer biologischer Vorgänge ein Einblick in die Zusammenarbeit von Zellen oder Systemen – mit dem Ziel eine systemische Funktion zu erfüllen – gewonnen wird. Das Modell des virtuellen Herzens auf der angegebenen Website ist sehenswert, denn es veranschaulicht wie die Zellen als zusammenhängendes System funktionieren. Auf der Website des iGEM Wettbewerbs kann man neuartige Anwendungen der Gentechnologie und Systembiologie erkunden. So könnten interessante Diskussionen hervorgerufen werden, und einige Schüler mag es sogar dazu anregen, auf eigene Ideen zu kommen.
Anhand dieses Artikels könnten folgende Themen behandelt werden:
- Struktur und Funktion des Herzens
- Gentechnologie: soziale, ethische, und kommerzielle Anwendungen (von Produkten wie zum Beispiel Joghurt, der die Zähne reinigt!)
- Physiologie: Verflechtung mehrerer Systeme.
Mögliche Fragen zum Verständnis, die man den Schülern stellen könnte, sind folgende:
- Was ist das Ziel der Systembiologie?
- Erkläre, was mit einer molekularen Signalkaskade gemeint ist.
- Haben verschiedene Personen gleiche Entzündungsreaktionen?
- Erkläre, wie man sich die Systembiologie zunutze machen könnte, um die Reaktion einer bestimmten Person auf medikamentöse Therapien vorherzusagen.
Shelley Goodman, Großbritannien





