Supporting materials
Bioinformatik mit Stift und Papier: wie man einen phylogenetischen Stammbaum aufstellt (Word)
Download
Download this article as a PDF

Übersetzt von Hildegard Kienzle-Pfeilsticker. Bioinformatik wird üblicher weise an einem leistungsstarken Computer betrieben. Cleopatra Kozlowski jedoch verhilft uns dazu, unsere Primaten-Vorfahren zu erforschen – ausgerüstet nur mit einem Stift und Papier.
Dank neuer technologischer Fortschritte kann man eine DNA-Sequenz oder eine Proteinsequenz relativ schnell und einfach bestimmen. Natürlich sagen uns diese Sequenzen allein sehr wenig: GAATCCA, zum Beispiel. Wir müssen wissen, was diese Sequenzen bedeuten. Welche Proteine werden durch diese DNA-Sequenz codiert; codiert die Sequenz überhaupt für ein Protein? Welchen Effekt hat eine kleine Änderung in der DNA-Sequenz auf die Struktur des codierten Proteins? Welche Funktion hat das Protein in der Zelle? Und, natürlich, was sagt uns die DNA-Sequenz über die Geschichte unserer Evolution?
Diese und andere wichtige biologische Fragestellungen können mit Hilfe der Bioinformatik angegangen werden: im Wesentlichen durch den Vergleich von DNA- oder Proteinsequenzen – zum Beispiel durch den Vergleich neu entdeckter Sequenzen mit Sequenzen, zu denen uns schon viele Informationen vorliegen (haben sie vielleicht eine ähnliche Funktion?) oder indem wir ähnliche Sequenzen aus verschiedenen Spezies vergleichen.
Natürlich wird Bioinformatik üblicherweise mit Hilfe eines leistungsstarken Computers betrieben. Man macht es sich jedoch zu einfach, wenn man einen Computer die ganze Arbeit machen lässt, ohne die zu Grunde liegenden Prinzipien zu verstehen. Deshalb sind diese Aufgaben so angelegt, dass sie auf einem Stück Papier ausgeführt werden können, damit die Schüler verstehen, wie eine bioinformatische Analyse funktioniert.
Dieser Artikel enthält eine aus einer Gruppe von vier Unterrichtsaktivitäten. Die zwei einführenden („Auffindung von Genen“ und „Mutationen“) und die abschließende Unterrichtsaktivität („Mobile DNA“) können von der Webseite des „European Learning Laboratory for the Life Sciences“ (ELLS)w1 heruntergeladen werden. Alle nötigen Tabellen zur Vervollständigung dieser Aufgabe können, zusammen mit der Schritt-für-Schritt-Anleitung und den Antworten der Verständnisfragen von der Science- in-School-Webseite heruntergeladen werdenw2.
Die Akkumulierung von Mutationen ist die Ursache für DNA-Sequenzänderungen über Generationen hinweg. Die folgende Unterrichtsaktivität zeigt, wie dies für die Ableitung evolutionärer Verwandtschaften von Organismen genutzt werden kann. Es dauert etwa 90 Minuten und man braucht nur einen Stift und die Tabellen, die von der Science- in-School-Webseite heruntergeladen werden könnenw2.
Denkt darüber nach, wie ihr verschiedene Tiere einteilen würdet. Traditionell verwendete man körperliche Unterschiede zwischen Organismen, um evolutionäre Verwandtschaften abzuleiten, beispielsweise ob ein Organismus ein Rückgrat hat oder Flügel. Das könnte jedoch problematisch werden. Vögel, Fledermäuse und Insekten haben alle ein Rückgrat, aber sind sie eng miteinander verwandt? Wie kann man ermessen, wann die Organismen sich von einem gemeinsamen Vorfahr auseinanderentwickelt haben?
Aus DNA-Sequenzstudien wissen wir, dass Mutationen der DNA zufällig mit geringer Rate geschehen und dass sie von den Eltern an die Nachkommen weitergegeben werden. Angenommen, alle Organismen haben einen gemeinsamen Vorfahren, dann kann man die Unterschiede von homologen Sequenzen nutzen, um den Zeitpunkt der Divergenz der Organismen zu messen. Anders gesagt, je länger es her ist, dass zwei Spezies sich von einem gemeinsamen Vorfahr auseinanderentwickelt haben, desto unterschiedlicher werden ihre DNA-Sequenzen sein.
Homologe Sequenzen sind als die Sequenzen zweier Organismen mit gemeinsamem Ursprung definiert. Es gibt natürlich keinen Beweis für zwei homologe Sequenzen (wir waren ja nicht dabei, als sich die DNA nach und nach verändert hat), aber wenn sie sich ausreichend ähnlich sind, dann nehmen wir oft an, dass sie „homolog“ sind. Um zu erfahren, wie ähnlich sich zwei Sequenzen sind, muss man sie genau abgleichen (aber das ist nicht Teil dieser Aufgabe).
Zu beachten ist, dass verschiedene Bereiche der DNA – codierende und nicht-codierende Bereiche – sich mit unterschiedlicher Geschwindigkeit entwickeln. Allgemein entwickeln sich codierende Regionen langsamer weiter, denn eine Mutation mit einer Veränderung eines Proteins kommt den Organismus teurer zu stehen – er überlebt und pflanzt sich fort mit geringerer Wahrscheinlichkeit. Dieses Thema wird in der Unterrichtsaktivität „Mobile DNA“ diskutiert.
Um das Konzept der Homologie zu veranschaulichen, kann man die Philologie als Beispiel anführen – das Studium der Sprachenentwicklung. Es gibt wirklich viele Parallelen beim Studium der Entwicklung von Sprache und von Organismen.
Vergleicht man die Unterschiede zwischen DNA-Fragmenten, dann ist das ein bisschen wie ein Wort derselben Bedeutung in unterschiedlichen Sprachen zu vergleichen, um zu sehen wie eng sie miteinander verwandt sind.
| Armenisch | gatz |
| Baskisch | katu |
| Niederländisch | kat |
| Englisch | cat |
| Estnisch | kass |
| Finnisch | kissa |
| Isländisch | kottur |
| Italienisch | gatto |
| Norwegisch | katt |
| Polnisch | kot |
| Portugiesisch | gato |
| Russisch | kot |
| Spanisch | gato |
| Schwedisch | katt |
Man sieht, dass die Worte für “Katze” auf italienisch, spanisch und portugiesisch fast dieselben sidn: gatto, gato und gato. Sowohl auf schwedisch und norwegisch heißt das Wort „katt“, aber man sieht, dass sich finnisch unterscheidet: „kissa“. Obwohl Finnland, wie Schweden und Norwegen, ein nordisches Land ist, ähnelt das finnische Wort für „Katze“ eher dem estnischen Wort, „kass“. Die zwei Sprachen sind tatsächlich enger miteinander verwandt. Auf diese Weise kann man etwas über die Verwandtschaft von Sprachen lernen, indem man studiert, wie sich die Wörter mit der Zeit geändert haben.

Mit dieser Aufgabe erstellen wir einen phylogenetischen Stammbaum mittels fünf homologer Sequenzen von Primaten. Da die Sequenzen ausgedacht sind, können damit keine wirklichen Abschätzungen der genetischen Distanz gemacht werden; um einen sinnvollen Stammbaum aus realen Daten zu erstellen, bräuchte man viel längere Sequenzen. Trotzdem sind die Sequenzen (in Tabelle 2) so ausgewählt, dass sich ein annähernd stimmiges Bild der Primatenverwandschaft ergibt.
Beachte: Alle Tabellen, die die Schüler zur Erledigung dieser Aufgabe benötigen, können von der Science-in-School-Webseiteheruntergeladen werdenw2.
| Primat | Sequenz |
|---|---|
| Neandertaler (n) | TGGTCCTGCAGTCCTCTCCTGGCGCCCCGGGCGCGAGCGGTTGTCC |
| Mensch (h) | TGGTCCTGCTGTCCTCTCCTGGCGCCCTGGGCGCGAGCGGATGTCC |
| Schimpanse (c) | TGATCCTGCAGTCCTCTTCTGGCGCCCTGGGCGCGTGCGGTTGTCC |
| Gorilla (g) | TGGACCTGCAGTCATCTTCTGCCCGCCCGAGCGCTTGCCGATGTCC |
| Orang-Utan (o) | ACAACCTGCACTCCTATTCTGCCGAGCCGGGCGCGTGGCAAAGTCC |
| Neandertaler | TGGTCCTGCAGTCCTCTCCTGGCGCCCCGGGCGCGAGCGGTTGTCC |
|---|---|
| Mensch | TGGTCCTGCTGTCCTCTCCTGGCGCCCTGGGCGCGAGCGGATGTCC |
| Schimpanse | TGATCCTGCAGTCCTCTTCTGGCGCCCTGGGCGCGTGCGGTTGTCC |
|---|---|
| Gorilla | TGGACCTGCAGTCATCTTCTGCCCGCCCGAGCGCTTGCCGATGTCC |
Vergleichstabellen für alle Speziespaare und die vervollständigte Tabelle der Speziesunterschiede (Tabelle 4) können von der Science-in-School-Webseiteheruntergeladen werdenw2.
| Neandertaler | Mensch | Schimpanse | Gorilla | Orang-Utan | |
|---|---|---|---|---|---|
| Neandertaler | 0 | 3 | |||
| Mensch | 3 | 0 | |||
| Schimpanse | 0 | 11 | |||
| Gorilla | 11 | 0 | |||
| Orang-Utan |
Die Zahl der Nukleotiddifferenzen zwischen zwei Sequenzen dividiert durch die Gesamtzahl der Nukleotide jeder Sequenz (hier 46) ergibt den verhältnismäßigen Abstand zweier Sequenzen.
Man nimmt an, dass die “mittlere Sequenz” zweier Spezies ihr Vorfahr ist. Mit dieser Übung berechnen wir nicht direkt die mittlere Sequenz beispielsweise von Neandertalern und Menschen, sondern die evolutionäre Distanz zwischen dem gemeinsamen Vorfahr von Neandertaler und Mensch und allen anderen Primaten in der Gruppe.
| Neandertaler / Mensch | Schimpanse | Gorilla | Orang-Utan | |
|---|---|---|---|---|
| Neandertaler/ Mensch | 0 | (4+5)/2 = 4.5 | (11+12)/2=11.5 | |
| Schimpanse | (4+5)/2 = 4.5 | 0 | ||
| Gorilla | (11+12)/2=11.5 | 0 | ||
| Orang-Utan |
Mit Hilfe von Tabelle 5 kann man mit der Konstruktion des Evolutionsstammbaums beginnen.
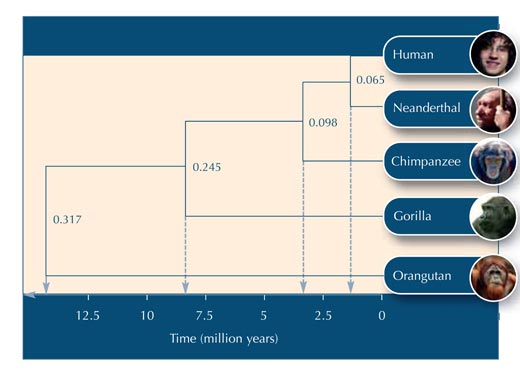
Unten sehen Sie einige Fragen, mit denen Sie das Verständnis Ihrer Schüler von dieser Aufgabe prüfen könnten. Antworten können von der Science-in-School-Webseitew2 heruntergeladen werden.
Diese Aufgaben wurden in einer speziellen Zusammenarbeit zwischen dem „European Learning Laboratory for the Life Sciences“ (ELLS)w1 und den E-STAR-Stipendiaten des European Molecular Biology Laboratory zur Entwicklung von Lehrmaterialien für Schulen erarbeitet. Cleopatra Kozlowski wurde durch ein E-STAR- Stipendium unterstützt, das durch das 6. Rahmenprogramm der Europäischen Kommission, Marie-Curie-Gaststipendium für „Early Stage Research Training” (Forschungsfrühförderung), finanziert wurde, Vertragsnummer.
Hodge R (2006) A new tree of life. Science in School 2: 17-19.
Beim Gedanken an Bioinformatik stellen wir uns wahrscheinlich große Computer und Sequenziermaschinen vor, obwohl die Methoden dieses neuen Wissenschaftszweigs mit einfachen Unterrichtsaufgaben vorgestellt werden können, ausgerüstet nur mit Papier und Stift, so wie es Cleopatra Kozlowski in diesem Artikel tut.
Die Autorin stellt uns vor die Aufgabe, einen Familienstammbaum des Menschen und anderer Primaten auf der Basis von genetischen Unterschieden zwischen kurzen (ausgedachten) DNA-Sequenzen zu erstellen. Dieser Vorschlag einer Unterichtsgestaltung kann in Schulen der Sekundarstufe gewinnbringend (und vergnüglich) ausgeschöpft werden, um einige knifflige Themen aus der Biologie, wie molekulare Uhren zum Studium der Evolution, anzugehen.
Der Beitrag richtet sich an Lehrer in den Naturwissenschaften, die am Ende des Textes nützliche Verständnisfragen finden werden; auch Schüler können die Fragen nutzen, um ihr Verständnis des Themas zu vertiefen. Die angeführten Internetreferenzen führen zu weiteren Informationen und Materialien
Giulia Realdon, Italien
Bioinformatik mit Stift und Papier: wie man einen phylogenetischen Stammbaum aufstellt (Word)
Download this article as a PDF