Expondo o nosso código genético Understand article
Traduzido por António Daniel Barbosa. Qual a função da maior parte do nosso DNA? Centenas de cientistas têm passado anos a examinar estas sequências “lixo”, que podem ser a chave para doenças graves – e muito mais.

iStockphoto
O Projeto Genoma Humano – a sequenciação de todo o genoma humano – foi o maior feito da década passada: expôs o nosso código genético, todas as três mil milhões de bases, mas a história não acaba aqui. Decifrar como esta sequência é interpretada pelas nossas células é essencial para perceber como funciona o genoma. Depois, talvez, possamos aplicar este conhecimento à investigação biomédica e cuidados de saúde.

máquina de sequenciação
automática usada pelo
Projeto Genoma Humano
para determinar a sequência
completa do DNA humano.
Cada pico mostra a presença
de uma base em particular.
O Projeto Genoma Humano
identificou as 3 mil milhões
de letras que compõem o
nosso genoma. O ENCODE
fornece agora detalhes de
como o nosso genoma
funciona. Clique na imagem
para ampliar.
Imagem cortesia de Genome
Research Limited
Uma das grandes surpresas do genoma humano foi o facto de apenas 2% do nosso genoma conter genes, as instruções para fazer proteínas. Apos contabilizar algumas partes do genoma como RNAs não codificantes, regiões envolvidas no controlo da atividade dos genes e intrões (as secções da sequência de um gene que são removidas antes da molécula de RNA ser traduzida), era uma ideia generalizada que o resto do genoma não tinha qualquer função biológica. Como tal, era frequentemente designado por DNA “lixo”.
Indo além da sequência
Uma vez sequenciado o genoma humano, foi o momento de determinar se estas sequências eram realmente lixo. Em 2003, o consórcio ENCODE foi formado para caracterizar os elementos não-codificantes mas funcionais do genoma humano. O consórcio foi financiado pelo National Human Genome Research Institute nos Estados Unidos da América e liderado pelo European Bioinformatics Institute (EBI; ver caixa) no Reino Unido. A fase piloto do ENCODE decorreu entre 2003 e 2007 e permitiu a uma rede global de investigadores testar, comparar e otimizar métodos experimentais e computacionais para a identificação de partes ativas em 1% do genoma – essencialmente filtrar algum do “lixo” genómico.
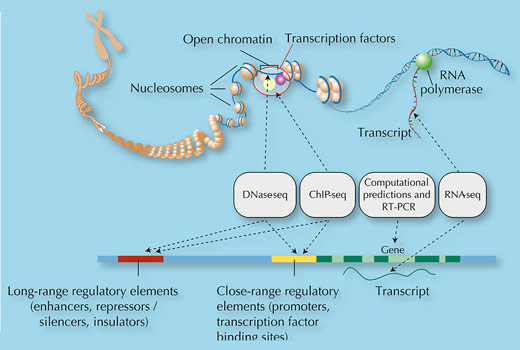
Imagem cortesia de Ian Dunham
analisou 147 tipos diferentes
de células para perceber
diferenças na regulação do
genoma em diferentes tipos
de tecidos. Este diagrama
apresenta 47 dos 147
diferentes tipos de células
incluídas neste estudo.
Diferentes tipos de células
foram usados porque, apesar
das células partilharem o
mesmo genoma, a forma
como usam esta informação
difere entre células. Clique na
imagem para ampliar.
Imagem cortesia de Darryl Leja
Os seus resultados iniciais, publicados em Junho de 2007 (O Consórcio Projeto, 2007), deram uma visão tentadora sobre o que o genoma faz. Por exemplo, dados resultantes de experiências de microarrays (ver Koutsos et al., 2009) e sequenciação mostraram que a maioria do genoma é transcrito, incluindo regiões que se pensava serem silenciosas (figura 2). Apesar do papel biológico da maioria dos transcritos ser ainda desconhecido, foi mostrado que alguns são importantes reguladores da expressão genética. No geral, esta imagem instantânea mostrou que a interação entre genes, regiões envolvidas na regulação da atividade dos genes e outro tipo de sequências de DNA é muito mais complexa do que se pensava. Os dados começavam já a indicar que o genoma continha muitas formas de elementos ativos e consequentemente menos sequências não usadas do que se pensava inicialmente.
Apos testarem com sucesso a sua abordagem, os investigadores do ENCODE começaram a examinar todo o genoma humano. Esta tarefa foi facilitada por desenvolvimentos na tecnologia de sequenciação de DNA e a disponibilidade de ensaios bioquímicos mais precisos.
A sua análise mapeou sistematicamente características do genoma, tal como um mapa descreve a paisagem física e as características geográficas como florestas, rios e montanhas. No genoma, os investigadores do ENCODE procuraram características como regiões do genoma assinaladas com sinais “shhh” (tipos específicos de grupos metil) indicando silenciamento genético, sinais de ligação de fatores de transcrição, regiões intensificadoras para aumentar a transcrição, e modificações de DNA que controlam como o DNA é empacotado (figura 3).
Inundação de informação

ENCODE fossem impressos,
o papel preencheria 12
autocarros.
Imagem cortesia de
marcus_jb1973 / Flickr
Em setembro de 2012, após 5 anos de experiências e análises de 442 investigadores oriundos de 32 institutos de investigação no Reino Unido, Estados Unidos da América, Espanha, Singapura e Japão, o projeto ENCODE anunciou os resultados da análise mais detalhada de todo o genoma até ao momento. O estudo usou cerca de 300 anos de tempo de computador para analisar 15 terabytes de dados (15 x 1012bytes), os quais estão disponíveis publicamente. Se a informação fosse impressa a uma densidade de 1000 pares de bases por cm2, a torre de papel teria 16 m de altura e mais de 30 m de comprimento: o equivalente ao volume de 12 autocarros de dois andares.
O projeto ENCODE é um exemplo do que pode ser alcançado por projetos de larga escala construídos com o contributo individual de centenas de investigadores, cada um adicionando uma peça ao puzzle para produzir uma imagem completa do genoma que não poderia ser obtida por uma única organização.
Dando vida à sequência
Um dos aspetos mais excitantes que as experiências do ENCODE mostraram foi que em vez de ser predominantemente uma sequência não-funcional, o nosso genoma está repleto de atividade: 80% do genoma é responsável por alguma função. O que faz exatamente resta ainda descobrir, mas certamente 9% dele (ou ainda mais) está envolvido na regulação da expressão genética, controlando quando e onde as proteínas são produzidas. Os 80% ativos do genoma contêm mais de 70 000 regiões promotoras – os locais de “ligação” para fatores de transcrição – e cerca de 40 000 regiões enhancer – impulsionadores que controlam a expressão de genes distantes.
Um imenso painel de controlo 3D

de um sonoplasta, a
expressão de genes é
controlada de uma forma
complexa, contendo o
genoma humano mais de 4
milhões de interruptores
genéticos.
Imagem cortesia de Stuart
Dallas Photography / Flickr
Ao todo, o ENCODE identificou mais de 4 milhões de interruptores de genes dispersos pelo genoma. O genoma poderia ser descrito como um imenso painel de controlo, tal como uma mesa de som de um sonoplasta, com imensos interruptores que ligam e desligam os genes. Esta informação aprofunda o nosso conhecimento da expressão genética e disponibiliza novas oportunidades para o tratamento de doenças. Por exemplo, uma pequena alteração num interruptor genético chamado CARD9 está associada a um aumento de 20% no risco de desenvolver a síndrome de Crohn, uma doença inflamatória do intestino. E se for possível restabelecer o interruptor genético ao seu estado normal, desligando de forma efetiva as causas da doença?
Os resultados do ENCODE também ajudaram a perceber como o genoma está organizado e as interações físicas que nele ocorrem. Os investigadores descobriram que estes interruptores genéticos estavam em contacto físico com os genes que controlam, apesar de poderem estar separados linearmente por centenas de quilobases. Nós tendemos a imaginar o genoma como uma sequência linear longa e reta mas na realidade tudo está comprimido no núcleo da célula, o que permite o contato de diferentes partes do genoma.
Compilando a informação

irão permitir um
conhecimento mais
aprofundado das bases
genéticas de doenças.
Imagem cortesia de AlexRaths
/ iStockphoto
O ENCODE fornece um mapa detalhado do genoma e abre novas áreas que a ciência pode explorar. Como Ian Dunham do EBI e autor principal do artigo ENCODE explica, “Em muitos casos é possível ter uma boa ideia dos genes envolvidos numa doença, mas pode desconhecer-se os interruptores envolvidos. Algumas vezes estes interruptores são surpreendentes – a sua localização pode ser mais logicamente associada a uma doença completamente diferente. O ENCODE dá-nos um conjunto de pistas valiosas a seguir para descobrir os mecanismos chave que desempenham um papel na saúde e doença. Estes podem ser explorados para criar novos medicamentos, ou para reformular os tratamentos existentes.”
Além de conhecer quais os genes envolvidos em doenças, os investigadores conhecem agora alguns dos interruptores que regulam a sua ativação e inativação. Esta informação será especialmente valiosa na interpretação de resultados de estudos com base em populações que identificam associações entre um gene e uma doença. Através da combinação da análise funcional do genoma do ENCODE com dados de estudos de associação em larga escala, os investigadores podem mapear as variações genéticas que têm sido associadas a doenças nas áreas de função regulatória, incluindo interruptores genéticos, identificados pelo ENCODE. As informações do ENCODE permitirão um conhecimento mais aprofundado das bases genéticas de doenças e ajudarão no trabalho dos cientistas nos anos futuros.
Mais sobre o EBI
O European Molecular Biology Laboratory (EMBL)w1 é uma das principais instituições de investigação do mundo, dedicado a investigação fundamental nas ciências da vida. O EMBL é internacional, inovador e interdisciplinar. Os seus investigadores, provenientes de 60 nações, têm formação em áreas da biologia, física, química e ciência dos computadores, e colaboram em investigação que abrange o espectro completo da biologia molecular.
O EBIw2, situado perto de Cambridge, Reino Unido, faz parte do EMBL. Fornece informações sobre experiências das ciências da vida gratuitamente para a comunidade científica global, e realiza investigação fundamental em biologia computacional. O EBI está empenhado em treinar investigadores do meio académico e indústria para aproveitar ao máximo a quantidade incrível de dados produzida diariamente nas experiências das ciências da vida.
O EMBL é um membro do EIROforumw3, o editor da Science in School.
References
- The ENCODE Project Consortium (2007) Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project. Nature 447: 799-816. doi: 10.1038/nature05874
- The ENCODE Project Consortium (2012) An integrated encyclopedia of elements in the human genome. Nature 489: 57–74. doi: 10.1038/nature11247
- Koutsos A, Manaia A, Willingale-Theune J (2009) Fishing for genes: DNA microarrays in the classroom. Science in School 12: 44-49.
Web References
- w1 – Aprenda mais sobre o EMBL.
- w2 – Aprenda mais sobre o EBI.
- w3 – EIROforum é uma colaboração entre oito das maiores organizações intergovernamentais Europeias de investigação científica, que partilham os seus recursos, instalações e conhecimentos para apoiar a ciência Europeia a atingir o seu pleno potencial. Como parte das suas atividades de educação e divulgação, o EIROforum publica a Science in School.
Resources
- Alojado no sítio da Nature, o ENCODE Explorer permite navegar pelos dados do ENCODE nos seus 13 tópicos. Pode também descarregar um poster gratuitamente(20 MB), que mostra um subgrupo dos dados ENCODE.
- Num vídeo online, a editora da Nature Magdalena Skipper e Ewan Birney do EBI discutem o projeto ENCODE.
- A Sua História: ENCODE e o Genoma Humano mostra o ENCODE no formato de cartoon.
- Ewan Birney do EBI, Tim Hubbard do Wellcome Trust Sanger Institute e Roderic Guigo do CRG apresentam ENCODE num vídeo (legendas em espanhol).
- Para introduzir a bioinformática nas suas aulas, por que não tentar uma destas atividades?
-
Kozlowski C (2010) Bioinformática com papel e lápis: construção de uma árvore filogenética. Science in School 17.
-
Communication and Public Engagement team (2010) Consegue localizar uma mutação cancerígena? Science in School 16.
-
- Para aprender mais sobre bioinformática, consultar:
-
Hayes E (2011) Um Arqueólogo do genoma: Svante Pääbo. Science in School 20.
-
Pathmanathan S, Hayes E (2007) Nicky Mulder, bioinformatician. Science in School 6: 75-77.
-
Institutions
Review
Este artigo explica um dos mais recentes avanços na genética humana: o projeto ENCODE e como a sua investigação foi realizada.
Quando os alunos são introduzidos ao código genético, são frequentemente levados a crer que apenas 2% do DNA humano codifica de facto proteínas enquanto o resto é supostamente lixo. O projeto ENCODE investigou a função de algum deste DNA não codificante, e descobriu que não é realmente lixo.
Durante a discussão do Projeto Genoma Humano com os alunos, pode também introduzir o ENCODE. Fornecer aos alunos informações sobre regulação genética, doenças genéticas e os seus tratamentos, e técnicas usadas na investigação genética pode ser útil. O artigo pode espoletar um interesse na bioinformática entre os alunos, e pode encoraja-los a pesquisar na literatura sobre o projeto ENCODE.
Namrata Garware, Índia





